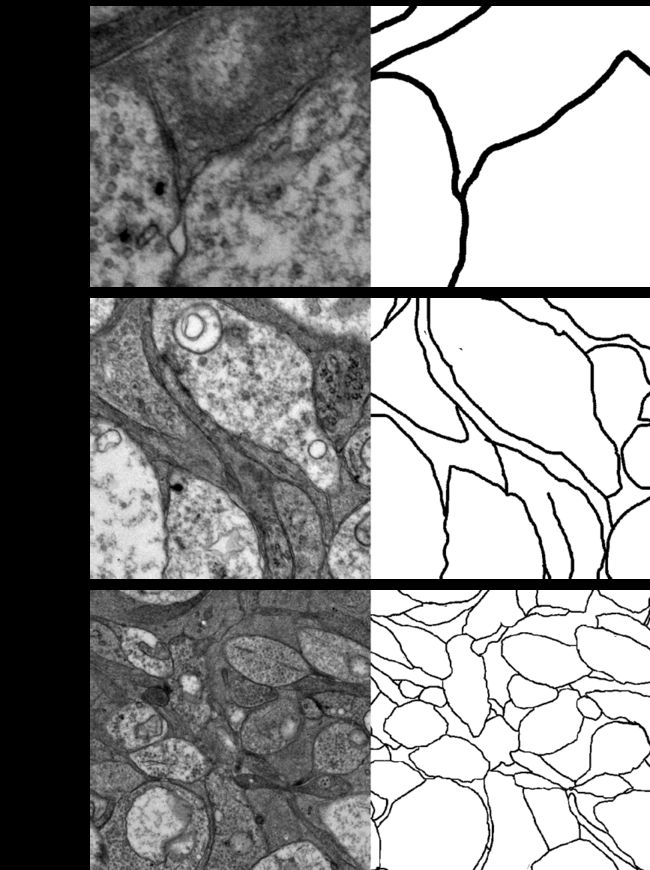
电镜图像神经元分割
适用于精细结构的语义分割网络
摘要
电子显微镜(EM)是一种广泛使用的技术,用于获取神经组织的高分辨率图像,以可视化神经元结构,如化学突触和囊泡。为了获得组织更精细的结构,超高分辨率的电镜图像已经越来越常见,但如何对这些图像进行有效的分割却是一个挑战。由于显存的限制,直接将原始图像输入网络进行训练是不允许的,而将图像裁剪成一堆小块进行训练又会损失图像的全局信息。如果直接将图像进行下采样然后再进行训练,这样又会损失图像的细节信息。如何在超高分辨图像上同时结合全局信息和细节信息是一个挑战。本文主要提出多尺度进行超高分辨率图像分割的方法,分别在三种尺度上(原始分辨率,下采样二倍和下采样四倍)独立进行网络训练,最后再进行不同分辨率下分割结果的融合。与单独的裁剪方法和下采样方法相比,我们提出的方法在保持全局信息的同时又能保持细节信息。另一方面,我们采用了两种不同模型的融合,每种模型使用不同的损失函数以达到对神经元边界分割的鲁棒性。最终实验在complex赛道上(https://www.biendata.com/competition/urisc/)获得第四名,并公布了实验代码:https://github.com/weihuang527/neuron_challenge。
关键词:神经元分割、 神经网络、 多尺度、融合
数据处理
训练数据准备
主办方提供20张大小为9958x9959带有手工标注结果(groundtruth)的训练数据集,还有10张同样大小不带有groundtruth的测试集。在实验过程中,我们在20张训练集中挑选4张作为验证集,剩余的16张作为训练集。
因为主办方提供的图像大小为9958x9959,不能被1024整除,为了后续实验的方便,我们统一将所有图像用0扩充成10240x10240的大小。
为了不同尺度网络可以同时进行训练,我们将扩充后的数据在三种尺度上各准备一份,包含原始分辨率、下采样2倍和下采样4倍。在三种分辨率上,我们分别把图像裁剪成大小为1024x1024,重叠512的图像块。
训练数据集增强
为了充分扩充训练数据集,在网络训练阶段,网络每次迭代过程中随机从裁剪块中选择一张,然后将1024x1024的图像随机裁剪成512x512的图像送进网络,这样可以充分保证训练数据的多样性。
当然本实验还包含一些常规的训练数据增强的方法,包含随机翻转(水平和竖直两个方向),随机旋转(90度、180度和270度三个方向),随机尺度变换(100像素内),随机颜色变换(对比度范围0.2,亮度范围0.2,饱和度范围0.2)。如图1所示,显示了三种分辨率下经过上述增强方法后的训练图像和对应的groundtruth。
实验方法
实验结构
P ∗ = 0.25 ∗ P f 0 + 0.1 ∗ P u 0 + 0.15 ∗ P f 2 + 0.25 ∗ P u 2 + 0.1 ∗ P f 4 + 0.15 ∗ P u 4 P^* = 0.25*P^{f0} + 0.1*P^{u0} + 0.15*P^{f2} + 0.25*P^{u2} + 0.1*P^{f4} + 0.15*P^{u4} P∗=0.25∗Pf0+0.1∗Pu0+0.15∗Pf2+0.25∗Pu2+0.1∗Pf4+0.15∗Pu4
为了得到二值化的结果,我们取阈值0.65,将小于等于0.65的像素值变为0(黑色边界),大于0.65的像素值变为1(白色背景)。
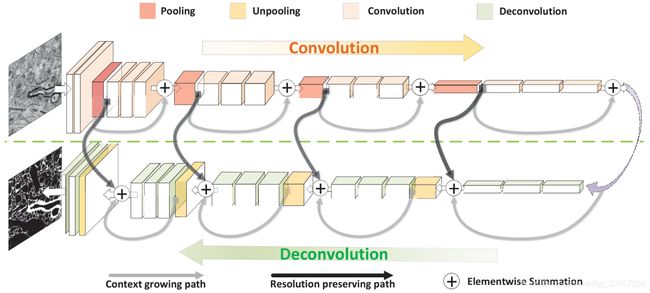
Fusionnet网络
如图3所示,Fusionnet与传统的Unet相比,它不仅包含了一个收缩路径以提取特征和一个对称的扩张路径以更好地定位,还在每层下采样和上采样之间嵌入了残差模块,同时了保留了Unet的跳层连接。从文献[1]中可以看出,Fusionnet比Unet在神经元边界提取方面,有着更强的优势。所以本文采取了文献[1]的fusionnet结构,网络第一个残差块的输出特征通道数为32,后面每下采样一次特征通道数增加2倍,共有4层下采样层。相同也有4层上采样层,每上采样一次特征通道数缩小2倍,网络的最后一层为Sigmoid函数,确保最后输出的概率图范围在0和1之间。
和文献[1]一样,我们的损失函数采用单像素的MSE函数,该损失函数能保证输出边界的宽度,不会比groundtruth更粗或者更细。
该模型在4张NVIDIA GTX XP上进行训练,在验证集上选择最优模型(原始分辨下大约需要迭代100000次,下采样2倍下与大约需要迭代40000次,下采样4倍下大约需要迭代35000次)。Batch size为16,学习率为0.0005,采用学习率逐渐衰减,衰减下限为0.00001。优化器采用Adam模型,beta1=0.9,beta2=0.999。三种不同分辨率下网络的结构以及参数保持一致没有任何修改。
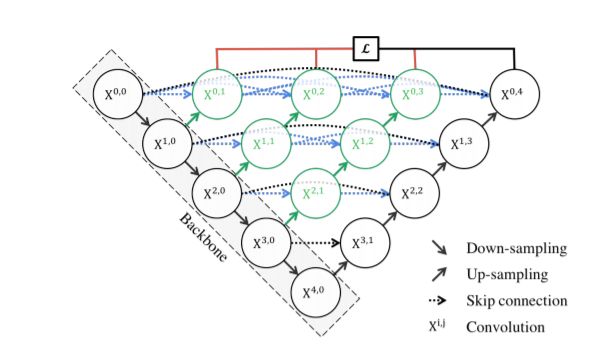
Unet++网络
如图4所示,我们选择Unet的另一个变形-Unet++[2]作为我们的分割模型。它与标准的Unet相比,它具有很多的跳层连接和不同尺度下的密集结合,极大地提升了网络提取特征的能力,并不同尺度下的训练提升了模型对不同尺度的鲁棒性,非常适用于神经元图像的分析,因为不同的神经元具有不用的大小,而多尺度训练正是克服了这一难点。同时我们将里面的BatchNorm换成了InstanceNorm,采用了Res-Net50作为backbone,因为发现Res-Net101会发生过拟合现象,Res-Net34效果不如Res-Net50。
我们采用了Cross Entropy(CE)和Dice两种函数的加权作为损失函数,其中CE的权重为0.1,Dice的权重为1。考虑到神经元边界提取中类别不平衡的问题(边界像素仅占整张图像的约5%),我们在CE中添加了类别的权重,权重值为各自的在图像中的占比。而Dice因为自带这种类别的平衡,所以我们并没有在Dice上添加权重。
同样该模型在4张NVIDIA GTX XP上进行训练,在验证集上选择最优模型(原始分辨下大约需要迭代130000次,下采样2倍下与大约需要迭代150000次,下采样4倍下大约需要迭代30000次)。Batch size为8,学习率为0.01,没有采用学习率逐渐衰减。优化器采用Adam模型,beta1=0.9,beta2=0.999。三种不同分辨率下网络的结构以及参数保持一致没有任何修改。
实验细节
本实验采用2折交叉验证,即另外从10张训练集中选择4张出来作为验证集,然后进行上述同样的训练,在测试阶段,将第1折的结果和第2折的结果直接取平均求和,然后再进行最后的加权求和。
在测试阶段,我们也采用了测试集增强方案,由于时间的限制,我们在下采样2倍和下采样4倍的模型上采用旋转90度、180度和270度,三种方式的增强,而在原始分辨率上不进行增强。
为了节省预测时间,在下采样4倍的模型下,预测时输入的图像大小为2048x2048,在下采样2倍和原始分辨率下,预测时输入的图像大小为1024x1024。
实验结果
定性分析
如图5所示,同样的模型在不同的分辨率下也有着不同的分割结果,从预测结果中,我们可以看出在低分辨率下,模型对全局信息把握得更好,细胞内的误判的边界更少或者概率更低,在全分辨率下,细节信息更准确,体现在边界粗细更准确,但缺少全局信息,导致误判的细胞内部边界更多。
定量分析
多尺度融合的有效性
| 模型 | 阈值 | 测试分数 |
|---|---|---|
| Fusionnet_x0 | 0.7 | 0.63870 |
| Fusionnet_x2 | 0.7 | 0.62883 |
| Fusionnet_x4 | 0.7 | 0.59674 |
| W_F_X0 | W_F_X2 | W_F_X4 | 阈值 | 测试分数 |
|---|---|---|---|---|
| 0.33 | 0.33 | 0.33 | 0.7 | 0.64301 |
| 0.50 | 0.30 | 0.20 | 0.7 | 0.64566 |
| 0.40 | 0.35 | 0.25 | 0.7 | 0.64490 |
从表1、2中我们可以发现,三种尺度下的融合可以极大的提升Fusionnet的分割结果。(我们所有的评测指标都在F1下进行)
多模型融合的有效性
| 模型 | Fusionnet | Unet++ | 两者融合 |
|---|---|---|---|
| 测试分数 | 0.39089 | 0.44362 | 0.45007 |
从表3中我们可以看出,两种不同的模型的融合可以显著提升分割分数。
权重以及阈值选择
| W_F_X0 | W_F_X2 | W_F_X4 | W_U_X0 | W_U_X2 | W_U_X4 | 阈值 | 分数 |
|---|---|---|---|---|---|---|---|
| 0.17 | 0.17 | 0.17 | 0.17 | 0.17 | 0.17 | 0.70 | 0.64677 |
| 0.17 | 0.17 | 0.17 | 0.17 | 0.17 | 0.17 | 0.60 | 0.64506 |
| 0.17 | 0.17 | 0.17 | 0.17 | 0.17 | 0.17 | 0.65 | 0.64955 |
| 0.25 | 0.15 | 0.10 | 0.10 | 0.15 | 0.25 | 0.65 | 0.65033 |
| 0.25 | 0.15 | 0.10 | 0.10 | 0.25 | 0.15 | 0.65 | 0.65116 |
从表4中我们可以发现,当阈值取0.65和权重分别为0.25、0.15、0.10、0.10、0.25、0.10时可以在测试集上取得相对较好的分割分数。
结论
优点:本实验考虑不同尺度和不同模型的融合,结合神经元图像的全局信息和局部细节信息,针对不同大小的神经元有着更好地鲁棒性,同时在两种不同的网络中使用不同的损失函数,可以弥补每个损失函数的缺陷,从而达到取长补短的效果。
缺点:我们发现不同的阈值对着最后的分割结果有着显著的影响,而我们对阈值的选择有一定的盲目性,只能凭借经验去选择。另一方面,六个模型求和的权重也是一个超参,目前也只能凭借进行选择。
参考文献
[1] Quan T M, Hildebrand D G C, Jeong W K. Fusionnet: A deep fully residual convolutional neural network for image segmentation in connectomics[J]. arXiv preprint arXiv:1612.05360, 2016. https://arxiv.org/abs/1612.05360
[2] Zhou Z, Siddiquee M M R, Tajbakhsh N, et al. Unet++: A nested u-net architecture for medical image segmentation[M]//Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support. Springer, Cham, 2018: 3-11.
[3] Fakhry A, Zeng T, Ji S. Residual deconvolutional networks for brain electron microscopy image segmentation[J]. IEEE transactions on medical imaging, 2016, 36(2): 447-456. https://ieeexplore.ieee.org/abstract/document/7575638