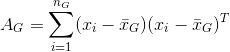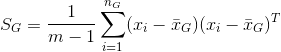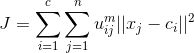【机器学习(六)】聚类算法一篇就够。无监督聚类算法:层次聚类、Kmeans聚类、模糊聚类(FCM/模糊Cmeans),从原理、案例和代码详细讲解。
目录
1.聚类概念
2.相似度或距离度量
2.1 闵可夫斯基距离(Minkowski distance)
2.2 欧式距离(Euclidean distance)
2.3 曼哈顿距离(Manhattan distance)
2.4 马哈拉诺比斯距离(Mahalanobis distance)
2.5 相关系数
2.6 夹角余弦
2.7 度量方式
3. 类或簇的基本概念
3.1 聚类分类
3.2 类或簇
3.3 类的中心
3.4 类的直径
3.5 类的散布矩阵
3.6 类的协方差
3.7 类之间最短距离或单连接
3.8 类之间最长距离或完全连接
3.9 中心距离
3.10 平均距离
4 层次聚类
4.1 层次聚合
4.2 算法流程:
4.3 案例
5. K均值聚类(Kmeans)
5.1 K均值的聚类策略
5.2 算法流程
5.3 实际案例
5.4 Kmeans的k值选择
5.5 程序
5.6 K均值聚类特性
6. 模糊聚类(FCM/模糊Cmeans)
6.1 算法流程
6.2 案例
1.聚类概念
聚类是指:把给定样本,依据它们之间的距离或相似度,划分为若干个“类”或“簇”。使得相似度较高的样本聚集在同一个类,不相似的样本分散在不同的类。
聚类属于无监督学习,方法很多,最常用的是:层次聚类、K均值聚类(Kmeans)、模糊聚类(模糊Cmeans)。在聚类中十分重要的概念就是距离或者相似度度量。
2.相似度或距离度量
聚类的输入是样本数据,每个样本又由m个属性的特征组成,所以输入可以定义为 ,其中,行数是维度的个数,列数是样本的个数。在计算距离或相似度的时候,有多种方法,一般是距离、相关系数夹角余弦。通常情况下:
,其中,行数是维度的个数,列数是样本的个数。在计算距离或相似度的时候,有多种方法,一般是距离、相关系数夹角余弦。通常情况下:
距离越小,相似度越高;距离越大,相似度越低。
相关系数越接近1,相似度越高;相关系数越接近0,相似度越低。
夹角余弦越接近1,相似度越高;夹角余弦越接近0,相似度越低。
2.1 闵可夫斯基距离(Minkowski distance)
2.2 欧式距离(Euclidean distance)
2.3 曼哈顿距离(Manhattan distance)
2.4 马哈拉诺比斯距离(Mahalanobis distance)
马哈拉诺比斯距离简称为马氏距离,考虑各个分量之间的相关性与各个分量的尺度相关性。马氏距离越大,则相似度越小;马氏距离越小,则相似度越大。
定义输入样本![]() 的协方差矩阵为
的协方差矩阵为![]() ,则样本
,则样本![]() 与
与![]() 的马氏距离为:
的马氏距离为:![]() ,其中
,其中![]() 即第i列特征值,
即第i列特征值,![]() ,即第j列特征值。
,即第j列特征值。
2.5 相关系数
样本之间的相似度除了用距离度量外,还可以使用相关系数(correlation coefficient)来表示。与距离度量不同,相关系数越接近1,则表示样本越相似;相关系数越接近于0,表示样本越不相似。
2.6 夹角余弦
样本之间的相似度还可以用夹角余弦表示,夹角余弦值越接近1,表示样本越相似;夹角预余弦值越接近0,表示样本越不相似。
2.7 度量方式
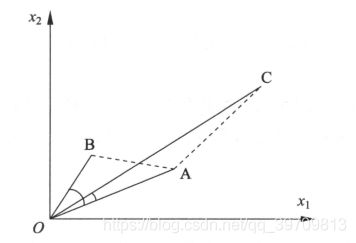
不同度量方式得到的结果不一定一致,具体采用哪种度量方式,需要结合自己实际情况出发。例如下图
从距离上看,A与B的距离更近,因此A和B比A和C更相似;从夹角余弦上看,A和C比A和B更相似。
3. 类或簇的基本概念
通过聚类,可以得到类或簇,它是输入样本的子集。了解距离和相似度量的方式后,就需要了解聚类中常用的一些概念,这样在后续的计算中,能够不使得概念混淆。
3.1 聚类分类
硬聚类:假定一个样本智能属于一个类,或类的交集为空集,这种聚类属于硬聚类。本文中的层次聚类、K均值聚类属于硬聚类。
软聚类:如果一个样本可以属于多个类,或类的交集不为空集,那么这种方法称为软聚类。
3.2 类或簇
用![]() 表示类或簇,
表示类或簇,![]() ,
,![]() 表示勒种的样本,
表示勒种的样本,![]() 表示
表示![]() 中样本的个数。
中样本的个数。![]() 表示
表示![]() ,
,![]() 的距离。
的距离。
哪怎样算是一个类或簇?
给定一个正数
,若
,则可以称
为一个类或簇
所以,当某个集合中内,任意样本之间距离小于等于给定的正数,那么就可以认为这个集合是一个类或簇。
3.3 类的中心
3.4 类的直径
类的直径是指任意两个样本之间最大的距离,定义为:![]()
3.5 类的散布矩阵
3.6 类的协方差
3.7 类之间最短距离或单连接
类![]() 与类
与类![]() 样本之间的最短距离定义为两个类之间的最短距离:
样本之间的最短距离定义为两个类之间的最短距离:![]()
3.8 类之间最长距离或完全连接
类![]() 与类
与类![]() 样本之间的最短距离定义为两个类之间的最短距离:
样本之间的最短距离定义为两个类之间的最短距离:![]()
3.9 中心距离
类![]() 与类
与类![]() 的类中心
的类中心![]() 和
和![]() 之间的距离为两个类之间的中心距离:
之间的距离为两个类之间的中心距离:![]()
3.10 平均距离
4 层次聚类
了解聚类的距离和相似度以及基础概念,就可以开始进行聚类分析。
所谓层次聚类,是指按照层次的方法找类之间的层次关系,有层次聚合(自下而上)、层次分裂(自上而下)两种方法。
如下图,层次聚合是指,初始的时候,每个样本单独属于一个类,然后按照类之间距离最小合并的原则,迭代合并类,直至合并到指定类的个数,或者只有1个类。层次分裂聚类与层次聚合相反,我们仅介绍聚合聚类。
4.1 层次聚合
输入:n个样本组成的样本集合![]()
输出:对![]() 的一个层次化聚类
的一个层次化聚类
4.2 算法流程:
- 计算n个样本两两之间的欧式距离,记做距离矩阵
;
- 构造n个类,每一个类只包含一个样本;
- 合并类间距离最小的两个类,构建一个新的类;
- 计算新的类与当前各类的距离。若类的个数为1,或者指定个数,则终止计算;否则回到3
4.3 案例
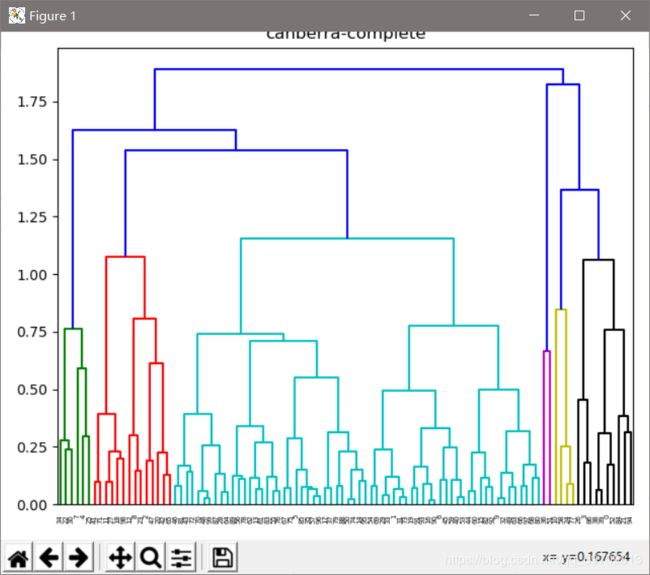
随机生成100个二维数据,按照层次聚类进行聚类。当K=4的时候,得到的聚类效果如下。
这100个样本的类标签:cluster labels:[1 0 0 1 3 0 0 3 0 0 1 0 0 0 0 0 0 0 0 0 0 1 3 0 1 3 1 0 0 0 3 0 0 0 3 0 2 0 1 1 0 0 0 0 0 0 0 0 0 0 0 2 1 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 1 0 1 0 0 0 0 0 0 0 1 0 0 0 0 0]
代码如下:
import numpy as np
import matplotlib.pyplot as plt
from scipy.spatial.distance import pdist
from scipy.cluster.hierarchy import linkage
from scipy.cluster.hierarchy import dendrogram
from sklearn.cluster import AgglomerativeClustering
colo = ['b', 'g', 'r', 'c', 'm', 'y', 'k']
# x = np.random.randint(0, 10, [100, 2])
x = 10 * np.random.rand(100, 2)
shape = x.shape
K = 4 # 簇个数
# 分析层次聚类
row_clusters = linkage(pdist(x, metric='canberra'), method='complete')
'''
pdist:计算样本距离,其中参数metric代表样本距离计算方法
(euclidean:欧式距离、minkowski:明氏距离、chebyshev:切比雪夫距离、canberra:堪培拉距离)
linkage:聚类,其中参数method代表簇间相似度计算方法
(single: MIN、ward:沃德方差最小化、average:UPGMA、complete:MAX)
'''
row_dendr = dendrogram(row_clusters)
plt.tight_layout()
plt.title('canberra-complete')
plt.show()
# 层次聚类
clf = AgglomerativeClustering(n_clusters=K, affinity='canberra', linkage='complete')
labels = clf.fit_predict(x)
print('cluster labels:%s' % labels)
for k, col in zip(range(0, K), colo):
X = labels == k
plt.plot(x[X, 0], x[X, -1], col + 'o')
plt.xlabel('x')
plt.ylabel('y')
plt.title('hierarchical_clustering')
plt.show()
5. K均值聚类(Kmeans)
K均值聚类是最常用的聚类方法,这是一种基于集合划分的聚类算法。
K均值聚类把样本集合划分为k个子集,构成k个类,将n个样本分配到k个类中,每个样本到所属类的中心距离最小。
5.1 K均值的聚类策略
k均值聚类是通过最小化损失函数选取最优的划分或函数![]() ,即采用欧式距离作为样本之间的距离度量
,即采用欧式距离作为样本之间的距离度量![]() ,然后定义样本与述所类的中心的距离总和损失函数:
,然后定义样本与述所类的中心的距离总和损失函数: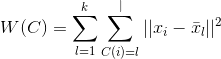 ,其中
,其中![]() 代表聚类的中心。所以K均值聚类就是最优化
代表聚类的中心。所以K均值聚类就是最优化![]() 的问题,在实现中,解决这类
的问题,在实现中,解决这类
问题的方法是通过迭代方法求解。
5.2 算法流程
输入:n个样本的集合![]()
输出:样本的聚类![]()
- 初始化,令t=0,随机选择k个样本作为初始类中心
;
- 样本聚类:计算每个样本到每个类中心的距离,将每个样本指派到与其最近的类,构成聚类结果
;
- 计算新的类中心:对聚类结果
,计算类中所有样本的均值,作为新的聚类中心;
- 如果迭代收敛,或者符合停止条件,输出
;否则,令t=t+1,返回第2步。
5.3 实际案例
5.4 Kmeans的k值选择
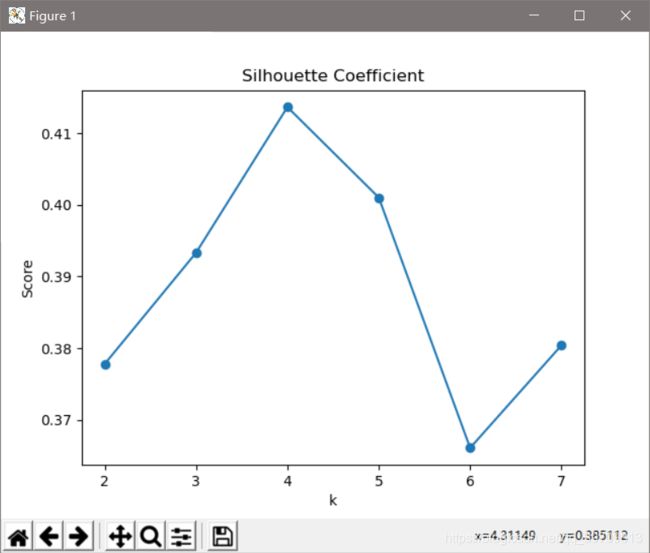
K均值的类别数,通常情况下是人为指定的。在实际应用应用中,往往不知道K取多少是最合适,因此通常是使用尝试的方法,即令k=2到x(x自己设定,少于样本个数的一半即可)。可以使用手肘法选择k值,即通过每个k值下的均方差曲线的手肘点作为最优的k值。也可以选择使用轮廓系数法作为依据,选择使轮廓最大的值对应的k值为最优k值。
5.5 程序
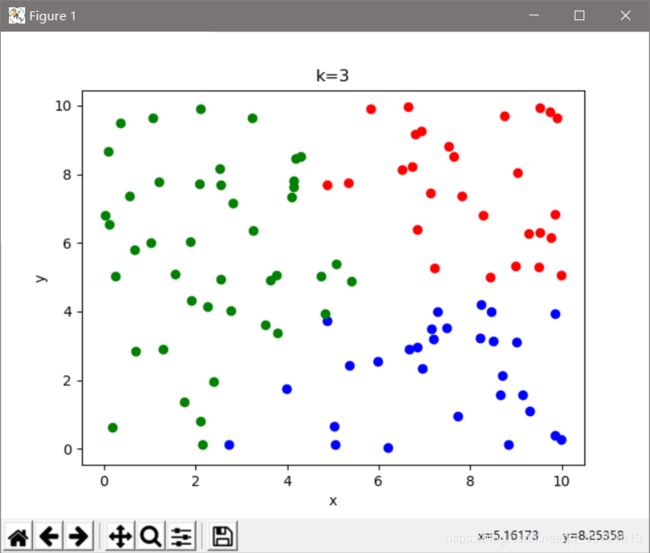
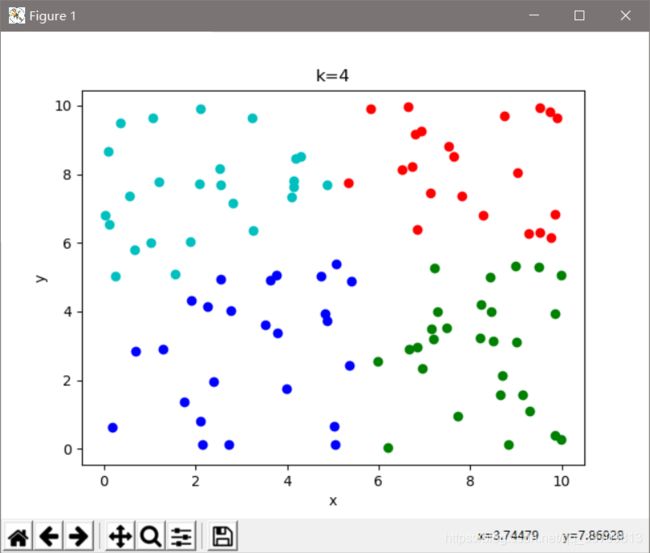
下面程序实现的是,随机选择100个二维样本,对其进行聚类,k值的从2依次递增到7。
下图是k取不同值时候的均方误差曲线,从图中可以看出,手肘点在k=4的时候。要是以此为选择依据的话,K=4时有最优的聚类结果
下图是轮廓系数法求出的值,可以看出,当K=4的时候,轮廓系数最大。若以轮廓系数为选择依据,那么K=4的时候有最优的聚类结果。
代码如下:
import numpy as np
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_score
import matplotlib.pyplot as plt
# colo = [[0, 0, 255], [0, 255, 0], [255, 0, 0], [0, 255, 255], [255, 255, 0], [255, 0, 255], [255, 255, 255]]
colo = ['b', 'g', 'r', 'c', 'm', 'y', 'k']
# x = np.random.randint(0, 10, [100, 2])
x = 10 * np.random.rand(100, 2)
shape = x.shape
sse = []
score = []
K = 8
for k in range(2, K):
clf = KMeans(n_clusters=k)
clf.fit(x)
sse.append(clf.inertia_)
lab = clf.fit_predict(x)
score.append(silhouette_score(x, clf.labels_, metric='euclidean'))
for i in range(shape[0]):
plt.xlabel('x')
plt.ylabel('y')
plt.title('k=' + str(k))
plt.plot(x[i, 0], x[i, -1], colo[lab[i]] + 'o')
plt.show()
X = range(2, K)
plt.xlabel('k')
plt.ylabel('sse')
plt.title('sum of the squared errors')
plt.plot(X, sse, 'o-', )
plt.show()
plt.xlabel('k')
plt.ylabel('Score')
plt.title('Silhouette Coefficient')
plt.plot(X, score, 'o-', )
plt.show()
5.6 K均值聚类特性
- K均值聚类往往是事先指定k值,以欧式距离作为衡量样本距离的方法。以样本到所属类中心距离总和作为目标函数。算法是使用迭代计算,往往不能得到全局最优。
- K均值聚类属于启发式方法,初始中心的选择会直接影响到聚类的结果。
- K均值聚类如何选择K,通常需要尝试不同的k值,通过手肘法或者轮廓系数法确定最优k值。
6. 模糊聚类(FCM/模糊Cmeans)
模糊聚类是指模糊C均值聚类(Fuzzy Cmeans),简写为FCM或者模糊Cmeans,同Kmeans一样,是一种被广泛应用的聚类算法。通过测试,发现,模糊Cmeans的聚类效果比Kmeans要优秀。
模糊聚类引入一个隶属度矩阵![]() ,它是衡量当前样本属于某个类的可能性大小。当前样本可能属于类1,也可能属于累2,因此这种聚类成为模糊聚类。
,它是衡量当前样本属于某个类的可能性大小。当前样本可能属于类1,也可能属于累2,因此这种聚类成为模糊聚类。
若数据集![]() 被划分成c个类,那么有c个类中心,样本
被划分成c个类,那么有c个类中心,样本![]() 与类中心
与类中心![]() 的隶属度为
的隶属度为![]() ,于是就有约束函数:
,于是就有约束函数:
接下来就是利用约束函数求解![]() 和
和![]() ,采用的拉格朗日乘数法(具体推导,点击:聚类之详解FCM算法原理及应用,这里就不做详细介绍),得到
,采用的拉格朗日乘数法(具体推导,点击:聚类之详解FCM算法原理及应用,这里就不做详细介绍),得到![]() 和
和![]() 是相互关联的。在算法开始的时候,假设其中一个值(例如
是相互关联的。在算法开始的时候,假设其中一个值(例如![]() ),通过迭代计算,
),通过迭代计算,![]() 和
和![]() 通过对方得计算自己。
通过对方得计算自己。
可以简单![]() 是样本属于摸个类的可能性大小,
是样本属于摸个类的可能性大小,![]() 是样本与贡献度乘积的结果。
是样本与贡献度乘积的结果。
6.1 算法流程
输入:样本![]() ,类的个数c,隶属度因子m(一般取2),迭代次数
,类的个数c,隶属度因子m(一般取2),迭代次数
输出:模糊Cmeans聚类结果
- 确定分类个数c,指数m的值,确定迭代次数(这是结束的条件,当然结束的条件可以有多种)。
- 初始化一个隶属度
(和为1);
- 根据
计算聚类中心
;
- 这个时候可以计算目标函数
了
- 根据
返回去计算
,回到步骤3,一直循环直到结束。
6.2 案例
随机取100个二维样本,分类样本个数为4,隶属度因子为2.
得到分类结果:cluster labels:[1 2 2 2 2 0 1 1 0 2 2 2 1 0 0 1 2 3 2 3 1 0 1 2 0 2 1 2 1 0 1 0 0 1 1 3 0 3 2 0 0 0 2 1 0 1 3 1 0 2 0 0 1 3 1 2 1 1 0 0 0 3 3 0 3 3 3 1 3 0 0 0 3 1 1 0 2 3 2 2 1 3 1 0 2 0 1 3 1 1 1 1 2 0 1 1 1 0 1 3]
代码:
import numpy as np
import matplotlib.pyplot as plt
colo = ['b', 'g', 'r', 'c', 'm', 'y', 'k']
# x = np.random.randint(0, 10, [100, 2])
x = 10 * np.random.rand(100, 2)
shape = x.shape
K = 4 # 簇个数
def FCM(X, c_clusters=3, m=2, eps=10):
membership_mat = np.random.random((len(X), c_clusters))
membership_mat = np.divide(membership_mat, np.sum(membership_mat, axis=1)[:, np.newaxis])
while True:
working_membership_mat = membership_mat ** m
Centroids = np.divide(np.dot(working_membership_mat.T, X),
np.sum(working_membership_mat.T, axis=1)[:, np.newaxis])
n_c_distance_mat = np.zeros((len(X), c_clusters))
for i, x in enumerate(X):
for j, c in enumerate(Centroids):
n_c_distance_mat[i][j] = np.linalg.norm(x - c, 2)
new_membership_mat = np.zeros((len(X), c_clusters))
for i, x in enumerate(X):
for j, c in enumerate(Centroids):
new_membership_mat[i][j] = 1. / np.sum((n_c_distance_mat[i][j] / n_c_distance_mat[i]) ** (2 / (m - 1)))
if np.sum(abs(new_membership_mat - membership_mat)) < eps:
break
membership_mat = new_membership_mat
return np.argmax(new_membership_mat, axis=1)
labels = FCM(x, c_clusters=K, m=2, eps=10)
print('cluster labels:%s' % labels)
for k, col in zip(range(0, K), colo):
X = labels == k
plt.plot(x[X, 0], x[X, -1], col + 'o')
plt.xlabel('x')
plt.ylabel('y')
plt.title('FCM')
plt.show()
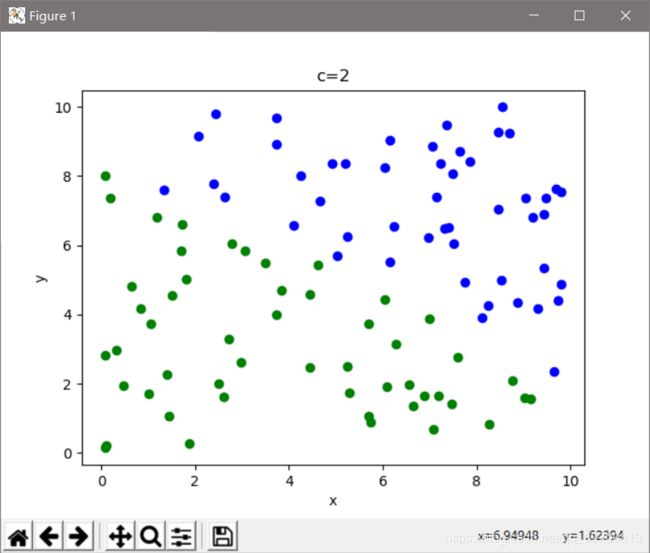
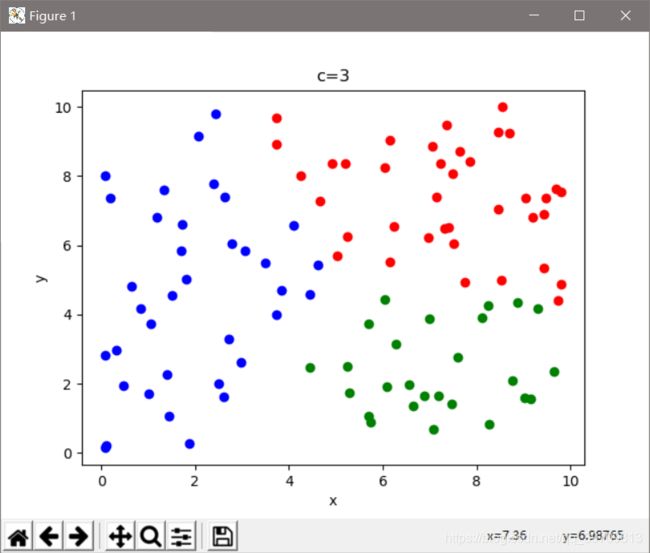
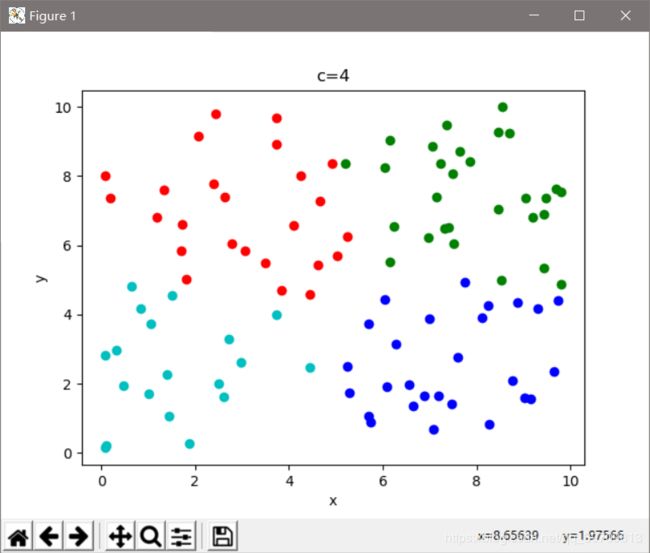
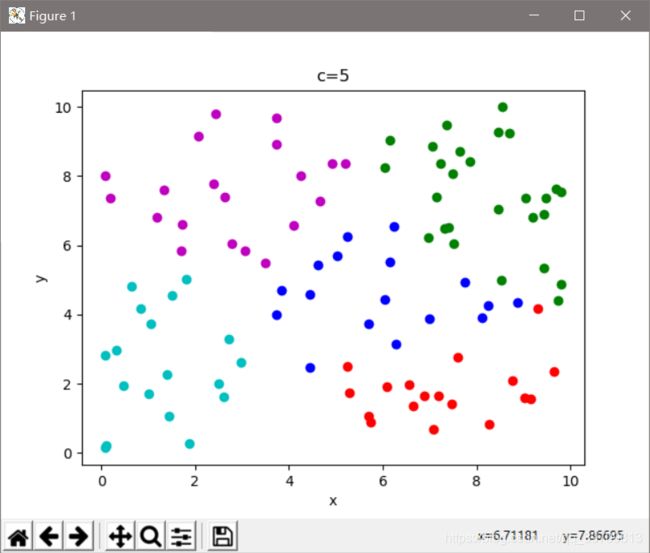
下面是使用机器学习库实现的模糊Cmeans算法,可以直观看出,在聚类效果上要比Kmeans要优秀些。
代码:
import numpy as np
from skfuzzy.cluster import cmeans
from sklearn.metrics import silhouette_score
import matplotlib.pyplot as plt
# colo = [[0, 0, 255], [0, 255, 0], [255, 0, 0], [0, 255, 255], [255, 255, 0], [255, 0, 255], [255, 255, 255]]
colo = ['b', 'g', 'r', 'c', 'm', 'y', 'k']
# x = np.random.randint(0, 10, [100, 2])
x = 10 * np.random.rand(100, 2)
shape = x.shape
FPC = []
K = 8
labels = []
for k in range(2, K):
center, u, u0, d, jm, p, fpc = cmeans(x.T, m=2, c=k, error=0.5, maxiter=10000)
'''
注意输入数据是kemans数据的转置
center是聚类中心、u是最终的隶属度矩阵、u0是初始化隶属度矩阵
d是每个数据到各个中心的欧式距离矩阵,jm是目标函数优化、p是迭代次数
fpc是评价指标,0最差,1最好
'''
label = np.argmax(u, axis=0)
for i in range(shape[0]):
plt.xlabel('x')
plt.ylabel('y')
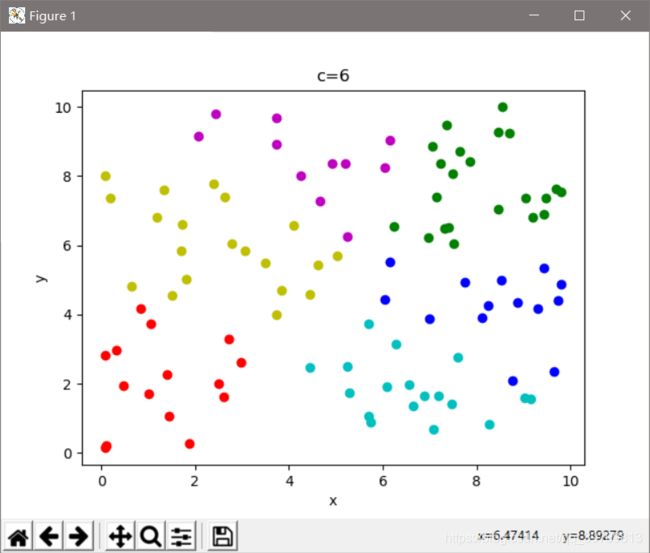
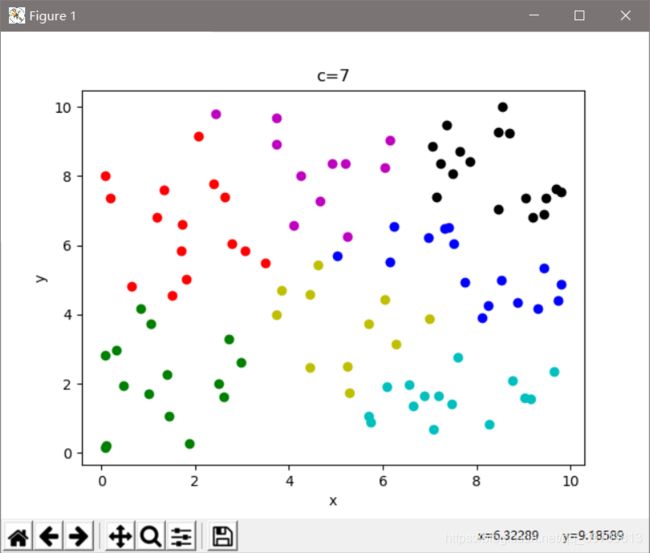
plt.title('c=' + str(k))
plt.plot(x[i, 0], x[i, -1], colo[label[i]] + 'o')
plt.show()
print('%d c\'s FPC is:%0.2f' % (k, fpc))
FPC.append(fpc)
X = range(2, K)
plt.xlabel('c')
plt.ylabel('FPC')
plt.title('FCM-cmeans')
plt.plot(X, FPC, 'o-', )
plt.show()
注意skfuzzy.cluster的安装方法pip install -U scikit-fuzzy
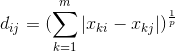
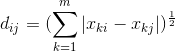

![r_i_j=\frac{\sum_{k=1}^{m}(x_k_i-\bar{x}_i)(x_k_j-\bar{x}_j)}{[\sum_{k=1}^{m}(x_k_i-\bar{x}_i)^2(x_k_j-\bar{x}_j)^2]^\frac{1}{2}}](http://img.e-com-net.com/image/info8/4924100a2201471c956890e1845fd233.gif)
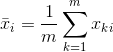
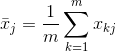
![s_i_j=\frac{\sum_{k=1}^{m}x_k_ix_k_j}{[\sum_{k=1}^{m}x_k_i^2\sum_{k=1}^{m}x_k_j^2]^\frac{1}{2}}](http://img.e-com-net.com/image/info8/c4a364e9ce0a45db93bd741e9293da6a.gif)