- c++读取文件中图像信息并用opencv展示
送分童子笑嘻嘻
#include#include#include#include#include#include#include//usingnamespacestd;usingnamespacecv;//字符串分割函数,std::vectorsplit(std::stringstr,std::stringpattern){std::string::size_typepos;std::vectorresult;s
- 数据标注管理工具:AI燃料工厂的精益引擎
花海如潮淹
人工智能经验分享笔记
标注团队的三重灾难链1.质量波动的死亡螺旋某自动驾驶公司因漏标3%的障碍物边缘,导致感知模型误判引发事故(IEEE2024案例)。质检员发现标注员A的错误率超行业标准5倍,但传统抽检仅覆盖8%数据量(ScaleAI白皮书)。2.任务调度的纳什困境某医疗影像标注项目,肝脏分割任务积压2周,而简单分类任务空闲率达37%(Labelbox调度报告)。标注员平均28%时间浪费在任务切换(Appen生产力研
- 【免费下载】 音频切割器:一键智能化裁剪音频空白
沈昂钧
音频切割器:一键智能化裁剪音频空白在音频处理的世界里,精确而高效的工具是创作的灵魂。今天要向大家隆重推荐一个名为AudioSlicer的开源宝藏应用,它通过直观的图形界面,帮助用户基于静音检测来自动分割音频文件。项目介绍AudioSlicer是一款简洁高效的小工具,专为那些希望快速无痛地将音频依据静默部分进行切割的创作者们设计。其界面友好,操作简单,即便是音频处理新手也能轻松上手。无论是播客后期制
- 深度学习在环境感知中的应用:案例与代码实现
让机器学会“看”世界:深度学习如何赋能环境感知?关键词深度学习|环境感知|计算机视觉|传感器融合|语义分割|目标检测|自动驾驶摘要环境感知是机器与外界互动的“眼睛和耳朵”——从自动驾驶汽车识别行人,到智能机器人避开障碍物,再到城市监控系统检测异常,所有智能系统都需要先“理解”环境,才能做出决策。传统环境感知方法依赖手工特征提取,难以应对复杂场景;而深度学习通过数据驱动的方式,让机器从大量数据中自动
- 深入解析Hadoop中的Region分裂与合并机制
码字的字节
hadoop布道师hadoop大数据分布式Region分裂合并
Hadoop与Region的基本概念Hadoop的分布式架构基础作为大数据处理的核心框架,Hadoop通过分布式存储和计算解决了海量数据的处理难题。其架构核心由HDFS(HadoopDistributedFileSystem)和MapReduce组成,前者负责数据的分布式存储,后者实现分布式计算。在HDFS中,数据被分割成固定大小的块(默认128MB)分散存储在集群节点上,而MapReduce则通
- 使用MMDetection中的Mask2Former和X-Decoder训练自定义数据集及结果复现
神经网络15044
算法python分类矩阵人工智能数据挖掘深度学习
使用MMDetection中的Mask2Former和X-Decoder训练自定义数据集及结果复现前些天发现了一个巨牛的人工智能学习网站,通俗易懂,风趣幽默,忍不住分享一下给大家,觉得好请收藏。点击跳转到网站。1.引言1.1研究背景实例分割是计算机视觉领域的重要任务,它要求模型不仅要检测图像中的对象,还要精确地分割出每个对象的像素级掩码。近年来,基于Transformer的模型在实例分割任务上取得
- 【Python】LEGB作用域 + re模块 + 正则表达式
文章目录一LEGB作用域二re(RegularExpression)预览1.`re.match()`——从字符串开头匹配2.`re.search()`——搜索整个字符串3.`re.findall()`——返回所有匹配的字符串列表4.`re.finditer()`——返回所有匹配的迭代器5.`re.sub()`——替换匹配的字符串6.`re.split()`——按正则表达式分割字符串7.`re.co
- 今日立冬-素食养身指南
上善若水ggg
导语又到立冬时节,古籍《月令七十二候集解》中对“冬”的解释是:“冬,终也,万物收藏也”,意思是说秋季作物全部收晒完毕,收藏入库,动物也已藏起来准备冬眠。完整地说,立冬是表示冬季开始,万物收藏,规避寒冷的意思。冬季如何进补更健康?请看看立冬素食进补指南吧!立冬宜吃哪些蔬菜?佛教浮云花纹分割线元代忽思慧所著《饮膳正要》曰:“……冬气寒,宜食黍以热性治其寒。”也就是说,少食生冷,但也不宜躁热,有的放矢地
- 《孤单是你给的苦》又名《瑕不遮玉的爱》又《我想我会一直孤单》孟思遐邵珈宸【一米文库】看全文
一米文库
《孤单是你给的苦》又名《瑕不遮玉的爱》又《我想我会一直孤单》孟思遐邵珈宸【一米文库】看全文书名:《孤单是你给的苦》《瑕不遮玉的爱》《我想我会一直孤单》主角:孟思遐,邵珈宸全文:一米文库简介:可在得知萧莞的存在后,被冷落了三年都没放弃的孟思遐,第一次感到了疲惫想要放手。她不想成为邵珈宸忘记别人的工具。也不想再等一个心里始终惦记着白月光的男人。所以,她决定瞒着他,彻底离开!------我是分割线---
- CR/DR多功能检测模体,助力提升影像诊断水平
gaoshengdainzi
CR/DR多功能检测模体
CR/DR多功能检测模体是医学影像质量控制领域不可或缺的专业工具,专为评估计算机X线摄影(ComputedRadiography,CR)和数字X线摄影(DigitalRadiography,DR)系统的成像性能而设计。在精准医疗时代,影像设备的稳定性和图像质量的可靠性直接影响临床诊断的准确性。该模体通过集成多种测试模块,可全面检测设备的空间分辨率、对比度灵敏度、均匀性、线性度等关键参数,确保成像系
- 在诛仙逍遥涧--引子
Karrybaby猪
在这科技发达的21世纪,谁也不会不相信,在这个世界的另一个地方,还有一个神秘而不可测的地方--遥池境地“仙界”。虽然这两个世界相隔甚远,但这两个世界中早已有一条无形的线将这世界中的几个人连了起来,这是命中注定的,是永远分割不开的。在那玄灵世界里,魔族刚刚被废大的青云门打败,虽已投降,但他们怎会就此罢休?魔族族长夜煞,以他的性格,怎会如此呢?但天真的青云门又怎会想到呢?他们还沉浸在胜利的喜悦当中,并
- 2022-02-28
一个人的日记0
今日跑步5公里感谢董老师分享语言伤害指数5行动指数0做一个最勤奋情绪稳定脚踏实地极致做事的人多赞美多鼓励多表扬不批评不抱怨不指责不确定不要求不对比不急躁不人生攻击日计划执行做总结25分钟分割时间今天我最伟大明天我最好看拜师全身心的信任目标不可能失败计划不要脸学会展示干就完了从善思考
- python学智能算法(二十七)|SVM-拉格朗日函数求解上
西猫雷婶
机器学习人工智能python学习笔记支持向量机python机器学习算法人工智能
【1】引言前序学习进程中,我们已经掌握了支持向量机算法中,为寻找最佳分割超平面,如何用向量表达超平面方程,如何为超平面方程建立拉格朗日函数。本篇文章的学习目标是:求解SVM拉格朗日函数。【2】求解方法【2.1】待求解函数支持量机算法的拉格朗日函数为:L(w,b,α)=12∥w∥2−∑i=1mαi[yi(w⋅xi+b−1)]L(w,b,\alpha)=\frac{1}{2}{\left\|w\rig
- 高通手机跑AI系列之——人像与背景分割
伊利丹~怒风
Qualcomm人工智能智能手机pythonarmAI编程
环境准备手机测试手机型号:RedmiK60Pro处理器:第二代骁龙8移动--8gen2运行内存:8.0GB,LPDDR5X-8400,67.0GB/s摄像头:前置16MP+后置50MP+8MP+2MPAI算力:NPU48TopsINT8&&GPU1536ALUx2x680MHz=2.089TFLOPS提示:任意手机均可以,性能越好的手机运行速度越快软件APP:AidLux2.0系统环境:Ubunt
- 2023-11-03
彭佩荣律师
调解离婚还是协议离婚?离婚案件中,有很大的一部分是双方经过协商,就离婚中所涉及的财产分割、孩子的抚养以以及经济补偿均达成一致意见,下一步就是办理离婚手续,正常的选择就是去婚姻登记机关进行登记,30天后领取《离婚证》,但是总结我办理的案件,去民政局协议离婚并不是最佳的选择。分析如下首先,民政局办理离婚,需要30天的冷静期,加长了离婚的期限,在30天内会出现很多的款知因素,也许会因为对离婚协议中内容的
- 巴里坤,我的第二故乡!
河南妞
走出校门,背起行囊赶赴边疆,来到这座边境小城巴里坤,屈指数来已经32年,在这里度过了我的青春年华,也成就了我的人生,让我成为了半个新疆人,巴里坤,成为了我的第二故乡。巴里坤小城地处东天山北麓,南靠天山,被哈巴公路分割成小城和山坡,小城北面环城路下面便是美丽的巴里坤大草原,整个小城被大草原环抱,而大草原一直向外延申至中蒙边界。巴里坤小城实景巴里坤,全名巴里坤哈萨克自治县,是全国三个哈萨克自治县之一,
- HCL 三层知识总结
HCL三层知识总结一、网络层基础1.1网络层的核心功能网络层位于数据链路层之上,主要负责跨网络的数据包转发,实现不同网段(广播域)之间的通信。其核心功能包括:寻址与路由:通过IP地址标识网络中的主机,并选择最佳路径将数据包从源端发送到目的端。分段与重组:当数据包大小超过底层链路的MTU(最大传输单元)时,将其分割为更小的片段,到达目的端后重新组合。拥塞控制:通过流量调节避免网络因过载而瘫痪(HCL
- “显著性”(Saliency)是计算机视觉中的一个重要概念,主要指的是图像或视频中最吸引人注意力的区域或对象
步步咏凉天
计算机视觉人工智能
“显著性”(Saliency)是计算机视觉中的一个重要概念,主要指的是图像或视频中最吸引人注意力的区域或对象。它模拟的是人类视觉系统对视觉场景中“显著”区域的感知能力。显著性可以用于图像理解、目标检测、图像压缩、图像分割等多个任务。下面是对显著性在计算机视觉中的几个关键方面的解释:一、显著性检测(SaliencyDetection)显著性检测的目标是预测图像中最能吸引人注意的区域,通常输出一个与输
- 【图像分割】基于模糊聚类FCM和改进的模糊聚类算法实现CT图像分割matlab代码
天天Matlab科研工作室
图像处理Matlab各类代码算法聚类matlab
1简介医学影像分割的基本目标是将图像分割成不同的解剖组织,从而可以从背景中提取出感兴趣区域。因为图像的低分辨率和弱对比度,实现医学影像分割是一件具有挑战的任务。而且,这个任务由于噪声和伪阴影变得更加困难,这些干扰项可能是因器材限制、重建算法和患者移动等原因造成的。目前还没有通用的医学图像分割算法,算法的优点和缺点经常根据所研究的问题而变化。将分割概念具体到颅内出血CT图像上,就是将颅腔中的出血病灶
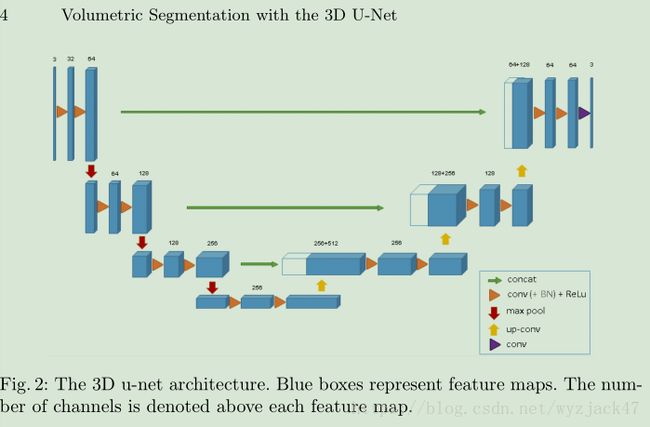
- Unet源码实现(pytorch)
wyn20001128
pytorch人工智能python
U-Net是一种用于生物医学图像分割的卷积神经网络架构。它通过引入一种新颖的网络结构和训练策略解决了传统方法在数据量不足时面临的挑战。U-Net的主要思想是利用数据增强技术来高效利用有限的标注样本,并通过独特的网络设计来提高分割精度。主要贡献U-Net的主要贡献包括:1、数据增强策略:使用随机弹性变形和其他形式的数据增强来增加训练数据的多样性,从而在有限的数据集上训练出更强大的模型。2、U形网络结
- 手持激光雷达单木分割——以河南工程学院杰出校友杨靖宇将军雕塑背后树林为例
河工点云智绘WangG
河工点云智绘教育培训
教学相长,最近带学生激光雷达实习,采集了河南工程学院校园机载、车载和手持激光雷达数据,针对手持激光雷达,也来玩玩单木分割。一、手持激光雷达单木分割概念单木分割(IndividualTreeSegmentation)是从激光雷达(LiDAR)点云数据中识别并分离出单棵树木的过程,是林业资源调查、森林碳汇估算、生物多样性研究的关键技术。二、关键技术步骤详解1.点云预处理去噪:移除飞点、鸟群等非地表物体
- 力扣题目算法分类【持续更新】
Gene_INNOCENT
比赛题解各类重要算法讲解力扣算法分类
基础算法二分704.二分查找-简单-整数二分34.在排序数组中查找元素的第一个和最后一个位置-中等69.x的平方根-简单-浮点二分287.寻找重复数-中等-二分答案410.分割数组的最大值-困难-二分答案4.寻找两个正序数组的中位数-困难
- 46. 携带研究材料(01背包二维数组) 46. 携带研究材料(01背包一维数组)LeetCode 416. 分割等和子集 Leetcode 1049. 最后一块石头的重量II
Tiny番茄
算法动态规划
46.携带研究材料(01背包二维数组)题目是给定一个物品的重量数组weight,和物品对应的价值数组value。另外给了背包需要装多少种物品,和背包的容量(即输入两个数组+背包所考虑的物品种类category和背包的容量bagweight)dp数组的定义,下标表示什么含义。dp[i][j]表示容量为j的背包从编号[0,i]之间选取物品进行存放所能达到的最大价值。其中,横轴上的坐标可以考虑为是背包的
- 力扣第70题:爬楼梯 动态规划DP入门(C++)
Daking-
leetCode耐刷王leetcode动态规划算法c++
假设你正在爬楼梯。需要n阶你才能到达楼顶。每次你可以爬1或2个台阶。你有多少种不同的方法可以爬到楼顶呢?示例1:输入:n=2输出:2解释:有两种方法可以爬到楼顶。1.1阶+1阶2.2阶示例2:输入:n=3输出:3解释:有三种方法可以爬到楼顶。1.1阶+1阶+1阶2.1阶+2阶3.2阶+1阶思路什么叫动态规划?我们分割原始问题为多个子问题,在遍历数据的过程中,如果能根据之前得到的信息动态解决当前的子
- 深入探讨DICOM医学影像中的MPPS服务及其具体实现
猿享天开
DICOM医学影像专业知识精讲DICOM医学影象PACSMPPS
深入探讨DICOM医学影像中的MPPS服务及其具体实现1.引言在医疗影像的管理和传输过程中,DICOM(数字影像和通信医学)标准发挥着至关重要的作用。除了DICOM影像的存储和传输(如影像存储SCP和影像传输SCP),DICOM还定义了其他一系列服务以支持医疗影像的完整管理。其中,**MPPS(ModalityPerformedProcedureStep)**服务是医疗影像工作流中的一个重要环节,
- 深入详解DICOM医学影像定位线相关知识:理解定位线的概念、定位线的作用以及定位线显示和计算原理
猿享天开
DICOM医学影像专业知识精讲DICOM医学影象PACS定位线
DICOM医学影像中的定位线(LocalizationLine)在医学影像学中,DICOM是用于存储和交换医学影像的标准格式。定位线(LocalizationLine)在医学影像的显示和分析中起着重要作用,它帮助医生和医学专业人员在影像中精确地标定重要的解剖结构、区域或特征,辅助进行定位、治疗计划和诊断分析。目录DICOM医学影像中的定位线(LocalizationLine)引言1.定位线的概念2
- 【TPAMI2024】计算机视觉|即插即用|FreqFusion:炸裂!告别模糊,精准分割,视觉新高度!
爆改模型
计算机视觉人工智能
论文地址:https://arxiv.org/pdf/2408.12879代码地址:https://github.com/Linwei-Chen/FreqFusion关注UPCV缝合怪,分享最计算机视觉新即插即用模块,并提供配套的论文资料与代码。https://space.bilibili.com/473764881摘要密集图像预测任务需要在高分辨率下具有强大的类别信息和精确空间边界细节的特征。为
- BEV+Transformer
Monkey PilotX
自动驾驶transformer深度学习人工智能
在自动驾驶系统中,BEV(Bird’sEyeView)+Transformer主要应用于感知与环境建图(Perception&SceneUnderstanding)环节,尤其是在多传感器融合、目标检测、语义分割、轨迹预测等任务中。在自动驾驶中的关键应用场景应用环节BEV+Transformer的作用感知(Perception)多摄像头图像融合成BEV视角,进行目标检测、语义分割预测(Predict
- 【DICOM后处理】qt+vs 实现DICOM数据四视图显示
leafpipi
实战练习qtc++算法图像处理DICOM学习
目录1、DICOM四视图2、vtkImageViewer2实现二维平面图显示3、vtkVolume实现三维体数据显示4、实现界面图1、DICOM四视图DICOM四视图通常指同时显示医学影像的四个不同平面或视角,用于全面分析三维数据(如CT、MRI等)。标准四视图布局:横截面(Axial)水平切面,从上向下观察(类似传统CT/MRI的横断面切片)。显示解剖结构的横向分布,常用于定位病变。矢状面(Sa
- 基于densenet网络创新的肺癌识别研究
深度学习乐园
深度学习人工智能densenet
获取项目源码点击文末名片摘要本项目针对肺癌CT图像识别等医学影像分析场景,基于DenseNet网络进行创新性改进,综合引入多尺度卷积、深度可分离卷积、注意力机制以及空间金字塔池化(SPP)等模块,以期提升对不同大小的肺结节及关键病理特征的识别能力。同时,通过深度可分离卷积和可选的通道剪枝等策略,将网络参数量和计算开销显著降低,为实际临床应用(如实时诊断系统)提供可行性。本项目的核心内容包括以下几个
- 怎么样才能成为专业的程序员?
cocos2d-x小菜
编程PHP
如何要想成为一名专业的程序员?仅仅会写代码是不够的。从团队合作去解决问题到版本控制,你还得具备其他关键技能的工具包。当我们询问相关的专业开发人员,那些必备的关键技能都是什么的时候,下面是我们了解到的情况。
关于如何学习代码,各种声音很多,然后很多人就被误导为成为专业开发人员懂得一门编程语言就够了?!呵呵,就像其他工作一样,光会一个技能那是远远不够的。如果你想要成为
- java web开发 高并发处理
BreakingBad
javaWeb并发开发处理高
java处理高并发高负载类网站中数据库的设计方法(java教程,java处理大量数据,java高负载数据) 一:高并发高负载类网站关注点之数据库 没错,首先是数据库,这是大多数应用所面临的首个SPOF。尤其是Web2.0的应用,数据库的响应是首先要解决的。 一般来说MySQL是最常用的,可能最初是一个mysql主机,当数据增加到100万以上,那么,MySQL的效能急剧下降。常用的优化措施是M-S(
- mysql批量更新
ekian
mysql
mysql更新优化:
一版的更新的话都是采用update set的方式,但是如果需要批量更新的话,只能for循环的执行更新。或者采用executeBatch的方式,执行更新。无论哪种方式,性能都不见得多好。
三千多条的更新,需要3分多钟。
查询了批量更新的优化,有说replace into的方式,即:
replace into tableName(id,status) values
- 微软BI(3)
18289753290
微软BI SSIS
1)
Q:该列违反了完整性约束错误;已获得 OLE DB 记录。源:“Microsoft SQL Server Native Client 11.0” Hresult: 0x80004005 说明:“不能将值 NULL 插入列 'FZCHID',表 'JRB_EnterpriseCredit.dbo.QYFZCH';列不允许有 Null 值。INSERT 失败。”。
A:一般这类问题的存在是
- Java中的List
g21121
java
List是一个有序的 collection(也称为序列)。此接口的用户可以对列表中每个元素的插入位置进行精确地控制。用户可以根据元素的整数索引(在列表中的位置)访问元素,并搜索列表中的元素。
与 set 不同,列表通常允许重复
- 读书笔记
永夜-极光
读书笔记
1. K是一家加工厂,需要采购原材料,有A,B,C,D 4家供应商,其中A给出的价格最低,性价比最高,那么假如你是这家企业的采购经理,你会如何决策?
传统决策: A:100%订单 B,C,D:0%
&nbs
- centos 安装 Codeblocks
随便小屋
codeblocks
1.安装gcc,需要c和c++两部分,默认安装下,CentOS不安装编译器的,在终端输入以下命令即可yum install gccyum install gcc-c++
2.安装gtk2-devel,因为默认已经安装了正式产品需要的支持库,但是没有安装开发所需要的文档.yum install gtk2*
3. 安装wxGTK
yum search w
- 23种设计模式的形象比喻
aijuans
设计模式
1、ABSTRACT FACTORY—追MM少不了请吃饭了,麦当劳的鸡翅和肯德基的鸡翅都是MM爱吃的东西,虽然口味有所不同,但不管你带MM去麦当劳或肯德基,只管向服务员说“来四个鸡翅”就行了。麦当劳和肯德基就是生产鸡翅的Factory 工厂模式:客户类和工厂类分开。消费者任何时候需要某种产品,只需向工厂请求即可。消费者无须修改就可以接纳新产品。缺点是当产品修改时,工厂类也要做相应的修改。如:
- 开发管理 CheckLists
aoyouzi
开发管理 CheckLists
开发管理 CheckLists(23) -使项目组度过完整的生命周期
开发管理 CheckLists(22) -组织项目资源
开发管理 CheckLists(21) -控制项目的范围开发管理 CheckLists(20) -项目利益相关者责任开发管理 CheckLists(19) -选择合适的团队成员开发管理 CheckLists(18) -敏捷开发 Scrum Master 工作开发管理 C
- js实现切换
百合不是茶
JavaScript栏目切换
js主要功能之一就是实现页面的特效,窗体的切换可以减少页面的大小,被门户网站大量应用思路:
1,先将要显示的设置为display:bisible 否则设为none
2,设置栏目的id ,js获取栏目的id,如果id为Null就设置为显示
3,判断js获取的id名字;再设置是否显示
代码实现:
html代码:
<di
- 周鸿祎在360新员工入职培训上的讲话
bijian1013
感悟项目管理人生职场
这篇文章也是最近偶尔看到的,考虑到原博客发布者可能将其删除等原因,也更方便个人查找,特将原文拷贝再发布的。“学东西是为自己的,不要整天以混的姿态来跟公司博弈,就算是混,我觉得你要是能在混的时间里,收获一些别的有利于人生发展的东西,也是不错的,看你怎么把握了”,看了之后,对这句话记忆犹新。 &
- 前端Web开发的页面效果
Bill_chen
htmlWebMicrosoft
1.IE6下png图片的透明显示:
<img src="图片地址" border="0" style="Filter.Alpha(Opacity)=数值(100),style=数值(3)"/>
或在<head></head>间加一段JS代码让透明png图片正常显示。
2.<li>标
- 【JVM五】老年代垃圾回收:并发标记清理GC(CMS GC)
bit1129
垃圾回收
CMS概述
并发标记清理垃圾回收(Concurrent Mark and Sweep GC)算法的主要目标是在GC过程中,减少暂停用户线程的次数以及在不得不暂停用户线程的请夸功能,尽可能短的暂停用户线程的时间。这对于交互式应用,比如web应用来说,是非常重要的。
CMS垃圾回收针对新生代和老年代采用不同的策略。相比同吞吐量垃圾回收,它要复杂的多。吞吐量垃圾回收在执
- Struts2技术总结
白糖_
struts2
必备jar文件
早在struts2.0.*的时候,struts2的必备jar包需要如下几个:
commons-logging-*.jar Apache旗下commons项目的log日志包
freemarker-*.jar
- Jquery easyui layout应用注意事项
bozch
jquery浏览器easyuilayout
在jquery easyui中提供了easyui-layout布局,他的布局比较局限,类似java中GUI的border布局。下面对其使用注意事项作简要介绍:
如果在现有的工程中前台界面均应用了jquery easyui,那么在布局的时候最好应用jquery eaysui的layout布局,否则在表单页面(编辑、查看、添加等等)在不同的浏览器会出
- java-拷贝特殊链表:有一个特殊的链表,其中每个节点不但有指向下一个节点的指针pNext,还有一个指向链表中任意节点的指针pRand,如何拷贝这个特殊链表?
bylijinnan
java
public class CopySpecialLinkedList {
/**
* 题目:有一个特殊的链表,其中每个节点不但有指向下一个节点的指针pNext,还有一个指向链表中任意节点的指针pRand,如何拷贝这个特殊链表?
拷贝pNext指针非常容易,所以题目的难点是如何拷贝pRand指针。
假设原来链表为A1 -> A2 ->... -> An,新拷贝
- color
Chen.H
JavaScripthtmlcss
<!DOCTYPE HTML PUBLIC "-//W3C//DTD HTML 4.01 Transitional//EN" "http://www.w3.org/TR/html4/loose.dtd"> <HTML> <HEAD>&nbs
- [信息与战争]移动通讯与网络
comsci
网络
两个坚持:手机的电池必须可以取下来
光纤不能够入户,只能够到楼宇
建议大家找这本书看看:<&
- oracle flashback query(闪回查询)
daizj
oracleflashback queryflashback table
在Oracle 10g中,Flash back家族分为以下成员:
Flashback Database
Flashback Drop
Flashback Table
Flashback Query(分Flashback Query,Flashback Version Query,Flashback Transaction Query)
下面介绍一下Flashback Drop 和Flas
- zeus持久层DAO单元测试
deng520159
单元测试
zeus代码测试正紧张进行中,但由于工作比较忙,但速度比较慢.现在已经完成读写分离单元测试了,现在把几种情况单元测试的例子发出来,希望有人能进出意见,让它走下去.
本文是zeus的dao单元测试:
1.单元测试直接上代码
package com.dengliang.zeus.webdemo.test;
import org.junit.Test;
import o
- C语言学习三printf函数和scanf函数学习
dcj3sjt126com
cprintfscanflanguage
printf函数
/*
2013年3月10日20:42:32
地点:北京潘家园
功能:
目的:
测试%x %X %#x %#X的用法
*/
# include <stdio.h>
int main(void)
{
printf("哈哈!\n"); // \n表示换行
int i = 10;
printf
- 那你为什么小时候不好好读书?
dcj3sjt126com
life
dady, 我今天捡到了十块钱, 不过我还给那个人了
good girl! 那个人有没有和你讲thank you啊
没有啦....他拉我的耳朵我才把钱还给他的, 他哪里会和我讲thank you
爸爸, 如果地上有一张5块一张10块你拿哪一张呢....
当然是拿十块的咯...
爸爸你很笨的, 你不会两张都拿
爸爸为什么上个月那个人来跟你讨钱, 你告诉他没
- iptables开放端口
Fanyucai
linuxiptables端口
1,找到配置文件
vi /etc/sysconfig/iptables
2,添加端口开放,增加一行,开放18081端口
-A INPUT -m state --state NEW -m tcp -p tcp --dport 18081 -j ACCEPT
3,保存
ESC
:wq!
4,重启服务
service iptables
- Ehcache(05)——缓存的查询
234390216
排序ehcache统计query
缓存的查询
目录
1. 使Cache可查询
1.1 基于Xml配置
1.2 基于代码的配置
2 指定可搜索的属性
2.1 可查询属性类型
2.2 &
- 通过hashset找到数组中重复的元素
jackyrong
hashset
如何在hashset中快速找到重复的元素呢?方法很多,下面是其中一个办法:
int[] array = {1,1,2,3,4,5,6,7,8,8};
Set<Integer> set = new HashSet<Integer>();
for(int i = 0
- 使用ajax和window.history.pushState无刷新改变页面内容和地址栏URL
lanrikey
history
后退时关闭当前页面
<script type="text/javascript">
jQuery(document).ready(function ($) {
if (window.history && window.history.pushState) {
- 应用程序的通信成本
netkiller.github.com
虚拟机应用服务器陈景峰netkillerneo
应用程序的通信成本
什么是通信
一个程序中两个以上功能相互传递信号或数据叫做通信。
什么是成本
这是是指时间成本与空间成本。 时间就是传递数据所花费的时间。空间是指传递过程耗费容量大小。
都有哪些通信方式
全局变量
线程间通信
共享内存
共享文件
管道
Socket
硬件(串口,USB) 等等
全局变量
全局变量是成本最低通信方法,通过设置
- 一维数组与二维数组的声明与定义
恋洁e生
二维数组一维数组定义声明初始化
/** * */ package test20111005; /** * @author FlyingFire * @date:2011-11-18 上午04:33:36 * @author :代码整理 * @introduce :一维数组与二维数组的初始化 *summary: */ public c
- Spring Mybatis独立事务配置
toknowme
mybatis
在项目中有很多地方会使用到独立事务,下面以获取主键为例
(1)修改配置文件spring-mybatis.xml <!-- 开启事务支持 --> <tx:annotation-driven transaction-manager="transactionManager" /> &n
- 更新Anadroid SDK Tooks之后,Eclipse提示No update were found
xp9802
eclipse
使用Android SDK Manager 更新了Anadroid SDK Tooks 之后,
打开eclipse提示 This Android SDK requires Android Developer Toolkit version 23.0.0 or above, 点击Check for Updates
检测一会后提示 No update were found
![]() ,其中两个超参是学习出来的。
,其中两个超参是学习出来的。![]()