随机抽样一致算法(Random sample consensus,RANSAC 简单版)PYTHON实现
一、RANSAC理论介绍
普通最小二乘是保守派:在现有数据下,如何实现最优。是从一个整体误差最小的角度去考虑,尽量谁也不得罪。
RANSAC是改革派:首先假设数据具有某种特性(目的),为了达到目的,适当割舍一些现有的数据。
给出最小二乘拟合(红线)、RANSAC(绿线)对于一阶直线、二阶曲线的拟合对比:
![]()
可以看到RANSAC可以很好的拟合。RANSAC可以理解为一种采样的方式,所以对于多项式拟合、混合高斯模型(GMM)等理论上都是适用的。
RANSAC简化版的思路就是:
第一步:假定模型(如直线方程),并随机抽取Nums个(以2个为例)样本点,对模型进行拟合:
![]()
第二步:由于不是严格线性,数据点都有一定波动,假设容差范围为:sigma,找出距离拟合曲线容差范围内的点,并统计点的个数:
![]()
第三步:重新随机选取Nums个点,重复第一步~第二步的操作,直到结束迭代:
![]()
第四步:每一次拟合后,容差范围内都有对应的数据点数,找出数据点个数最多的情况,就是最终的拟合结果:
![]()
至此:完成了RANSAC的简化版求解。
这个RANSAC的简化版,只是给定迭代次数,迭代结束找出最优。如果样本个数非常多的情况下,难不成一直迭代下去?其实RANSAC忽略了几个问题:
- 每一次随机样本数Nums的选取:如二次曲线最少需要3个点确定,一般来说,Nums少一些易得出较优结果;
- 抽样迭代次数Iter的选取:即重复多少次抽取,就认为是符合要求从而停止运算?太多计算量大,太少性能可能不够理想;
- 容差Sigma的选取:sigma取大取小,对最终结果影响较大;
代码:
# _*_ coding:utf-8 _*_
import numpy as np
import scipy as sp
import scipy.linalg as sl
# ransac_fit, ransac_data = ransac(all_data, model, 50, 1000, 7e3, 300, debug=debug, return_all=True)
def ransac(data, model, n, k, t, d, debug=False, return_all=False):
'''
参考:http://scipy.github.io/old-wiki/pages/Cookbook/RANSAC
伪代码:http://en.wikipedia.org/w/index.php?title=RANSAC&oldid=116358182
输入:
data - 样本点
model - 假设模型:事先自己确定
n - 生成模型所需的最少样本点
k - 最大迭代次数
t - 阈值:作为判断点满足模型的条件
d - 拟合较好时,需要的样本点最少的个数,当做阈值看待
输出:
bestfit - 最优拟合解(返回nil,如果未找到)
iterations = 0
bestfit = nil #后面更新
besterr = something really large #后期更新besterr = thiserr
while iterations < k
{
maybeinliers = 从样本中随机选取n个,不一定全是局内点,甚至全部为局外点
maybemodel = n个maybeinliers 拟合出来的可能符合要求的模型
alsoinliers = emptyset #满足误差要求的样本点,开始置空
for (每一个不是maybeinliers的样本点)
{
if 满足maybemodel即error < t
将点加入alsoinliers
}
if (alsoinliers样本点数目 > d)
{
%有了较好的模型,测试模型符合度
bettermodel = 利用所有的maybeinliers 和 alsoinliers 重新生成更好的模型
thiserr = 所有的maybeinliers 和 alsoinliers 样本点的误差度量
if thiserr < besterr
{
bestfit = bettermodel
besterr = thiserr
}
}
iterations++
}
return bestfit
'''
iterations = 0
bestfit = None
besterr = np.inf # 设置默认值
best_inlier_idxs = None
while iterations < k:
maybe_idxs, test_idxs = random_partition(n, data.shape[0])
maybe_inliers = data[maybe_idxs, :] # 获取size(maybe_idxs)行数据(Xi,Yi)
test_points = data[test_idxs] # 若干行(Xi,Yi)数据点
maybemodel = model.fit(maybe_inliers) # 拟合模型
test_err = model.get_error(test_points, maybemodel) # 计算误差:平方和最小
also_idxs = test_idxs[test_err < t]
also_inliers = data[also_idxs, :]
if debug:
print ('test_err.min()', test_err.min())
print ('test_err.max()', test_err.max())
print ('numpy.mean(test_err)', numpy.mean(test_err))
print ('iteration %d:len(alsoinliers) = %d' % (iterations, len(also_inliers)))
if len(also_inliers > d):
betterdata = np.concatenate((maybe_inliers, also_inliers)) # 样本连接
bettermodel = model.fit(betterdata)
better_errs = model.get_error(betterdata, bettermodel)
thiserr = np.mean(better_errs) # 平均误差作为新的误差
if thiserr < besterr:
bestfit = bettermodel
besterr = thiserr
best_inlier_idxs = np.concatenate((maybe_idxs, also_idxs)) # 更新局内点,将新点加入
iterations += 1
if bestfit is None:
raise ValueError("did't meet fit acceptance criteria")
if return_all:
return bestfit, {'inliers': best_inlier_idxs}
else:
return bestfit
def random_partition(n, n_data):
"""return n random rows of data and the other len(data) - n rows"""
all_idxs = np.arange(n_data) # 获取n_data下标索引
np.random.shuffle(all_idxs) # 打乱下标索引
idxs1 = all_idxs[:n]
idxs2 = all_idxs[n:]
return idxs1, idxs2
class LinearLeastSquareModel:
# 最小二乘求线性解,用于RANSAC的输入模型
def __init__(self, input_columns, output_columns, debug=False):
self.input_columns = input_columns
self.output_columns = output_columns
self.debug = debug
def fit(self, data):
A = np.vstack([data[:, i] for i in self.input_columns]).T # 第一列Xi-->行Xi
B = np.vstack([data[:, i] for i in self.output_columns]).T # 第二列Yi-->行Yi
x, resids, rank, s = sl.lstsq(A, B) # residues:残差和
return x # 返回最小平方和向量
def get_error(self, data, model):
A = np.vstack([data[:, i] for i in self.input_columns]).T # 第一列Xi-->行Xi
B = np.vstack([data[:, i] for i in self.output_columns]).T # 第二列Yi-->行Yi
B_fit = sp.dot(A, model) # 计算的y值,B_fit = model.k*A + model.b
err_per_point = np.sum((B - B_fit) ** 2, axis=1) # sum squared error per row
return err_per_point
def test():
# 生成理想数据
n_samples = 500 # 样本个数
n_inputs = 1 # 输入变量个数
n_outputs = 1 # 输出变量个数
A_exact = 20 * np.random.random((n_samples, n_inputs)) # 随机生成0-20之间的500个数据:行向量
perfect_fit = 60 * np.random.normal(size=(n_inputs, n_outputs)) # 随机线性度即随机生成一个斜率
B_exact = sp.dot(A_exact, perfect_fit) # y = x * k
# 加入高斯噪声,最小二乘能很好的处理
A_noisy = A_exact + np.random.normal(size=A_exact.shape) # 500 * 1行向量,代表Xi
B_noisy = B_exact + np.random.normal(size=B_exact.shape) # 500 * 1行向量,代表Yi
if 1:
# 添加"局外点"
n_outliers = 100
all_idxs = np.arange(A_noisy.shape[0]) # 获取索引0-499
np.random.shuffle(all_idxs) # 将all_idxs打乱
outlier_idxs = all_idxs[:n_outliers] # 100个0-500的随机局外点
A_noisy[outlier_idxs] = 20 * np.random.random((n_outliers, n_inputs)) # 加入噪声和局外点的Xi
B_noisy[outlier_idxs] = 50 * np.random.normal(size=(n_outliers, n_outputs)) # 加入噪声和局外点的Yi
# setup model
all_data = np.hstack((A_noisy, B_noisy)) # 形式([Xi,Yi]....) shape:(500,2)500行2列
input_columns = range(n_inputs) # 数组的第一列x:0
output_columns = [n_inputs + i for i in range(n_outputs)] # 数组最后一列y:1
debug = False
model = LinearLeastSquareModel(input_columns, output_columns, debug=debug) # 类的实例化:用最小二乘生成已知模型
linear_fit, resids, rank, s = sp.linalg.lstsq(all_data[:, input_columns], all_data[:, output_columns])
# run RANSAC 算法
ransac_fit, ransac_data = ransac(all_data, model, 100, 1e4, 7, 30, debug=debug, return_all=True)
# ransac_fit, ransac_data = ransac(all_data, model, 100, 1e4, 0.01, 30, debug=debug, return_all=True)
'''
输入:
data - 样本点
model - 假设模型:事先自己确定
n - 生成模型所需的最少样本点
k - 最大迭代次数
t - 阈值:作为判断点满足模型的条件
d - 拟合较好时, 需要的样本点最少的个数, 当做阈值看待
输出:
bestfit - 最优拟合解(返回nil, 如果未找到)
'''
if 1:
import pylab
sort_idxs = np.argsort(A_exact[:, 0])
A_col0_sorted = A_exact[sort_idxs] # 秩为2的数组
if 1:
pylab.plot(A_noisy[:, 0], B_noisy[:, 0], 'k.', label='data') # 散点图
pylab.plot(A_noisy[ransac_data['inliers'], 0], B_noisy[ransac_data['inliers'], 0], 'bx',
label="RANSAC data")
else:
pylab.plot(A_noisy[non_outlier_idxs, 0], B_noisy[non_outlier_idxs, 0], 'k.', label='noisy data')
pylab.plot(A_noisy[outlier_idxs, 0], B_noisy[outlier_idxs, 0], 'r.', label='outlier data')
pylab.plot(A_col0_sorted[:, 0],
np.dot(A_col0_sorted, ransac_fit)[:, 0],
label='RANSAC fit')
pylab.plot(A_col0_sorted[:, 0],
np.dot(A_col0_sorted, perfect_fit)[:, 0],
# label='exact system')
label='perfect fit')
pylab.plot(A_col0_sorted[:, 0],
np.dot(A_col0_sorted, linear_fit)[:, 0],
label='linear fit')
pylab.legend()
pylab.show()
if __name__ == "__main__":
test()
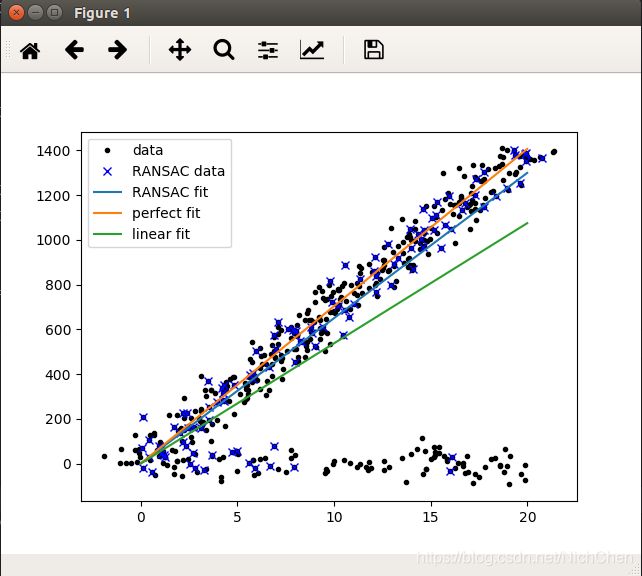
运行结果:
注:
- RANSAC的作用有点类似:将数据一切两段,一部分是自己人,一部分是敌人,自己人留下商量事,敌人赶出去。
- RANSAC开的是家庭会议,不像最小二乘总是开全体会议。
- RANSAC在每一代的循环开始时,时随机选取的一些点,做最小二乘法来确定拟合曲线的,在多次循环后保留最佳的那一次随机生成的拟合曲线。
参考资料:
http://www.cnblogs.com/xingshansi/p/6763668.html
https://blog.csdn.net/vict_wang/article/details/81027730