python手写神经网络之优化器(Optimizer)SGD、Momentum、Adagrad、RMSProp、Adam实现与对比——《深度学习入门——基于Python的理论与实现(第六章)》
vanila SGD先不写了,很简单,主要从Momentum开始。
老规矩,先手写,再对照书本:
其实这个还真难手写出一样的,尤其v的初始化,我就没想到他怎么做。
他默认了很多规则在里边,他的v没在init初始化,也不能动态,二是在第一次update时定型。
其他方面,有些地方k、v对,其实用k或者v都能达到效果,就不赘述
class Momentum():
def __init__(self,lr = 0.01,momentum = 0.9):
self.lr = lr
self.v = None#先不考虑初始优化问题,vanilla版本
self.momentum = momentum
def update(self,params,grads):
if self.v is None:
self.v = {}
for k,val in grads.items():#.items用法
# self.v[k] = grads[k]#写错了,初始化不是把grad赋值过去,只是做一个shape相同的0集
#两种写法都行
if 0:
self.v[k] = np.zeros_lize(params[k])
else:
self.v[k] = np.zeros_like(val)
for k in self.v.keys():
# self.v[k] -= self.lr * grads[k]#写错了,这还是SGD,而是-=的写法不能有动量衰减,必须用正常=
self.v[k] = self.v[k] * self.momentum - self.lr * grads[k]
params[k] += self.v[k]#一致性,只要v对grad都是减法,这里就是加法
最后就是拿来和SGD对比一下,同样的网络结构,同样是mnist,同样的其他条件,(额外的,momentum有个参数momentum,或者叫摩擦系数反比,或者就叫动量系数,其他如学习率等参数一致。)
网络见:https://blog.csdn.net/huqinweI987/article/details/103149203
本文代码见:https://github.com/huqinwei/python_deep_learning_introduction/blob/master/chap06_optimizer_Momentum.py
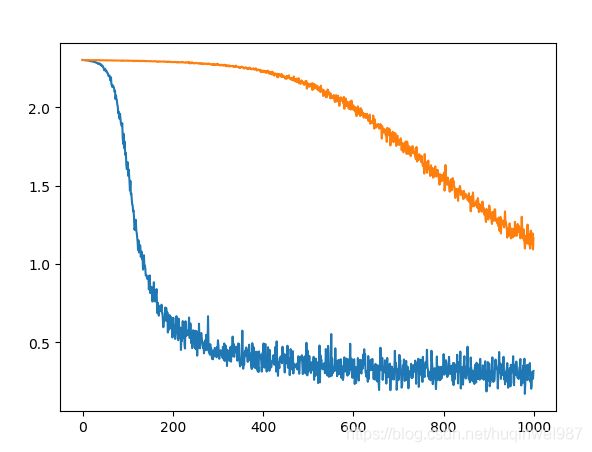
结果对比:batch=256,图一差距明显,图二迭代到10000次才逐渐拉小差距,但是也没能接近
附:Adagrad
注意的点也就是分母防0,还有学习率不能更新到h上,具体的代码见
https://github.com/huqinwei/python_deep_learning_introduction/blob/master/chap06_optimizer_Adagrad.py
class Adagrad():
def __init__(self,lr = 0.01):
self.lr = lr
self.h = None
def update(self,params,grads):
if self.h is None:
self.h = {}
for k,val in grads.items():#.items用法
#两种写法都行
if 0:
self.h[k] = np.zeros_lize(grads[k])
else:
self.h[k] = np.zeros_like(val)
for k in self.h.keys():
self.h[k] += grads[k] ** 2#分母不能加学习率
params[k] -= self.lr * grads[k] / (np.sqrt(self.h[k]) + 1e-7)#分母防0
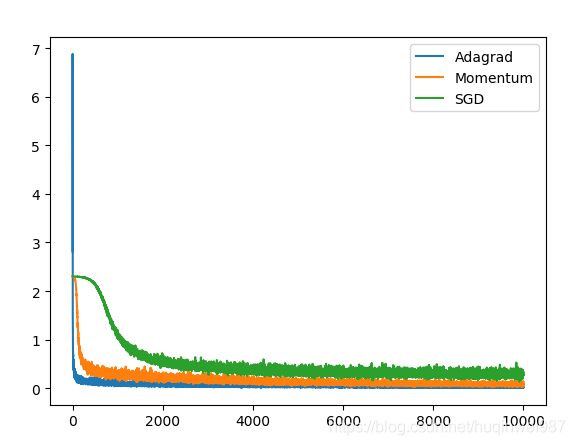
图一:错误实现版本(lr更新进了h)
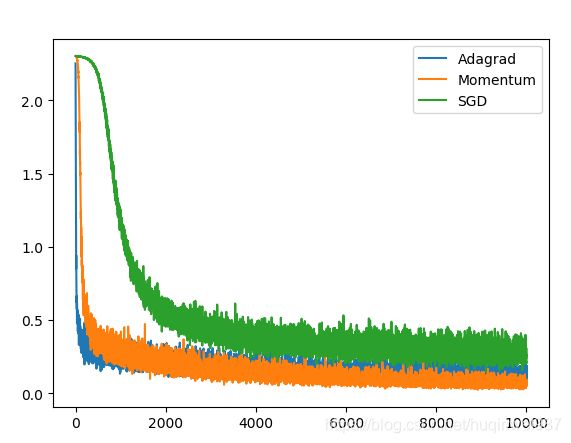
图二:正确版本对照
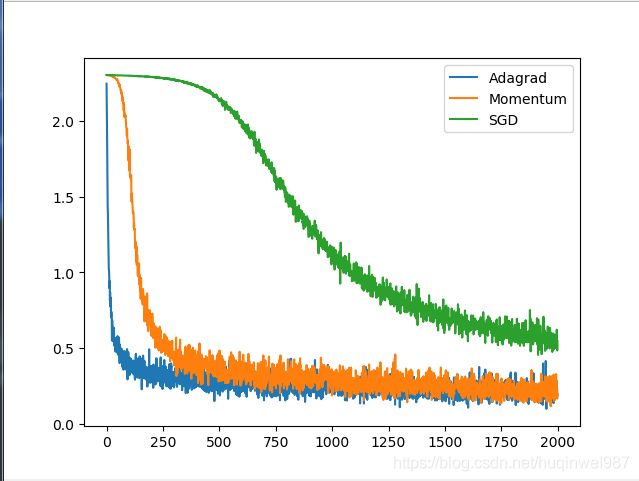
图三:正确版本前2000迭代图示
由图二可知,Adagrad后期学习率明显衰减,被Momentum追上。当然,Adagrad的优势不一定在这里体现,但是至少可以看到学习率降低的趋势,时间关系,不展开,这里主要是检验实现正确与否。Adagrad主要解决的是不同维度之间step的差异,在某些复杂情况下会比Momentum表现好。
RMSProp——“leaky Adagrad”:
因为Adagrad的分母是单调的,这样学习率衰减不可逆,最后会变0。这是个缺点,所以需要让它leaky,这样才会有学习率,才会有step。
class RMSProp():
def __init__(self,lr = 0.01,decay_rate = 0.9):
self.lr = lr
self.h = None
self.decay_rate = decay_rate
def update(self,params,grads):
if self.h is None:
self.h = {}
for k,val in grads.items():
self.h[k] = np.zeros_like(val)
for k in self.h.keys():
self.h[k] *= self.decay_rate
self.h[k] += (1 - self.decay_rate) * grads[k] ** 2#
params[k] -= self.lr * grads[k] / (np.sqrt(self.h[k]) + 1e-7)#如图:可以看到,Adagrad学习率衰减而被Momentum追上甚至有点反超的趋势,在RMSProp已经被逆转了。
但是小孩才做选择,成年人全都要——Adam。
声明,本书代码可能不是唯一甚至标准实现,所以会有些困惑,尤其参考代码中的lr_t。和我常见的如下版本就不同,感觉这个参考版本更直接,他的bias-correction(初始化偏差修正)很直接,而本例中加入了另一个因素。当然,原版Adam可能也不止一种实现,时间关系,暂时也不展开分析了。
根据描述先用直觉实现一版(结合Adagrad(RMSProp)与momentum),但是感觉直觉上不太好解释,因为分子是动量,之前的Adagrad的直觉解释是,一次导数比二次导数的简化版,但是动量怎么解释?动量可能方向和量级都不同~!
(代码中有bug,看完下边再参考)
class Adam():
def __init__(self,lr = 0.001,beta1 = 0.9,beta2 = 0.999):
self.lr = lr
self.v = None
self.m = None
self.beta1 = beta1
self.beta2 = beta2
def update(self,params,grads):
if self.v is None:
self.v = {}
for k,val in grads.items():
self.v[k] = np.zeros_like(val)
if self.m is None:
self.m = {}
for k,val in grads.items():
self.m[k] = np.zeros_like(val)
for k in self.h.keys():
self.m[k] = (self.m[k] * self.beta1) + ((1-self.beta1)*grads[k])#todo 正负号一致性检查
self.v[k] += (self.v[k] * self.beta2) + ((1 - self.beta2) * grads[k] ** 2)#bug!!!!!!!
params[k] -= self.lr * self.m[k] / (np.sqrt(self.v[k]) + 1e-7)#实测下来,发现效果不理想,很快就停滞了,我的直觉是,分子有个动量在,这样的step太大了?这个vanilla版本有一个改善没有做(初始几次迭代的权重补偿),但是感觉不是核心影响。核心问题是step太大,那么除了自己手动改一个decay出来,看看原书参考代码,他其实是做了学习率补偿,不过不是用的简单的decay,而是利用beta1和beta2做了一个公式,
self.iter += 1
lr_t = self.lr * np.sqrt(1.0 - self.beta2**self.iter) / (1.0 - self.beta1**self.iter) 直觉上怎么解释?分子有个根号,beta2是0.999,分子会是根号下0.001,0.002,0.003这样变化,分母会是0.1,0.19,.027这样变化,所以相对来说分子量级很稳定,分母在变大,但是变大的有限!(个人认为如果目的只是学习率衰减,分子分母通用一个beta也可以,当然,他也可能考虑了初始权重补偿,具体一小点差别先不纠结了,如果把两个beta反过来写,初期学习率其实还要更大,)
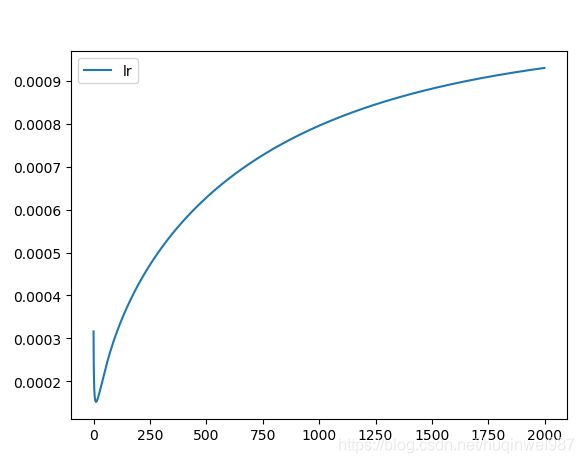
先打印看一下原始版本的学习率变化(所谓原始,对比的是如果我用其他参数组合来更新lr,放在尾部对比):
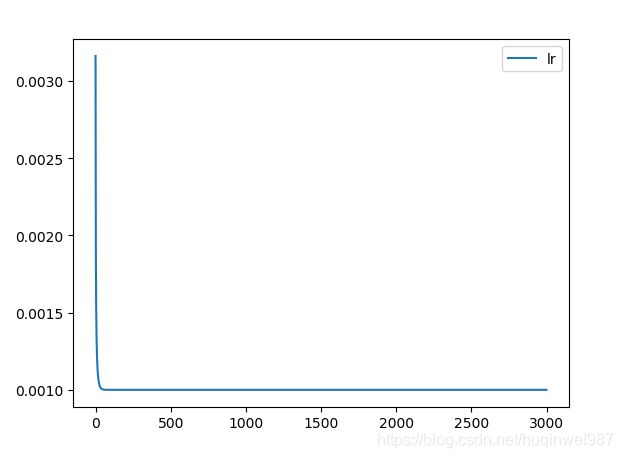
数量级方面:传进来的lr是0.001,下图是lr_t,后边没有的部分,在0.0009趋于0.001。
前10次迭代,从0.0003到0.00015,后边开始上升。
似乎,背道而驰了(至少可以肯定,学习率不是逐步降低的,不是我想的那个用处——均衡掉动量递增问题。事实上,因为有leaky的存在,似乎,动量递增也不是个问题?),问题出在哪?动量在增加,学习率在增加,而分母~~~~原来分母还写错了(从早期的adagrad改过来,没改掉+=),其实这才是错误原因!!!
self.v[k] += (self.v[k] * self.beta2) + ((1 - self.beta2) * grads[k] ** 2)#已经包含了self.v[k]*self.beta2的部分了,前边还用了+=符号这里不要纠结,用+=和用=都行,但是要保持一致(我觉得这种复杂公式就不要用+=了,乱),虽然下边的两种写法是等价的~
#self.m[key] = self.beta1*self.m[key] + (1-self.beta1)*grads[key]
#self.v[key] = self.beta2*self.v[key] + (1-self.beta2)*(grads[key]**2)
self.m[key] += (1 - self.beta1) * (grads[key] - self.m[key])
self.v[key] += (1 - self.beta2) * (grads[key]**2 - self.v[key])解决了这个错误之后,无论那个lr更新要不要,后期的结果都变好了!!!
lr_t修正版代码:
def update(self, params, grads):
if self.m is None:
self.m, self.v = {}, {}
for key, val in params.items():
self.m[key] = np.zeros_like(val)
self.v[key] = np.zeros_like(val)
self.iter += 1
lr_t = self.lr * np.sqrt(1.0 - self.beta2**self.iter) / (1.0 - self.beta1**self.iter)
for key in params.keys():
#self.m[key] = self.beta1*self.m[key] + (1-self.beta1)*grads[key]
#self.v[key] = self.beta2*self.v[key] + (1-self.beta2)*(grads[key]**2)
self.m[key] += (1 - self.beta1) * (grads[key] - self.m[key])
self.v[key] += (1 - self.beta2) * (grads[key]**2 - self.v[key])
params[key] -= lr_t * self.m[key] / (np.sqrt(self.v[key]) + 1e-7)
下面对比一下前期用不用lr修正的效果
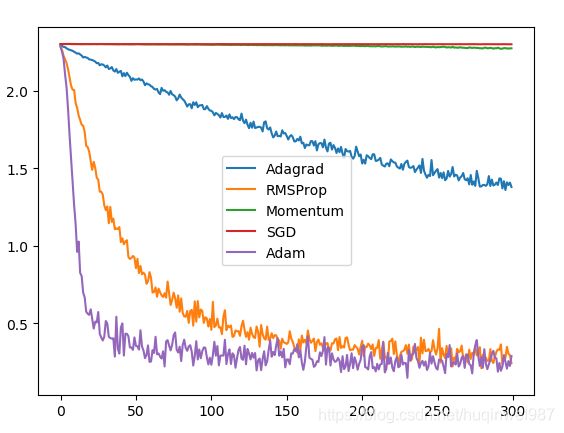
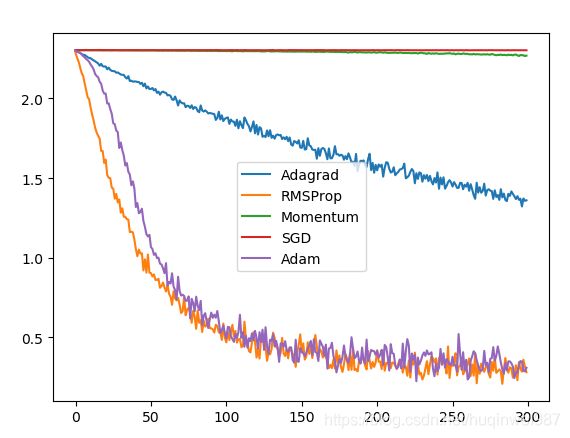
图一:不使用lr_t优化,图二:使用lr_t优化(单从这个图看,图二似乎,至少,没变得更好)
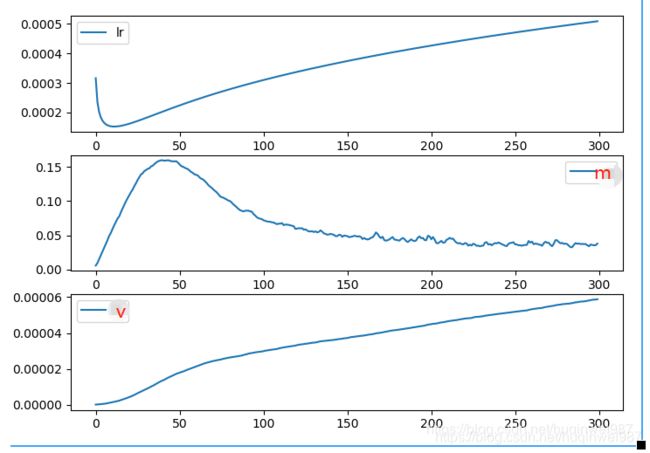
那么,Adam内的lr_t是干什么的呢?其实可能公式重新排列组合一下,结果会更清晰,lr_t其实应该和v的更新合并在一起,
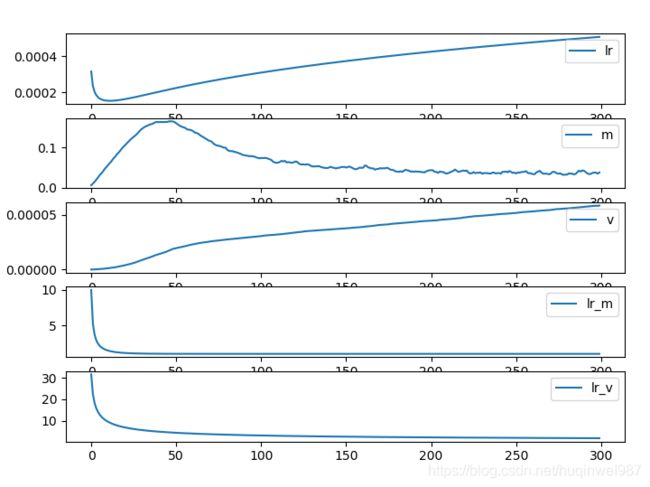
下图,v是分母,m是分子,lr是分子要乘以的系数,m*lr是完整的分子部分,lr和v趋势正好是一个互补,分母是逐渐变大的,分子经过互补之后如果是均匀的,那么整体的step就是逐渐变小,并且初期可能会大一些(因为m的上升),这,满足初期动量补偿的效果(图中,m和v只取W1作为参考,使用np.linalg.norm()得到矩阵范式)
其实不知道算不算巧合,我之前了解的bias-correction,动量前期的校正(可能是Momentum算法,Adam本来就是结核性的,所以可能有所不同),貌似只有分母部分:/ (1.0 - self.beta1**self.iter)) 。加入了分子np.sqrt(1.0 - self.beta2**self.iter)之后,(初期lr的那个下降)变得难以解释起来,如果忽略那一小段iters,整体是好解释的。(这个lr难解释就难在,他是两部分的合成,后边我会给出说明)
此图可以有nike赞助
我找来了论文的伪代码:
犯了个错误,我使用v,h,其他人都使用m,v,看来是惯例,应该遵守,不同名,分析起来很绕!在确认了beta1、beta2和参数1、参数2的对应关系后,下面我打算也用m、v来描述参数1和参数2.(为了方便阅读,前文也都改了)
在循环中,先计算梯度,然后完成mt和vt的更新,然后,mt和vt都做了bias-corrected,看来分子分母都要做。
最后就是更新,这里学习率清晰一些,没和其他的公式合并。分子分母就是mt和vt对应的该有的样子。
其实这里是能对应的:这个就是mt和vt分别的修正,分子系数beta1,lr_t中beta1是分母,刚好符合论文除以1-beta1**t的操作(唯一的区别,书中参考代码中(v相关的)beta2的补偿用了根号,不知道是刻意还是失误?因为beta2对应的v本来就是梯度平方?因为在总公式中v本来就被开根号?应该是后者,但是我没论文作者的真实代码,暂时无法验证),所以结论就是,这个lr_t是分子分母都做了初始动量补偿操作
lr_t = self.lr * np.sqrt(1.0 - self.beta2 ** self.iter) / (1.0 - self.beta1 ** self.iter) for k in self.h.keys():
#对参数名进行了修正,与前文不同
self.m[k] = (self.m[k] * self.beta1) + ((1-self.beta1)*grads[k])#todo 正负号一致性检查
self.v[k] = (self.v[k] * self.beta2) + ((1 - self.beta2) * grads[k] ** 2)#
params[k] -= (lr_t * self.m[k]) / (np.sqrt(self.v[k]) + 1e-7)#那么lr_t拆解清了,应该重新打印跟踪指标了
我进行拆解之后,lr_m和lr_v就一致了,都是初期高的一个补偿(合并到lr会出现“nike”是因为,他们分别是分子和分母,两个分别是0.9和0.999,补偿倍率不一致出现的波动,没有太多意义)
相关代码
https://github.com/huqinwei/python_deep_learning_introduction/chap06_optimizer_Adam.py
网络结构也在总目录下
https://github.com/huqinwei/python_deep_learning_introduction/
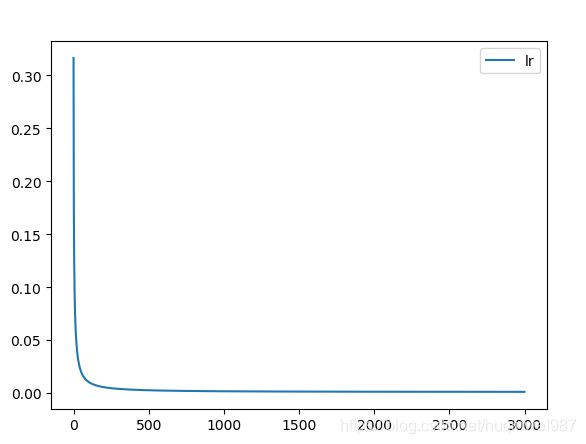
附:lr更新公式使用不同的beta组合对应的效果
如果不考虑初始的补偿或者其他因素,我感觉似乎3、4更好呢,但是只是从lr的角度,其实lr可以通过外部decay来处理,此处核心目的应该不是lr decay(最终趋近于1,也证明它无意干涉真正的lr)
默认方法:lr_t = self.lr * np.sqrt(1.0 - self.beta2 ** self.iter) / (1.0 - self.beta1 ** self.iter)lr_t = self.lr * np.sqrt(1.0 - self.beta1 ** self.iter) / (1.0 - self.beta1 ** self.iter)lr_t = self.lr * np.sqrt(1.0 - self.beta2 ** self.iter) / (1.0 - self.beta2 ** self.iter)lr_t = self.lr * np.sqrt(1.0 - self.beta1 ** self.iter) / (1.0 - self.beta2 ** self.iter)