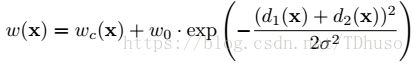- 03 数据可视化的世界非常广阔,除了已提到的类型,还有许多更细分或前沿的可视化形式。
晨曦543210
信息可视化人工智能
十五、机器学习与数据科学专用图表特征重要性图(FeatureImportancePlot)用途:展示机器学习模型中各特征对预测结果的贡献度。示例:随机森林模型中影响房价预测的关键因素。混淆矩阵热力图(ConfusionMatrixHeatmap)用途:分类模型性能评估,显示预测结果与真实标签的对比。示例:疾病诊断模型的真阳性/假阳性分布。学习曲线(LearningCurve)用途:分析模型训练过程
- 当争论者还在讨论AI的边界,实践者早已用这些技术解决实际问题
渡难繁辰
人工智能拥抱AI人工智能ai
——普通人参与AI革命的关键路径一、AI应用五大核心组件(通俗拆解版)1️⃣LLM:AI的「决策核心」本质:大型语言模型(如DeepSeek、通义千问),具备语言理解与生成能力能力边界:✅处理文本类任务(写作/翻译/摘要)❌无法获取实时信息(如最新股价)⚠️存在“幻觉”(虚构信息)风险案例对比:问:“鲁迅和周树人什么关系?”基础LLM:“两位都是著名作家”(错误)增强版LLM:“周树人是鲁迅本名”
- 全球 AI HR 浪潮下的中国实践:从效率革命到战略重构
weixin_54980836
人工智能重构
一、全球AIHR的技术跃迁与价值重构在DeepSeek、ChatGPT引发的生成式AI革命中,人力资源管理领域正经历着从“工具替代”到“认知重构”的范式转变。Gartner《2025年人力资源技术趋势报告》指出,AI在HR场景的应用已从简历筛选、薪资计算等基础效率工具,升级为支持组织战略决策的“数字伙伴”。这种转变的底层逻辑,源于大模型技术带来的三大突破:多模态交互能力:AI已能同时处理文本、语音
- SpringBoot多数据源动态切换方案:AbstractRoutingDataSource详解
fanxbl957
Webspringboot后端java
博主介绍:Java、Python、js全栈开发“多面手”,精通多种编程语言和技术,痴迷于人工智能领域。秉持着对技术的热爱与执着,持续探索创新,愿在此分享交流和学习,与大家共进步。DeepSeek-行业融合之万象视界(附实战案例详解100+)全栈开发环境搭建运行攻略:多语言一站式指南(环境搭建+运行+调试+发布+保姆级详解)感兴趣的可以先收藏起来,希望帮助更多的人SpringBoot多数据源动态切换
- 构建企业级大模型运行监控体系:健康度五级指标与实战部署路径全解析
构建企业级大模型运行监控体系:健康度五级指标与实战部署路径全解析关键词:模型运行监控、健康度分级体系、DeepSeek、私有化部署、Prometheus、Grafana、异常检测、推理稳定性、性能观测、可视化大屏摘要:在DeepSeek大模型私有化部署的生产环境中,传统的“是否可用”监控已难以满足对模型稳定性、推理质量与异常风险的精细管理需求。为此,企业必须构建一套基于五级健康度模型的全维监控体系
- 企业级多模型服务架构(MaaS)私有部署实战指南:统一调度、模型隔离与服务编排全路径解析
观熵
架构人工智能私有化部署
企业级多模型服务架构(MaaS)私有部署实战指南:统一调度、模型隔离与服务编排全路径解析关键词:多模型服务架构、MaaS、私有化部署、模型管理、推理调度、模型编排、TritonInferenceServer、DeepSeek、模型隔离、企业级AI平台摘要:随着企业对多任务、多模型能力的需求日益增长,MaaS(Model-as-a-Service)架构已成为私有部署中的关键支撑技术之一。该文聚焦当前
- 【人工智能】ChatGPT、DeepSeek-R1、DeepSeek-V3 辨析
G皮T
#大语言模型人工智能LLM大语言模型chatgptdeepseekDeepSeek-R1DeepSeek-V3
ChatGPT、DeepSeek-R1、DeepSeek-V3辨析1.ChatGPT对比DeepSeek1.1技术相似点1.2主要差异1.3关键区别1.4如何选择1.5总结2.DeepSeek-R1对比DeepSeek-V32.1DeepSeek-R12.2DeepSeek-V32.3核心区别总结2.4如何选择3.R1和V3有什么含义3.1DeepSeekR1的"R"3.2DeepSeekV3的"
- AlphaEvolve:谷歌的算法进化引擎 | 从数学证明到芯片设计的AI自主发现新纪元
大千AI助手
人工智能Python#OTHER算法人工智能深度学习AlphaEvolvegooglegemini
AlphaEvolve:谷歌的算法进化引擎|从数学证明到芯片设计的AI自主发现新纪元——结合大语言模型与进化计算,重塑科学发现与工程优化的通用智能体本文由「大千AI助手」原创发布,专注用真话讲AI,回归技术本质。拒绝神话或妖魔化。搜索「大千AI助手」关注我,一起撕掉过度包装,学习真实的AI技术!⚙️一、核心定义与技术架构AlphaEvolve是由谷歌DeepMind开发的通用科学AI智能体,其核心
- The Illusion of Thinking: Understanding the Strengths and Limitations of Reasoning Models
UnknownBody
LLMDailyCausalandReasoning语言模型人工智能
文章主要内容总结本文围绕大推理模型(LRMs)的推理能力展开系统研究,通过可控谜题环境分析其在不同问题复杂度下的表现,揭示其优势与局限性:研究背景与问题:当前LRMs(如OpenAIo1/o3、DeepSeek-R1等)虽在推理基准测试中表现提升,但对其底层能力、缩放特性及局限性的理解不足。现有评估依赖数学和编码基准,存在数据污染且缺乏对推理轨迹的深度分析。研究方法:采用可控谜题环境(如汉诺塔、跳
- DeepSeek 帮助自己的工作
引言简述人工智能助手在职场中的普及趋势DeepSeek作为智能创作助手的核心功能概述DeepSeek的核心能力信息检索与整合:基于用户意图精准搜索并生成答案多场景应用:技术文档撰写、数据分析、代码生成等交互优化:遵循用户指定的格式与内容规范职场应用场景与实操案例技术文档撰写自动生成API文档框架根据需求补充技术细节示例代码块与公式的规范化输出数据分析支持快速检索行业数据并生成可视化建议数学建模中的
- 【DeepSeek实战】17、MCP地图服务集成全景指南:高德、百度、腾讯三大平台接入实战
引言:为什么MCP是地图服务的下一代革命?在数字化时代,位置服务已成为电商、出行、物流等行业的核心基础设施。但单一地图服务商的局限性日益凸显:某外卖平台因高德地图API突发故障导致30分钟订单配送延迟,某打车软件因百度地图路线规划偏差引发用户投诉激增,某物流企业因腾讯地图POI数据不全导致仓库定位错误……这些问题的根源,在于缺乏一套能统一管理多地图服务的智能平台。MCP(地图控制平台)应运而生。它
- Python机器学习与深度学习:决策树、随机森林、XGBoost与LightGBM、迁移学习、循环神经网络、长短时记忆网络、时间卷积网络、自编码器、生成对抗网络、YOLO目标检测等
WangYan2022
机器学习/深度学习Python机器学习深度学习随机森林迁移学习
融合最新技术动态与实战经验,旨在系统提升以下能力:①掌握ChatGPT、DeepSeek等大语言模型在代码生成、模型调试、实验设计、论文撰写等方面的实际应用技巧②深入理解深度学习与经典机器学习算法的关联与差异,掌握其理论基础③熟练运用PyTorch实现各类深度学习模型,包括迁移学习、循环神经网络(RNN)、长短时记忆网络(LSTM)、时间卷积网络(TCN)、自编码器、生成对抗网络(GAN)、YOL
- DeepSeek API 完整调用指南:从基础到高级应用实战
独立开发者阿乐
原创DeepseekapiAPI调用api接口核心能力模型DeepSeekAPI
文章目录DeepSeekAPI调用全流程详解:从入门到生产级实践1.DeepSeekAPI概述1.1DeepSeekAPI核心能力1.2API版本与计费2.API调用准备工作2.1注册与认证2.2环境准备2.3API密钥管理3.基础API调用实现3.1RESTfulAPI调用3.2流式响应处理4.高级API使用技巧4.1多轮对话管理4.2文件上传与处理5.生产环境最佳实践5.1错误处理与重试机制5
- DeepSeek API生产级集成策略:性能优化与成本控制指南
独立开发者阿乐
原创性能优化DeepSeekAPIAPI集成API核心服务层封装
文章目录深度集成DeepSeekAPI:从接入到生产级应用实践引言一、DeepSeekAPI概述1.1DeepSeekAPI核心能力1.2API版本与定价二、项目集成方案设计2.1技术选型考量2.2架构设计模式2.2.1直接调用模式2.2.2服务层封装模式2.2.3异步微服务模式三、核心实现与优化3.1认证与安全3.1.1API密钥管理3.2性能优化技巧3.2.1流式响应处理3.2.2批量请求处理
- deepin uos 安装 vscode 开发工具提示缺失依赖包 libxkbfile1 的解决方案
deepinuos安装vscode开发工具提示缺失依赖包libxkbfile1的解决方案近日在deepin系统上安装vscode遇到如下报错:解决方法如下:下载高版本libxkbfile1安装高版本libxkbfile1安装vscode下载高版本libxkbfile1wgethttps://mirrors.ustc.edu.cn/debian/pool/main/libx/libxkbfile/l
- 优麒麟安装向日葵遇到的依赖缺少问题 未安装软件包 libwebkitgtk-3.0-0
orange_cy
linux
错误提示:未安装软件包libwebkitgtk-3.0-0在处理时有错误发生:sunloginclient具体错误如下图:1.前往官网下载向日葵软件官网地址:向日葵远程控制软件_app下载_视频监控软件_远程开机软件下载-Oray贝锐科技向日葵1.根据所使用的平台选择的位置linux2.选择:Ubuntu/Deepin下载最新文件安装包:SunloginClient-10.1.1.38139_am
- 多智能体深度强化学习:一项综述 Multi-agent deep reinforcement learning: a survey
资源存储库
笔记
Abstract抽象Theadvancesinreinforcementlearninghaverecordedsublimesuccessinvariousdomains.Althoughthemulti-agentdomainhasbeenovershadowedbyitssingle-agentcounterpartduringthisprogress,multi-agentreinforc
- ReAct (Reason and Act) OR 强化学习(Reinforcement Learning, RL)
SugarPPig
人工智能人工智能
这个问题触及了现代AI智能体(Agent)构建的两种核心思想。简单来说,ReAct是一种“调用专家”的模式,而强化学习(RL)是一种“从零试错”的模式。为了让你更清晰地理解,我们从一个生动的比喻开始,然后进行详细的对比。一个生动的比喻想象一下你要完成一项复杂的任务,比如“策划一场完美的生日派对”。ReAct的方式(像一位经验丰富的活动策划师)你是一位知识渊博的专家(大语言模型LLM)。你首先会思考
- DeepSeek 部署中的常见问题及解决方案
tonngw
后端
技术文章大纲:DeepSeek部署中的常见问题及解决方案引言简要介绍DeepSeek及其应用场景,说明部署过程中可能遇到的挑战。环境配置问题硬件兼容性问题:GPU型号、驱动版本不匹配的解决方案。依赖库冲突:Python版本、CUDA/cuDNN不兼容的排查方法。虚拟环境配置:Anaconda或Docker环境的最佳实践。模型加载与初始化问题预训练模型下载失败:网络代理设置、手动下载替代方案。显存不
- Python机器学习入门必看!从原理到实战,手把手教你线性回归模型
小张在编程
python机器学习线性回归
引言在人工智能浪潮席卷全球的今天,机器学习(MachineLearning)早已不再是实验室的“黑科技”——打开购物APP的“猜你喜欢”、输入搜索词后的“相关推荐”、甚至天气预报中的温度预测,背后都有机器学习模型的身影。而在线性回归(LinearRegression)作为机器学习中最基础、最经典的监督学习模型,堪称机器学习的“敲门砖”。本文将从原理到实战,带你彻底掌握这一核心算法。一、机器学习的“
- AI办公Agent之Skywork Super Agents
NeilNiu
AI+开源项目人工智能
5月22日,昆仑万维科技股份有限公司(以下简称“昆仑万维”)面向全球市场同步发布天工超级智能体(SkyworkSuperAgents)。官网地址:https://www.tiangong.cn/这个工具主要采用了AIagent架构和DeepResearch技术,一站式生成文档、PPT、表格(excel)、网页、播客和音视频多模态内容。它具有强大的DeepResearch能力,在GAIA榜单排名全球
- 满血DeepSeek加持的AlphaGPT,助力高文律师事务所全面拥抱AI
2025年初,中国团队精心雕琢的通用大模型DeepSeek凭借其创新的架构优化以及深入的数据挖掘技术,在逻辑推理、多轮对话和知识搜索等关键领域大放异彩,其为诸多垂直领域,特别是法律行业的智能化转型,开拓了全新的方向。2月8日,法律科技领域的领军者iCourt将旗下的AlphaGPT与DeepSeek深度融合,重磅推出业内首款“DeepSeek+法律专业”AI大模型。这一创举彻底打破了传统法律智能工
- AI人工智能助力联邦学习通信效率优化的解决方案
AI智能应用
人工智能ai
AI驱动的联邦学习通信效率优化:从理论到实践的全面解决方案元数据框架标题AI驱动的联邦学习通信效率优化:从理论到实践的全面解决方案关键词联邦学习(FederatedLearning)、通信优化(CommunicationEfficiency)、AI赋能(AI-Enabled)、参数压缩(ParameterCompression)、客户端选择(ClientSelection)、联邦蒸馏(Federa
- 大语言模型(LLM)课程学习(Curriculum Learning)、数据课程(data curriculum)指南:从原理到实践
在人工智能的浪潮之巅,我们总会惊叹于GPT-4、Llama3.1、Qwen2.5这些顶尖大语言模型(LLM)所展现出的惊人能力。它们似乎无所不知,能写诗、能编程、能进行复杂的逻辑推理。一个自然而然的问题是:它们是如何“学”会这一切的?大多数人会回答:“用海量数据喂出来的。”这个答案只说对了一半。如果你认为只要把互联网上能找到的所有数据(比如15万亿个token)随机打乱,然后“一锅烩”地喂给模型,
- 人工智能学习资源
Hemy08
人工智能学习
无机器学习基础:https://www.coursera.org/learn/machine-learning有机器学习基础:MachineYearning深度学习入门:https://www.coursera.org/learn/neural-networks-deep-learning
- 【DeepSeek开源周】Day 4:DualPipe & EPLB 学习笔记
蓝海星梦
DeepSeek开源周探秘开源学习笔记人工智能云计算分布式
目录一、DualPipe&EPLB概述二、DualPipe详解1.流水线并行策略(1)F-then-B策略(2)1F1B策略2.朴素流水线并行3.GPipe微批次流水线并行4.PipeStream5.ZBPP6.DualPipe7.DualPipeV8.流水线并行方案对比三、EPLB详解1.专家并行(EP)2.EPLB冗余专家策略3.负载均衡策略(1)分层负载均衡(2)全局负载均衡(3)接口和示例
- 蓝桥杯2023国赛-01游戏
weixin_66009678
蓝桥杯游戏深度优先
本题的dfs还是比较复杂的,需要兼顾的条件比较多。这一题是我看了其他人的题才写出来的,菜菜捏,哈哈哈哈哈!!!然后这一题如何确保行之前不相同,是通过二进制来的,具体的在代码中给出了详解。直接上代码://https://www.lanqiao.cn/problems/17100/learning/?page=1&first_category_id=1&sort=students_count&tags
- 【论文阅读】Dynamic Few-Shot Visual Learning without Forgetting
Bosenya12
论文阅读
系统概述如下:(a)一个基于卷积神经网络(ConvNet)的识别模型,该模型包含特征提取器和分类器;(b)一个少样本分类权重生成器。这两个组件都是在一组基础类别上训练的,我们为这些类别准备了大量训练数据。在测试阶段,权重生成器会接收少量新类别的训练数据以及基础类别的分类权重向量(分类器框内的绿色矩形),并为新类别生成相应的分类权重向量(分类器框内的蓝色矩形)。这样,卷积神经网络就能同时识别基础类别
- 【AI论文】GLM-4.1V-思考:借助可扩展强化学习实现通用多模态推理
东临碣石82
人工智能
摘要:我们推出GLM-4.1V-Thinking这一视觉语言模型(VLM),该模型旨在推动通用多模态推理的发展。在本报告中,我们分享了在以推理为核心的训练框架开发过程中的关键发现。我们首先通过大规模预训练开发了一个具备显著潜力的高性能视觉基础模型,可以说该模型为最终性能设定了上限。随后,借助课程采样强化学习(ReinforcementLearningwithCurriculumSampling,R
- 结合 deepseek R1 模型,新的 AI Cursor 编程最佳实践!让第三方 ai 成为我们和 cursor 沟通的桥梁
hello,我是魔王哒,流光卡片开发者,来分享一下自己最新的ai编程实践经验,不管你是零基础小白还是专业技术人这里的思路相信一定会对你有启发。一句话总结,让deepseek成为你与cursor沟通的桥梁,让其他deepseek来将作为编程小白的我们的语言转换为更适合与cursor对话的提示词。有人要问了,这能说是最佳实践吗?是不是不知道,但是他解决了很多技术人或者小白的痛点,那就是不知道该如何好好
- mongodb3.03开启认证
21jhf
mongodb
下载了最新mongodb3.03版本,当使用--auth 参数命令行开启mongodb用户认证时遇到很多问题,现总结如下:
(百度上搜到的基本都是老版本的,看到db.addUser的就是,请忽略)
Windows下我做了一个bat文件,用来启动mongodb,命令行如下:
mongod --dbpath db\data --port 27017 --directoryperdb --logp
- 【Spark103】Task not serializable
bit1129
Serializable
Task not serializable是Spark开发过程最令人头疼的问题之一,这里记录下出现这个问题的两个实例,一个是自己遇到的,另一个是stackoverflow上看到。等有时间了再仔细探究出现Task not serialiazable的各种原因以及出现问题后如何快速定位问题的所在,至少目前阶段碰到此类问题,没有什么章法
1.
package spark.exampl
- 你所熟知的 LRU(最近最少使用)
dalan_123
java
关于LRU这个名词在很多地方或听说,或使用,接下来看下lru缓存回收的实现
1、大体的想法
a、查询出最近最晚使用的项
b、给最近的使用的项做标记
通过使用链表就可以完成这两个操作,关于最近最少使用的项只需要返回链表的尾部;标记最近使用的项,只需要将该项移除并放置到头部,那么难点就出现 你如何能够快速在链表定位对应的该项?
这时候多
- Javascript 跨域
周凡杨
JavaScriptjsonp跨域cross-domain
- linux下安装apache服务器
g21121
apache
安装apache
下载windows版本apache,下载地址:http://httpd.apache.org/download.cgi
1.windows下安装apache
Windows下安装apache比较简单,注意选择路径和端口即可,这里就不再赘述了。 2.linux下安装apache:
下载之后上传到linux的相关目录,这里指定为/home/apach
- FineReport的JS编辑框和URL地址栏语法简介
老A不折腾
finereportweb报表报表软件语法总结
JS编辑框:
1.FineReport的js。
作为一款BS产品,browser端的JavaScript是必不可少的。
FineReport中的js是已经调用了finereport.js的。
大家知道,预览报表时,报表servlet会将cpt模板转为html,在这个html的head头部中会引入FineReport的js,这个finereport.js中包含了许多内置的fun
- 根据STATUS信息对MySQL进行优化
墙头上一根草
status
mysql 查看当前正在执行的操作,即正在执行的sql语句的方法为:
show processlist 命令
mysql> show global status;可以列出MySQL服务器运行各种状态值,我个人较喜欢的用法是show status like '查询值%';一、慢查询mysql> show variab
- 我的spring学习笔记7-Spring的Bean配置文件给Bean定义别名
aijuans
Spring 3
本文介绍如何给Spring的Bean配置文件的Bean定义别名?
原始的
<bean id="business" class="onlyfun.caterpillar.device.Business">
<property name="writer">
<ref b
- 高性能mysql 之 性能剖析
annan211
性能mysqlmysql 性能剖析剖析
1 定义性能优化
mysql服务器性能,此处定义为 响应时间。
在解释性能优化之前,先来消除一个误解,很多人认为,性能优化就是降低cpu的利用率或者减少对资源的使用。
这是一个陷阱。
资源时用来消耗并用来工作的,所以有时候消耗更多的资源能够加快查询速度,保持cpu忙绿,这是必要的。很多时候发现
编译进了新版本的InnoDB之后,cpu利用率上升的很厉害,这并不
- 主外键和索引唯一性约束
百合不是茶
索引唯一性约束主外键约束联机删除
目标;第一步;创建两张表 用户表和文章表
第二步;发表文章
1,建表;
---用户表 BlogUsers
--userID唯一的
--userName
--pwd
--sex
create
- 线程的调度
bijian1013
java多线程thread线程的调度java多线程
1. Java提供一个线程调度程序来监控程序中启动后进入可运行状态的所有线程。线程调度程序按照线程的优先级决定应调度哪些线程来执行。
2. 多数线程的调度是抢占式的(即我想中断程序运行就中断,不需要和将被中断的程序协商)
a)
- 查看日志常用命令
bijian1013
linux命令unix
一.日志查找方法,可以用通配符查某台主机上的所有服务器grep "关键字" /wls/applogs/custom-*/error.log
二.查看日志常用命令1.grep '关键字' error.log:在error.log中搜索'关键字'2.grep -C10 '关键字' error.log:显示关键字前后10行记录3.grep '关键字' error.l
- 【持久化框架MyBatis3一】MyBatis版HelloWorld
bit1129
helloworld
MyBatis这个系列的文章,主要参考《Java Persistence with MyBatis 3》。
样例数据
本文以MySQL数据库为例,建立一个STUDENTS表,插入两条数据,然后进行单表的增删改查
CREATE TABLE STUDENTS
(
stud_id int(11) NOT NULL AUTO_INCREMENT,
- 【Hadoop十五】Hadoop Counter
bit1129
hadoop
1. 只有Map任务的Map Reduce Job
File System Counters
FILE: Number of bytes read=3629530
FILE: Number of bytes written=98312
FILE: Number of read operations=0
FILE: Number of lar
- 解决Tomcat数据连接池无法释放
ronin47
tomcat 连接池 优化
近段时间,公司的检测中心报表系统(SMC)的开发人员时不时找到我,说用户老是出现无法登录的情况。前些日子因为手头上 有Jboss集群的测试工作,发现用户不能登录时,都是在Tomcat中将这个项目Reload一下就好了,不过只是治标而已,因为大概几个小时之后又会 再次出现无法登录的情况。
今天上午,开发人员小毛又找到我,要我协助将这个问题根治一下,拖太久用户难保不投诉。
简单分析了一
- java-75-二叉树两结点的最低共同父结点
bylijinnan
java
import java.util.LinkedList;
import java.util.List;
import ljn.help.*;
public class BTreeLowestParentOfTwoNodes {
public static void main(String[] args) {
/*
* node data is stored in
- 行业垂直搜索引擎网页抓取项目
carlwu
LuceneNutchHeritrixSolr
公司有一个搜索引擎项目,希望各路高人有空来帮忙指导,谢谢!
这是详细需求:
(1) 通过提供的网站地址(大概100-200个网站),网页抓取程序能不断抓取网页和其它类型的文件(如Excel、PDF、Word、ppt及zip类型),并且程序能够根据事先提供的规则,过滤掉不相干的下载内容。
(2) 程序能够搜索这些抓取的内容,并能对这些抓取文件按照油田名进行分类,然后放到服务器不同的目录中。
- [通讯与服务]在总带宽资源没有大幅增加之前,不适宜大幅度降低资费
comsci
资源
降低通讯服务资费,就意味着有更多的用户进入,就意味着通讯服务提供商要接待和服务更多的用户,在总体运维成本没有由于技术升级而大幅下降的情况下,这种降低资费的行为将导致每个用户的平均带宽不断下降,而享受到的服务质量也在下降,这对用户和服务商都是不利的。。。。。。。。
&nbs
- Java时区转换及时间格式
Cwind
java
本文介绍Java API 中 Date, Calendar, TimeZone和DateFormat的使用,以及不同时区时间相互转化的方法和原理。
问题描述:
向处于不同时区的服务器发请求时需要考虑时区转换的问题。譬如,服务器位于东八区(北京时间,GMT+8:00),而身处东四区的用户想要查询当天的销售记录。则需把东四区的“今天”这个时间范围转换为服务器所在时区的时间范围。
- readonly,只读,不可用
dashuaifu
jsjspdisablereadOnlyreadOnly
readOnly 和 readonly 不同,在做js开发时一定要注意函数大小写和jsp黄线的警告!!!我就经历过这么一件事:
使用readOnly在某些浏览器或同一浏览器不同版本有的可以实现“只读”功能,有的就不行,而且函数readOnly有黄线警告!!!就这样被折磨了不短时间!!!(期间使用过disable函数,但是发现disable函数之后后台接收不到前台的的数据!!!)
- LABjs、RequireJS、SeaJS 介绍
dcj3sjt126com
jsWeb
LABjs 的核心是 LAB(Loading and Blocking):Loading 指异步并行加载,Blocking 是指同步等待执行。LABjs 通过优雅的语法(script 和 wait)实现了这两大特性,核心价值是性能优化。LABjs 是一个文件加载器。RequireJS 和 SeaJS 则是模块加载器,倡导的是一种模块化开发理念,核心价值是让 JavaScript 的模块化开发变得更
- [应用结构]入口脚本
dcj3sjt126com
PHPyii2
入口脚本
入口脚本是应用启动流程中的第一环,一个应用(不管是网页应用还是控制台应用)只有一个入口脚本。终端用户的请求通过入口脚本实例化应用并将将请求转发到应用。
Web 应用的入口脚本必须放在终端用户能够访问的目录下,通常命名为 index.php,也可以使用 Web 服务器能定位到的其他名称。
控制台应用的入口脚本一般在应用根目录下命名为 yii(后缀为.php),该文
- haoop shell命令
eksliang
hadoophadoop shell
cat
chgrp
chmod
chown
copyFromLocal
copyToLocal
cp
du
dus
expunge
get
getmerge
ls
lsr
mkdir
movefromLocal
mv
put
rm
rmr
setrep
stat
tail
test
text
- MultiStateView不同的状态下显示不同的界面
gundumw100
android
只要将指定的view放在该控件里面,可以该view在不同的状态下显示不同的界面,这对ListView很有用,比如加载界面,空白界面,错误界面。而且这些见面由你指定布局,非常灵活。
PS:ListView虽然可以设置一个EmptyView,但使用起来不方便,不灵活,有点累赘。
<com.kennyc.view.MultiStateView xmlns:android=&qu
- jQuery实现页面内锚点平滑跳转
ini
JavaScripthtmljqueryhtml5css
平时我们做导航滚动到内容都是通过锚点来做,刷的一下就直接跳到内容了,没有一丝的滚动效果,而且 url 链接最后会有“小尾巴”,就像#keleyi,今天我就介绍一款 jquery 做的滚动的特效,既可以设置滚动速度,又可以在 url 链接上没有“小尾巴”。
效果体验:http://keleyi.com/keleyi/phtml/jqtexiao/37.htmHTML文件代码:
&
- kafka offset迁移
kane_xie
kafka
在早前的kafka版本中(0.8.0),offset是被存储在zookeeper中的。
到当前版本(0.8.2)为止,kafka同时支持offset存储在zookeeper和offset manager(broker)中。
从官方的说明来看,未来offset的zookeeper存储将会被弃用。因此现有的基于kafka的项目如果今后计划保持更新的话,可以考虑在合适
- android > 搭建 cordova 环境
mft8899
android
1 , 安装 node.js
http://nodejs.org
node -v 查看版本
2, 安装 npm
可以先从 https://github.com/isaacs/npm/tags 下载 源码 解压到
- java封装的比较器,比较是否全相同,获取不同字段名字
qifeifei
非常实用的java比较器,贴上代码:
import java.util.HashSet;
import java.util.List;
import java.util.Set;
import net.sf.json.JSONArray;
import net.sf.json.JSONObject;
import net.sf.json.JsonConfig;
i
- 记录一些函数用法
.Aky.
位运算PHP数据库函数IP
高手们照旧忽略。
想弄个全天朝IP段数据库,找了个今天最新更新的国内所有运营商IP段,copy到文件,用文件函数,字符串函数把玩下。分割出startIp和endIp这样格式写入.txt文件,直接用phpmyadmin导入.csv文件的形式导入。(生命在于折腾,也许你们觉得我傻X,直接下载人家弄好的导入不就可以,做自己的菜鸟,让别人去说吧)
当然用到了ip2long()函数把字符串转为整型数
- sublime text 3 rust
wudixiaotie
Sublime Text
1.sublime text 3 => install package => Rust
2.cd ~/.config/sublime-text-3/Packages
3.mkdir rust
4.git clone https://github.com/sp0/rust-style
5.cd rust-style
6.cargo build --release
7.ctrl
![]() ”表示卷积层(conv 3×3, ReLu),"
”表示卷积层(conv 3×3, ReLu),"![]() "表示裁剪和复制,“
"表示裁剪和复制,“![]() ”表示池化层(max pool 2×2),"
”表示池化层(max pool 2×2),"![]() "表示上采样(up-conv 2×2),“
"表示上采样(up-conv 2×2),“![]() ”表示卷积层(conv 1×1)。整个网络有19次卷积操作,4次池化操作,4次上采样操作,4次裁剪和复制操作。卷积层使用的是“valid ,padding=0, stride=1”的模式进行卷积,所以最终得到的输出图像要小于原始图像。若想得到与原图像同样尺寸的输出图像,可以在卷积操作,时使用“same”模式。
”表示卷积层(conv 1×1)。整个网络有19次卷积操作,4次池化操作,4次上采样操作,4次裁剪和复制操作。卷积层使用的是“valid ,padding=0, stride=1”的模式进行卷积,所以最终得到的输出图像要小于原始图像。若想得到与原图像同样尺寸的输出图像,可以在卷积操作,时使用“same”模式。![]() 指在第
指在第![]() 个通道中位置为
个通道中位置为![]() 的值,
的值,![]() 为分类的类别数。
为分类的类别数。![]() ,
,![]() 每个像素值的权重值,对重要的像素点给与更大的关注。
每个像素值的权重值,对重要的像素点给与更大的关注。![]() 为输入的分割图像掩码,如图fig.3.c, d1(x)为像素x到离它最近的细胞的距离,d2(x)为像素x到离它第二近的细胞的距离。在他们的实验中,设置
为输入的分割图像掩码,如图fig.3.c, d1(x)为像素x到离它最近的细胞的距离,d2(x)为像素x到离它第二近的细胞的距离。在他们的实验中,设置![]() 以及
以及![]() 。
。