SparkR的安装配置以及数据分析
1.1. R与Rstudio的安装
1.1.1. R的安装
我们的工作环境都是在Ubuntu下操作的,所以只介绍Ubuntu下安装R的方法:
1) 在/etc/apt/sources.list添加源
deb http://mirror.bjtu.edu.cn/cran/bin/linux/ubuntu precise/,
然后更新源apt-get update;
2) 通过apt-get安装:
sudo apt-get install r-base
1.1.2. Rstudio的安装
官网有详细介绍:
http://www.rstudio.com/products/rstudio/download-server/
sudo apt-get install gdebi-core
sudo apt-get install libapparmor1 # Required only for Ubuntu, not Debian
wget http://download2.rstudio.org/rstudio-server-0.97.551-amd64.deb
sudo gdebi rstudio-server-0.97.551-amd64.deb
1.2. rJava安装
1.2.1. rJava介绍
rJava是一个R语言和Java语言的通信接口,通过底层JNI实现调用,允许在R中直接调用Java的对象和方法。
rJava还提供了Java调用R的功能,是通过JRI(Java/R Interface)实现的。JRI现在已经被嵌入到rJava的包中,我们也可以单独试用这个功能。现在rJava包,已经成为很多基于Java开发R包的基础功能组件。
正是由于rJava是底层接口,并使用JNI作为接口调用,所以效率非常高。在JRI的方案中,JVM通过内存直接加载RVM,调用过程性能几乎无损耗,因此是非常高效连接通道,是R和Java通信的首选开发包。
1.2.2. rJava安装
1) 配置rJava环境
执行R CMD javareconf
root@testnode4:/home/payton# R CMD javareconf
2) 启动R并安装rJava
root@testnode4:/home/payton# R
install.packages(“rJava”)
1.3. SparkR的安装
1.3.1. SparkR的代码下载
从网页下载代码SparkR-pkg-master.zip https://github.com/amplab-extras/SparkR-pkg
1.3.2. SparkR的代码编译
1) 解压SparkR-pkg-master.zip,然后cd SparkR-pkg-master/
2) 编译的时候需要指明Hadoop版本和Spark版本
SPARK_HADOOP_VERSION=2.4.1 SPARK_VERSION=1.2.0 ./install-dev.sh
至此,单机版的SparkR已经安装完成。
1.3.3. 分布式SparkR的部署配置
1) 编译成功后,会生成一个lib文件夹,进入lib文件夹,打包SparkR为SparkR.tar.gz,这个是分布式SparkR部署的关键。
2) 由打包好的SparkR.tar.gz在各集群节点上安装SparkR
R CMD INSTALL SparkR.tar.gz
至此分布式SparkR搭建完成。
- SparkR的运行
2.1. SparkR的运行机制
SparkR是AMPLab发布的一个R开发包,为Apache Spark提供了轻量的前端。SparkR提供了Spark中弹性分布式数据集(RDD)的API,用户可以在集群上通过R shell交互性的运行job。SparkR集合了Spark 和R的优势,下面的这3幅图很好的阐释了SparkR的运行机制。

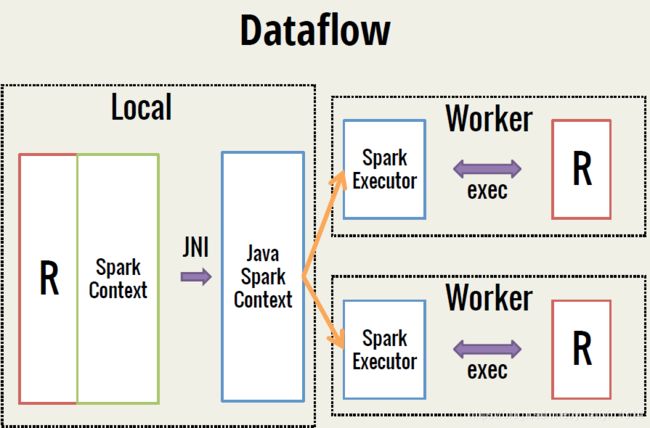
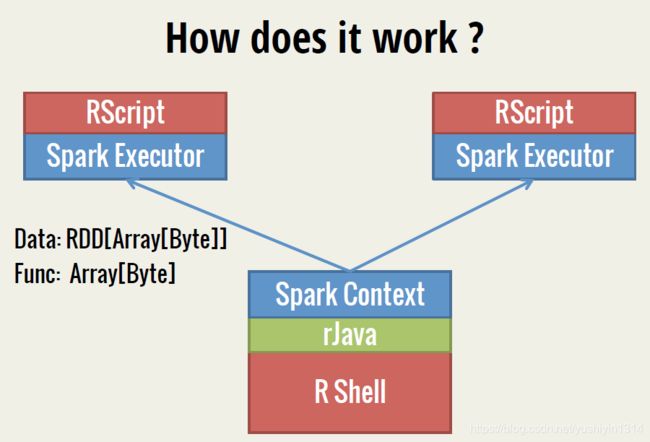
2.2. 用SparkR 进行数据分析
2.2.1. SparkR基本操作
首先介绍下SparkR的基本操作:
第一步,加载SparkR包
library(SparkR)
第二步,初始化Spark context
sc <- sparkR.init(master=" spark://localhost:7077"
,sparkEnvir=list(spark.executor.memory=“1g”,spark.cores.max=“10”))
第三步,读入数据,spark的核心是Resilient Distributed Dataset (RDD),RDDS可以从Hadoop的InputFormats来创建(例如,HDFS文件)或通过转化其它RDDS。例如直接从HDFS读取数据为RDD的示例如下:
lines <- textFile(sc, “hdfs://sparkR_test.txt”)
另外,也可以通过parallelize函数从向量或列表创建RDD,如:
rdd <- parallelize(sc, 1:10, 2)
到了这里,那么我们就可以运用RDD的动作(actions)和转换(transformations)来对RDD进行操作并产生新的RDD;也可以很容易地调用R开发包,只需要在集群上执行操作前用includePackage读取R开发包就可以了(例:includePackage(sc, Matrix));当然还可以把RDD转换为R语言格式的数据形式来对它进行操作。
具体可参见如下两个链接:
http://amplab-extras.github.io/SparkR-pkg/
https://github.com/amplab-extras/SparkR-pkg/wiki/SparkR-Quick-Start
那么下面我们就通过两个示例来看下 SparkR是如何运行的吧。
2.2.2. SparkR使用举例
1) Example1:word count
加载SparkR包
library(SparkR)
初始化 Spark context
sc <- sparkR.init(master=“spark://集群ip:7077”
,sparkEnvir=list(spark.executor.memory=“1g”,spark.cores.max=“10”))
从HDFS上读取文件
lines <- textFile(sc, “hdfs://集群ip:8020/tmp/sparkR_test.txt”)
按分隔符拆分每一行为多个元素,这里返回一个序列
words<-flatMap(lines,function(line) {strsplit(line,"\|")[[1]]})
使用 lapply 来定义对应每一个RDD元素的运算,这里返回一个(K,V)对
wordCount <-lapply(words, function(word) { list(word, 1L) })
对(K,V)对进行聚合计算
counts<-reduceByKey(wordCount,"+",2L)
以数组的形式,返回数据集的所有元素
output <- collect(counts)
按格式输出结果
for (wordcount in output) {
cat(wordcount[[1]], ": ", wordcount[[2]], “\n”)
}
2) Example2:logistic regression
加载SparkR包
library(SparkR)
初始化 Spark context
sc <- sparkR.init(master=“集群ip:7077”,
appName=‘sparkr_logistic_regression’,
sparkEnvir=list(spark.executor.memory=‘1g’,
spark.cores.max=“10”))
从hdfs上读取txt文件, 该RDD由spark集群的4个分区构成
input_rdd <- textFile(sc,
“hdfs://集群ip:8020/user/payton/german.data-numeric.txt”,
minSplits=4)
解析每个RDD元素的文本(在每个分区上并行)
dataset_rdd <- lapplyPartition(input_rdd, function(part) {
part <- lapply(part, function(x) unlist(strsplit(x, ‘\s’)))
part <- lapply(part, function(x) as.numeric(x[x != ‘’]))
part
})
我们需要把数据集dataset_rdd分割为训练集(train)和测试集(test)两部分,这里
ptest为测试集的样本比例,如取ptest=0.2,即取dataset_rdd的20%样本数作为测试集,80%的样本数作为训练集
split_dataset <- function(rdd, ptest) {
#以输入样本数ptest比例创建测试集RDD
data_test_rdd <- lapplyPartition(rdd, function(part) {
part_test <- part[1:(length(part)*ptest)]
part_test
})
用剩下的样本数创建训练集RDD
data_train_rdd <- lapplyPartition(rdd, function(part) {
part_train <- part[((length(part)*ptest)+1):length(part)]
part_train
})
返回测试集RDD和训练集RDD的列表
list(data_test_rdd, data_train_rdd)
}
接下来我们需要转化数据集为R语言的矩阵形式,并增加一列数字为1的截距项,
将输出项y标准化为0/1的形式
get_matrix_rdd <- function(rdd) {
matrix_rdd <- lapplyPartition(rdd, function(part) {
m <- matrix(data=unlist(part, F, F), ncol=25, byrow=T)
m <- cbind(1, m)
m[,ncol(m)] <- m[,ncol(m)]-1
m
})
matrix_rdd
}
由于该训练集中y的值为1与0的样本数比值为7:3,所以我们需要平衡1和0的样本数,使它们的样本数一致
balance_matrix_rdd <- function(matrix_rdd) {
balanced_matrix_rdd <- lapplyPartition(matrix_rdd, function(part) {
y <- part[,26]
index <- sample(which(y0),length(which(y1)))
index <- c(index, which(y==1))
part <- part[index,]
part
})
balanced_matrix_rdd
}
分割数据集为训练集和测试集
dataset <- split_dataset(dataset_rdd, 0.2)
创建测试集RDD
matrix_test_rdd <- get_matrix_rdd(dataset[[1]])
创建训练集RDD
matrix_train_rdd <- balance_matrix_rdd(get_matrix_rdd(dataset[[2]]))
将训练集RDD和测试集RDD放入spark分布式集群内存中
cache(matrix_test_rdd)
cache(matrix_train_rdd)
初始化向量theta
theta<- runif(n=25, min = -1, max = 1)
logistic函数
hypot <- function(z) {
1/(1+exp(-z))
}
损失函数的梯度计算
gCost <- function(t,X,y) {
1/nrow(X)(t(X)%%(hypot(X%*%t)-y))
定义训练函数
train <- function(theta, rdd) {
计算梯度
gradient_rdd <- lapplyPartition(rdd, function(part) {
X <- part[,1:25]
y <- part[,26]
p_gradient <- gCost(theta,X,y)
list(list(1, p_gradient))
})
agg_gradient_rdd <- reduceByKey(gradient_rdd, ‘+’, 1L)
一次迭代聚合输出
collect(agg_gradient_rdd)[[1]][[2]]
}
由梯度下降算法优化损失函数
alpha :学习速率
steps :迭代次数
tol :收敛精度
alpha <- 0.1
tol <- 1e-4
step <- 1
while(T) {
cat(“step: “,step,”\n”)
p_gradient <- train(theta, matrix_train_rdd)
theta <- theta-alpha*p_gradient
gradient <- train(theta, matrix_train_rdd)
if(abs(norm(gradient,type=“F”)-norm(p_gradient,type=“F”))<=tol) break
step <- step+1
}
用训练好的模型预测测试集信贷评测结果(“good”或“bad”),并计算预测正确率
test <- lapplyPartition(matrix_test_rdd, function(part) {
X <- part[,1:25]
y <- part[,26]
y_pred <- hypot(X%*%theta)
result <- xor(as.vector(round(y_pred)),as.vector(y))
})
result<-unlist(collect(test))
corrects = length(result[resultF])
wrongs = length(result[resultT])
cat("\ncorrects: “,corrects,”\n")
cat(“wrongs: “,wrongs,”\n”)
cat(“accuracy: “,corrects/length(y_pred),”\n”)