R in action读书笔记(11)-第八章:回归-- 选择“最佳”的回归模型
8.6 选择“最佳”的回归模型
8.6.1 模型比较
用基础安装中的anova()函数可以比较两个嵌套模型的拟合优度。所谓嵌套模型,即它的一
些项完全包含在另一个模型中
用anova()函数比较
> states<-as.data.frame(state.x77[,c("Murder","Population","Illiteracy","Income","Frost")])
> fit1<-lm(Murder~Population+Illiteracy+Income+Frost,data=states)
>fit2<-lm(Murder~Population+Illiteracy,data=states)
> anova(fit2,fit1)
Analysis of Variance Table
Model 1: Murder ~ Population + Illiteracy
Model 2: Murder ~ Population + Illiteracy + Income +Frost
Res.Df RSS Df Sum of Sq F Pr(>F)
1 47289.25
2 45289.17 2 0.078505 0.0061 0.9939
AIC(AkaikeInformation Criterion,赤池信息准则)也可以用来比较模型,它考虑了模型的
统计拟合度以及用来拟合的参数数目。AIC值越小的模型要优先选择,它说明模型用较少的参数
获得了足够的拟合度。
> AIC(fit1,fit2)
df AIC
fit1 6 241.6429
fit2 4 237.6565
8.6.2变量选择
1. 逐步回归stepwise method
逐步回归中,模型会一次添加或者删除一个变量,直到达到某个判停准则为止。向前
逐步回归(forward stepwise)每次添加一个预测变量到模型中,直到添加变量不会使模型有所改
进为止。向后逐步回归(backward stepwise)从模型包含所有预测变量开始,一次删除一个变量
直到会降低模型质量为止。而向前向后逐步回归(stepwise stepwise,通常称作逐步回归
),结合了向前逐步回归和向后逐步回归的方法,变量每次进入一个,但是每一步
中,变量都会被重新评价,对模型没有贡献的变量将会被删除,预测变量可能会被添加、删除好
几次,直到获得最优模型为止。。MASS包中的stepAIC()函数可以实现
逐步回归模型(向前、向后和向前向后),依据的是精确AIC准则。
> library(MASS)
>fit1<-lm(Murder~Population+Illiteracy+Income+Frost,data=states)
>stepAIC(fit1,direction="backward")
Start: AIC=97.75
Murder ~ Population +Illiteracy + Income + Frost
Df Sum of Sq RSS AIC
- Frost 1 0.021 289.19 95.753
- Income 1 0.057 289.22 95.759
<none> 289.17 97.749
- Population 1 39.238 328.41 102.111
- Illiteracy 1 144.264 433.43 115.986
Step: AIC=95.75
Murder ~ Population +Illiteracy + Income
Df Sum of Sq RSS AIC
- Income 1 0.057 289.25 93.763
<none> 289.19 95.753
- Population 1 43.658332.85 100.783
- Illiteracy 1 236.196 525.38 123.605
Step: AIC=93.76
Murder ~ Population +Illiteracy
Df Sum of Sq RSS AIC
<none> 289.25 93.763
- Population 1 48.517 337.76 99.516
- Illiteracy 1 299.646588.89 127.311
Call:
lm(formula = Murder ~Population + Illiteracy, data = states)
Coefficients:
(Intercept) Population Illiteracy
1.6515497 0.0002242 4.0807366
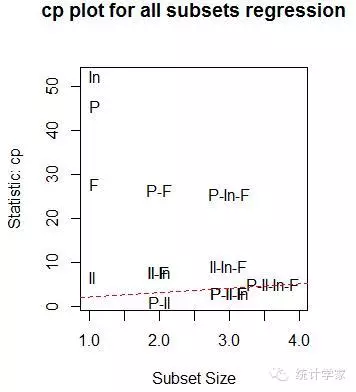
2. 全子集回归
全子集回归可用leaps包中的regsubsets()函数实现。你能通过R平方、调整R平方或
Mallows Cp统计量等准则来选择“最佳”模型
> library("leaps", lib.loc="d:/ProgramFiles/R/R-3.1.3/library")
>leaps<-regsubsets(Murder~Population+Illiteracy+Income+Frost,data=states,nbest=4)
> plot(leaps,scal="adjr2")
> library(car)
> subsets(leaps,statistic="cp",main="cpplot for all subsets regression")
> abline(1,1,lty=2,col="red")

8.7 深层次分析
8.7.1 交叉验证
所谓交叉验证,即将一定比例的数据挑选出来作为训练样本,另外的样本作保留样本,先在
训练样本上获取回归方程,然后在保留样本上做预测。由于保留样本不涉及模型参数的选择,该
样本可获得比新数据更为精确的估计。在k 重交叉验证中,样本被分为k个子样本,轮流将k1个子样本组合作为训练集,另外1个子样本作为保留集。这样会获得k 个预测方程,记录k 个保留样本的预测表现结果,然后求其平均值。[当n 是观测总数目,k 为n 时,该方法又称作刀切法(jackknifing)]bootstrap 包中的crossval() 函数可以实现k 重交叉验证。
fit<-lm(mpg~hp+wt+hp:wt,data=mtcars)
shrinkage<-function(fit,k=10){
require(bootstrap)
theta.fit<-function(x,y){lsfit(x,y)}
theta.predict<-function(fit,x){cbind(1,x)%*%fit$coef}
x<-fit$model[,2:ncol(fit$model)]
y<-fit$model[,1]
results<-crossval(x,y,theta.fit,theta.predict,ngroup=k)
r2<-cor(y,fit$fitted.values)^2
r2cv<-cor(y,results$cv.fit)^2
cat("original r-square=",r2,"\n")
cat(k,"fold cross-validated r-square =",r2cv,"\n")
cat("change=",r2-r2cv),"\n")
}
