【Task07】Pandas之缺失数据
前言
本章介绍pandas中的缺失数据,主要内容有:
- pandas中对np.nan的操作: 统计 、 删除 、 填充 、 插值
- pandas中的Nullable类型及相关操作
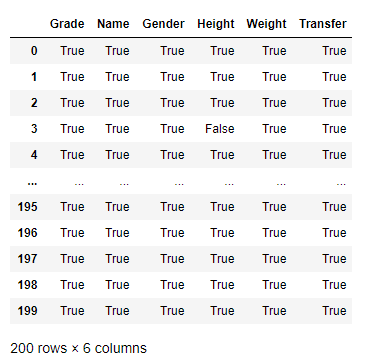
在无特殊说明时,本章主要采用的df数据如下,不再重复说明:
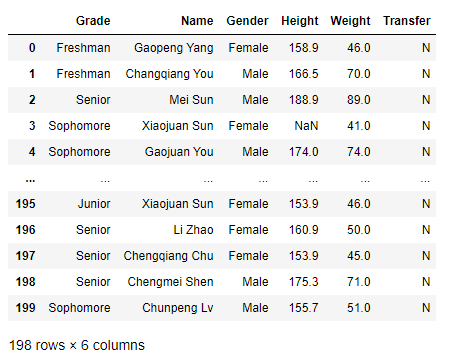
df = pd.read_csv('./data/learn_pandas.csv',usecols=['Grade','Name','Gender','Height','Weight','Transfer'])
df
一、缺失值的统计和删除
1.缺失值的统计
我们可以使用isna()和isnull()方法来统计数据中的np.nan数据:
df.isna()
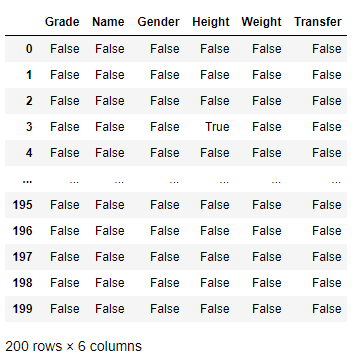
返回的是相同形状的数据,对于非np.nan的元素返回 Fasle ,否则返回 True 。
接下来让我们验证这两种方法的等效性:
>>> df.isna().equals(df.isnull())
True
notna()和notnull()方法与isna()方法正好相反,它对非缺失值返回的是True,缺失值返回Fasle:
同样地,我们来验证一下它们之间的关系:
>>> df.notna().equals(df.notnull())
True
>>> df.notna().equals(~df.isna())
True
证明这四个方法确实是两两相同,两两相反。
1)配合其他统计方法使用
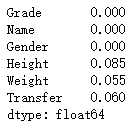
我们可以将isna()方法与一些其他统计方法一起使用,如统计每行数据缺失值的数量:
df.isna().sum(axis = 1)

axis = 1代表沿着每列去统计,结果返回的是每行的缺失值数
也可以统计每列缺失值所占的比例:
df.isna().sum(axis = 0)/df.shape[0]
其中,axis = 0代表代表沿着每行去统计,结果返回的是每列的缺失值数,df.shape[0]代表数据列的长度。
2)配合索引使用
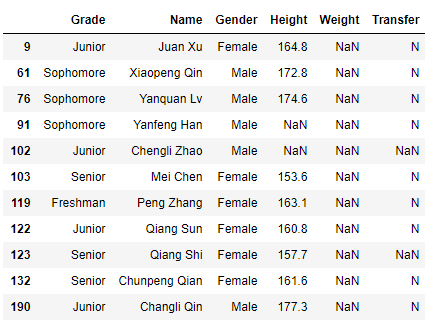
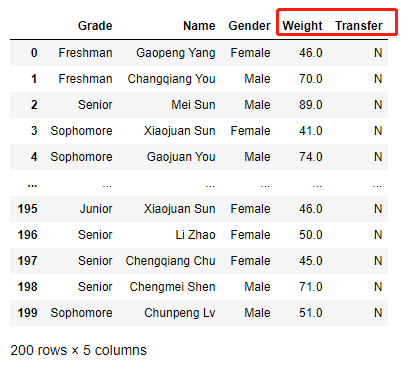
也可以将isna()方法与索引一起使用,如返回体重为缺失值的行:
df[df['Weight'].isna()]
3)配合逻辑方法使用
如果要返回身高体重同时缺失的行,就需要逻辑方法配合:
df[df[['Height','Weight']].isna().all(axis = 1)]
也可以统计df中有缺失值的列:
df.isna().any(axis = 0)

返回False代表该列无缺失值,True代表该列至少有一个缺失值。
总结规律如下:
| 缺失值方法 | 逻辑方法 | 结果 | 含义 |
|---|---|---|---|
| isna() or isnull() | all | True | 都是缺失值 情况4 |
| isna() or isnull() | all | False | 不都是缺失值 情况3 |
| isna() or isnull() | any | True | 至少有一个缺失值 情况2 |
| isna() or isnull() | any | False | 都不是缺失值 情况1 |
| notna() or notnull() | all | True | 都是非缺失值 情况1 |
| notna() or notnull() | all | False | 不都是非缺失值 情况2 |
| notna() or notnull() | any | True | 至少有一个非缺失值 情况3 |
| notna() or notnull() | any | False | 都不是非缺失值 情况4 |
进一步总结,上面的含义可以划分为4种情况,即isna()和notna()在逻辑方法不同,结果不同时,代表含义相同。
2.缺失值的删除
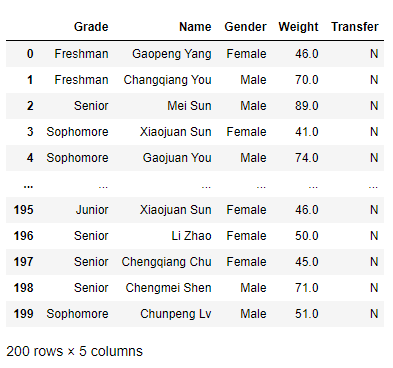
在pandas中利用dropna方法对缺失值进行删除:
res = df.dropna(how = 'all',subset=['Height','Weight'])
res
how参数可以设置成‘all’或‘any’,默认是‘any’,subset参数代表删除考虑的列名,作用和利用df索引进行访问等同。
来跟上面例子联动一下,查看一下行索引为91和102的同学:
>>> res.loc[91]
KeyError: 91
>>> res.loc[102]
KeyError: 102
可以看到确实是成功删除了。
1)thresh参数
thresh参数代表数据不被删除至少需要的非缺失值数量:
df.dropna(axis = 1,thresh = df.shape[0] - 15)
这里的axis参数跟上面sum方法中的axis参数相比,有一些不一样。我们知道sum方法中axis参数为1代表 沿着列 进行求和,最终返回的是 每行 的和,而dropna中axis参数为1仅代表对 每列 的非缺失值进行thresh值比较,意义是不同的,这也警告我们不要对所有方法中的axis参数统一去看待,要视情况而定。
2)自定义方法代替dropna方法
其实dropna的返回结果可以理解成按条件筛选,所以我们可以利用缺失值统计的相关方法进行自定义方法来代替方法:
#删除身高体重均为缺失值的行(保留身高体重至少有一个为非缺失值的行)
>>> res2 = df[df[['Height','Weight']].notna().any(axis = 1)]
>>> res2.equals(res)
True
>>> res = df.dropna(how = 'any',subset=['Height','Weight'])
>>> res2 = df[df[['Height','Weight']].notna().all(axis = 1)]
>>> res2.equals(res)
True
对于无thresh参数的简单dropna使用,总结如下:
| dropna的how参数 | 对应的缺失值统计方法和逻辑方法 |
|---|---|
| all | notna + any |
| any | notna + all |
这是由于,dropna()代表删除isna()在all判断下为True的数据,等价于保留notna()在any判断下为True的数据。
在来看如何代替带有thresh参数的dropna方法:
df.loc[:,df.notna().sum(axis = 0) >= df.shape[0]-15]
即利用notna方法和loc索引,返回非缺失值大于等于thresh的列。
二、缺失值的填充和插值
1.填充
我们利用fillna方法对缺失值进行填充,以Series举例,比较重要的参数有:
- value:标量或字典(索引到元素的映射)
- method:ffill代表由前面的非缺失值来填充,bfill代表由后面的来填充,默认是None
- limit:代表连续缺失值最多填充次数
- inplace:是否替换原数据
在这里使用的Series数据如下:
>>> s = pd.Series([np.nan,2,np.nan,np.nan,0,np.nan],list('Xiaomy'))
>>> s
X NaN
i 2.0
a NaN
o NaN
m 0.0
y NaN
dtype: float64
1)利用value参数进行填充
#使用标量进行填充
>>> s.fillna(1)
X 1.0
i 2.0
a 1.0
o 1.0
m 0.0
y 1.0
dtype: float64
#使用字典进行填充
>>> s.fillna({
'X':1,'y':100,'c':1})
X 1.0
i 2.0
a NaN
o NaN
m 0.0
y 100.0
dtype: float64
我们可以看到在使用字典进行填充时字典不需要包含所有缺失值的索引,且字典里面可以包含其他非索引值。
2)利用method方法进行填充
也可以利用method方法进行连续填充:
#由前面的非缺失值向后填充
>>> s.fillna(method='ffill')
X NaN
i 2.0
a 2.0
o 2.0
m 0.0
y 0.0
dtype: float64
#由后面的非缺失值向前填充
>>> s.fillna(method='bfill')
X 2.0
i 2.0
a 0.0
o 0.0
m 0.0
y NaN
dtype: float64
这里可以看到,对于ffill方式,开始的缺失值是不能填充的;对于bfill方式,结尾的缺失值也是不能填充的。
3)分组和缺失值填充的配合
对于df数据中身高的缺失项,最好利用对应年级的相同性别的平均身高来填充:
>>> df.groupby(['Grade','Gender'])['Height'].transform(lambda x:x.fillna(x.mean()))
0 158.900000
1 166.500000
2 188.900000
3 158.363158
4 174.000000
...
195 153.900000
196 160.900000
197 153.900000
198 175.300000
199 155.700000
Name: Height, Length: 200, dtype: float64
这里利用的是分组中的自定义变换方法配合缺失值填充使用。
另外,limit参数可以限制填充的次数,以配合method参数为例:
>>> s.fillna(method='ffill',limit=1)
X NaN
i 2.0
a 2.0
o NaN
m 0.0
y 0.0
dtype: float64
当然,limit参数也可以配合其他参数进行使用,这里不再赘述。
练一练
题目:对一个序列以如下规则填充缺失值:如果单独出现的缺失值,就用前后均值填充,如果连续出现的缺失值就不填充,即序列[1, NaN, 3, NaN, NaN]填充后为[1, 2, 3, NaN, NaN],请利用fillna函数实现。(提示:利用`limit``参数)
>>> s = pd.Series([1,np.nan,3,np.nan,np.nan])
>>> res = (s.fillna(method='ffill',limit=1)+s.fillna(method='bfill',limit=1))/2
>>> res
0 1.0
1 2.0
2 3.0
3 NaN
4 NaN
dtype: float64
思路:对于不满足题设条件的位置包括处于两端的位置,利用np.nan和其他值相加为本身的特性,依然保持为np.nan;对于左右均为非缺失值的位置,即可求得左右两侧之和,然后最后除2即可。
2.插值方法
在pandas中一般使用interpolate方法进行插值,重要的参数有:
- limit_direction:‘forward’代表由前面的非缺失值进行插值,‘backward’代表由前面的非缺失值进行插值,‘both’代表二者兼有
- method:插值方法,包括‘nearest’, ‘zero’, ‘slinear’, ‘quadratic’, ‘cubic’, ‘spline’, ‘barycentric’, ‘polynomial’,默认为‘linear’
- inplace:是否替换原数据
- limit:插值次数
本节使用的Series数据如下:
>>> s = pd.Series([np.nan, np.nan, 1, np.nan, np.nan, np.nan, 2, np.nan, np.nan])
>>> s
0 NaN
1 NaN
2 1.0
3 NaN
4 NaN
5 NaN
6 2.0
7 NaN
8 NaN
dtype: float64
1)线性插值
>>> s.interpolate(limit_direction='backward')
0 1.00
1 1.00
2 1.00
3 1.25
4 1.50
5 1.75
6 2.00
7 NaN
8 NaN
dtype: float64
>>> s.interpolate(limit_direction='both')
0 1.00
1 1.00
2 1.00
3 1.25
4 1.50
5 1.75
6 2.00
7 2.00
8 2.00
dtype: float64
这里举了向前和向两端分别进行线性插值的例子,这里注意如果缺失值的左侧或右侧完全没有非缺失值,那么它会由最近的非缺失值来填充。
2)最邻近插值
注意要提前install scipy库,否则会报错:
>>> s.interpolate(method = 'nearest')
0 NaN
1 NaN
2 1.0
3 1.0
4 1.0
5 2.0
6 2.0
7 NaN
8 NaN
dtype: float64
注意,这里会忽视两端,且距离两侧非缺失值相同的位置会默认插入前一个非缺失值。
3)索引插值
索引插值可以理解成不等比线性插值,它是根据索引的相对距离进行插入,有点百分位数的意思:
>>> s = pd.Series([0,np.nan,100],index=[0,1,10])
>>> s.interpolate(method = 'index')
0 0.0
1 10.0
10 100.0
dtype: float64
>>> s.interpolate()
0 0.0
1 50.0
10 100.0
dtype: float64
注意它和线性插值的区别。
三、Nullable类型
1.缺失值的本质和缺陷
python中用None表示缺失值,Numpy中用np.nan表示缺失值,它俩的共同点都是与其他值不等,而不同点是后者与自己也不等且没有用于关键字,需要通过numpy.nan来使用:
>>> None == True
False
>>> np.nan == True
False
>>> np.nan == []
False
>>> None == []
False
>>> np.nan == ''
False
>>> None == ''
False
>>> None == None
True
>>> np.nan == np.nan
False
>>> pd.Series([1, np.nan]) == pd.Series([1, np.nan])
True
虽然两个np.nan是不等的,但是对于包含np.nan变量的s或df数据,它们会跳过比较对应位置的np.nan变量。
np.nan的缺陷在于,它的本质是一种float类型的变量,当它和其他类型同时存在于数据中时,会改变整个数据的类型,如:
>>> pd.Series([100,np.nan]).dtype
dtype('float64')
>>> pd.Series(['1',np.nan]).dtype
dtype('O')
>>> pd.Series([False,np.nan]).dtype
dtype('O')
当np.nan和int型变量放在一起时,会使整个数据序列变成float64型;当np.nan和其他类型变量放在一起时,会使整个数据序列变成object型。
2.pandas中的Nullable类型
pandas设计了新的缺失类型pd.NA以及三种Nullable序列类型尝试解决这些缺陷
它们的作用之一是在比较时返回pd.NA本身而不是False:
>>> s = pd.Series(['a', 'b'])
>>> s_bool = pd.Series([True, np.nan])
>>> s_boolean = pd.Series([True, np.nan]).astype('boolean')
>>> s_bool & True
0 True
1 False
dtype: bool
>>> s_boolean & True
0 True
1 <NA>
dtype: boolean
即不会改变比较前后的结果。
3.缺失数据的计算和分组
对np.nan和pd.NA进行标量运算时,除了1的np.nan次幂和np.nan的0次幂两种情况以外,均返回其自身:
>>> np.nan + 1
nan
>>> pd.NA + 1
<NA>
>>> np.nan * 10
nan
>>> pd.NA * 10
<NA>
>>> np.nan ** 0
1.0
>>> 1 ** np.nan
1.0
在对含有np.nan的数据进行操作时,默认会忽略它们,如:
>>> s = pd.Series([7,6,np.nan,5,4])
>>> s.sum()
22.0
注意虽然忽视了np.nan,但由于np.nan的存在,还是让结果变为了float类型。
练习
Ex1:缺失值与类别的相关性检验
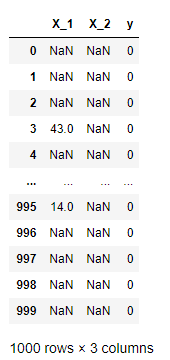
在数据处理中,含有过多缺失值的列往往会被删除,除非缺失情况与标签强相关。下面有一份关于二分类问题的数据集,其中X_1, X_2为特征变量,y为二分类标签:
df = pd.read_csv('./data/missing_chi.csv')
df
from scipy.stats import chi2
df = pd.read_csv('./data/missing_chi.csv')
#分别将两列的缺失值和非缺失值替换为字符‘NaN’和‘NotNaN’
cat_1 = df.X_1.fillna('NaN').mask(df.X_1.notna()).fillna("NotNaN")
cat_2 = df.X_2.fillna('NaN').mask(df.X_2.notna()).fillna("NotNaN")
#分别进行行列汇总 方便计算Eij和Fij
df_1 = pd.crosstab(cat_1, df.y, margins=True)
df_2 = pd.crosstab(cat_2, df.y, margins=True)
def compute_S(my_df):
#双层遍历,利用列表推导生成式去做
res = [((my_df.iat[i, j]-(my_df.iat[i, 2]*my_df.iat[2, j]/my_df.iat[2,2]))**2/(my_df.iat[i, 2]*my_df.iat[2, j]/my_df.iat[2,2])) for i in range(2) for j in range(2)]
#返回总和
return sum(res)
res1 = compute_S(df_1)
res2 = compute_S(df_2)
>>> print(chi2.sf(res1, 1) < 0.05)
False
>>> print(chi2.sf(res2, 1) < 0.05)
True
思路:照着参考答案写的,对每一步进行注释,并对循环进行了一定的优化。
Ex2:用回归模型解决分类问题
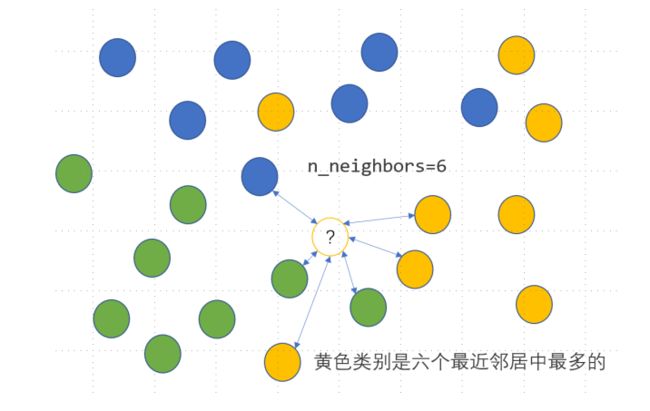
KNN是一种监督式学习模型,既可以解决回归问题,又可以解决分类问题。对于分类变量,利用KNN分类模型可以实现其缺失值的插补,思路是度量缺失样本的特征与所有其他样本特征的距离,当给定了模型参数n_neighbors=n时,计算离该样本距离最近的n个样本点中最多的那个类别,并把这个类别作为该样本的缺失预测类别,具体如下图所示,未知的类别被预测为黄色:
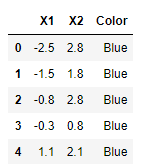
df = pd.read_excel('./data/color.xlsx')
df.head()
from sklearn.neighbors import KNeighborsClassifier
clf = KNeighborsClassifier(n_neighbors=6)
#传入前两列和预测值
clf.fit(df.iloc[:,:2], df.Color)
clf.predict([[0.8, -0.2]])
1.分类转回归
2.缺失值插补
df = pd.read_csv('./data/audit.csv')
df.head()
参考文献
1.pandas中Dataframe的查询方法([], loc, iloc, at, iat, ix)
https://blog.csdn.net/wr339988/article/details/65446138