异常检测TASK1
Task1:异常检测介绍(2天)
摘要:本博客对于Datawhale TASK1涉及到的知识点进行了思维导图的整理,而后对pyod使用绘制了思维导图,最后使用了两个数据集进行练习,一个是使用API产生的数据,一个是UCI上面的癫痫的数据集。本博客主要探究了evaluate_print这个API发现它并不能计算precision(准确率),转而采用sklearn的precision_score API,探究的过程主要采用Spyder编辑器的查看定义的功能。
目录
- 1.笔记
- 2.pyod api使用流程
- 3.练习
-
- 3.1 pyod库的安装
- 3.2 toy example
- 3.3 癫痫数据集
-
- 3.3.1 说明
- 3.3.2 归一化
- 3.3.3 训练结果
- 4.代码附件
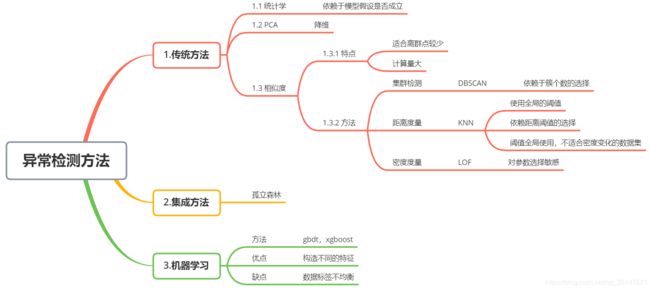
1.笔记
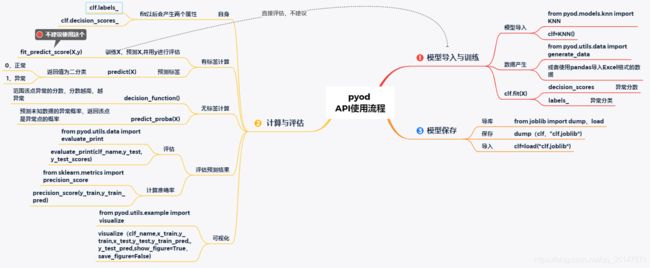
2.pyod api使用流程
3.练习
3.1 pyod库的安装
由于我使用的是anaconda,所以打开anaconda prompt(anaconda)程序,输入:pip install pyod -i http://pypi.douban.com/simple/ 就安装成功了
3.2 toy example
说明:我选择examples里面的LOF的例子做的,(Gitee确实很好用)LOF Gitee 链接
我把这个toy example熟悉的过程中,有了不少的感悟,总结如下:
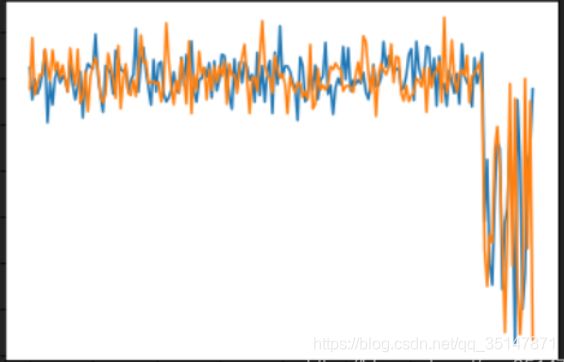
plt.plot(X_train)
导致画出来的图是这样的
plt.scatter(X_train[:,0],X_train[:,1])
3.precision指的是查准率,对应的还有查全率,与常采用的精度和错误率是不同的(虽然之前看过西瓜书,还是还是没能认出来,)(本来当成精度,以为代码有问题,就把代码复制到Spyder里面查看源码,在代码上面右击,点击go to definition 即可,我目前不会jupyter查看源码的方法)
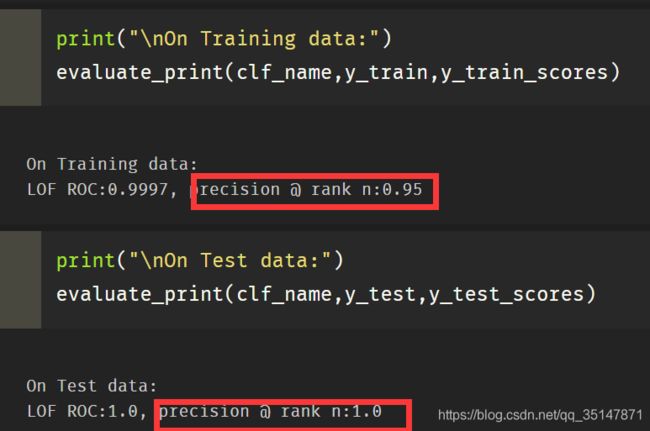
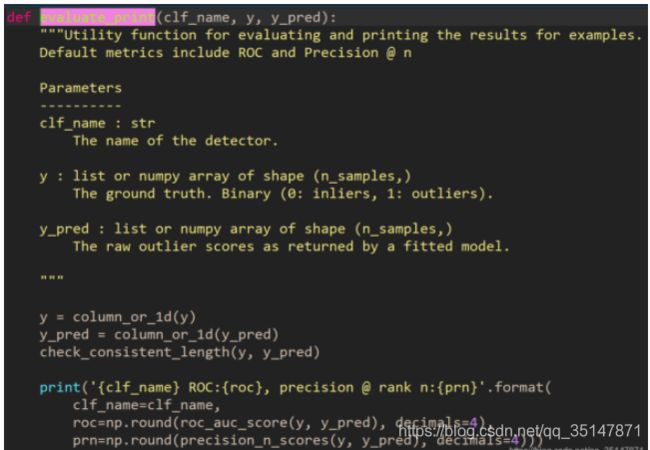
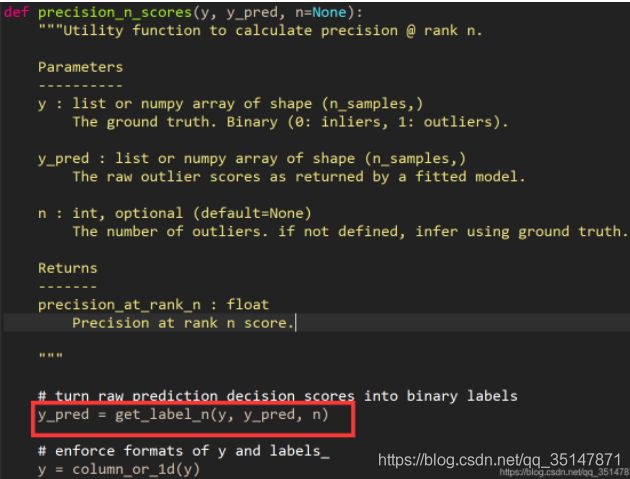
4. 我发现evaluate_print()做出来的查准率和查全率与本次toy example 我手动算的不一样(通过图可以看出来,而且test里面precision是1 就很奇怪),于是我查看了evaluate_print()的源码,发现这个函数,采用的y_pred不是模型预测的,下图2可以看到y_pred被重新赋值了。
y_pred的产生:
- .根据训练集里面的异常值占比,来确定判定异常的阈值;
- 根据阈值,将y_scores分类,分为0和1,并把这个新的标签赋值给y_pred
所以evaluate_print()函数与下面的图3是没有关系的。
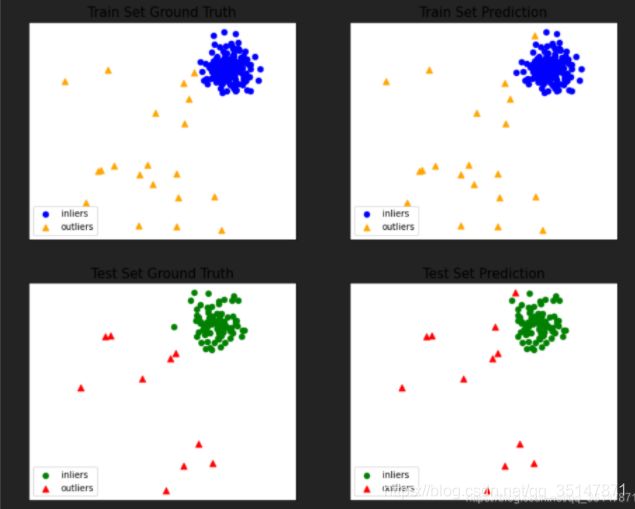
5.如何计算查准率呢?这样计算出来的结果与结果展示的那张图是对应的。
- from sklearn.metrics import precision_score
- precision_score(y_train,y_train_pred)
3.3 癫痫数据集
数据来源:来自UCI公开数据集
3.3.1 说明
Wu Qiuyi Wu
数学学院,
罗彻斯特理工学院,
电子邮件:qw9477’@'rit.edu
欧内斯特·福科厄
数学学院,
罗彻斯特理工学院,
电子邮件:epfeqa’@'rit.edu
电话:585 739 6893
数据集信息:
请在“ [Web链接] ”中 找到原始数据
属性信息:
参考文献的原始数据集包含5个不同的文件夹,每个文件夹包含100个文件,每个文件代表一个主题/人。每个文件记录了23.6秒的大脑活动。相应的时间序列被采样到4097个数据点中。每个数据点是在不同时间点的EEG记录的值。因此,我们共有500个人,每个人有4097个数据点,持续23.5秒。
我们将4097个数据点分为23个数据块,并将其洗牌,每个数据块包含178个数据点,持续1秒钟,每个数据点是在不同时间点的EEG记录值。因此,现在我们有23 x 500 = 11500条信息(行),每个信息包含1秒(列)的178个数据点,最后一列表示标签y {1,2,3,4,5}。
响应变量在列179中为y,说明变量X1,X2,…,X178
y包含178维输入向量的类别。具体来说,在{1,2,3,4,5}中的y:
5-睁开眼睛,表示他们正在记录大脑的EEG信号时,病人睁开眼睛
4-睁开眼睛,意味着他们在记录EEG信号时患者闭上眼睛
3-是的,他们确定了大脑中肿瘤的位置,并记录了健康大脑区域的脑电图活动
2-他们记录了肿瘤所在区域的脑电图
1-记录了癫痫发作活动
属于第2、3、4和5类的所有受试者都是没有癫痫发作的受试者。仅1级受试者患有癫痫发作。我们创建此版本数据的动机是通过创建.csv版本来简化对数据的访问。尽管有5个班级,大多数作者都进行了二分类,即相对于其他班级为1级(癫痫发作)。
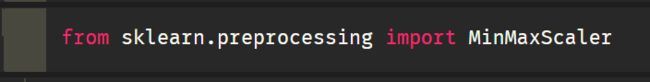
3.3.2 归一化
本次只区分正常和有疾病的(对某些类别进行了合并)
归一化与训练集、测试集的划分
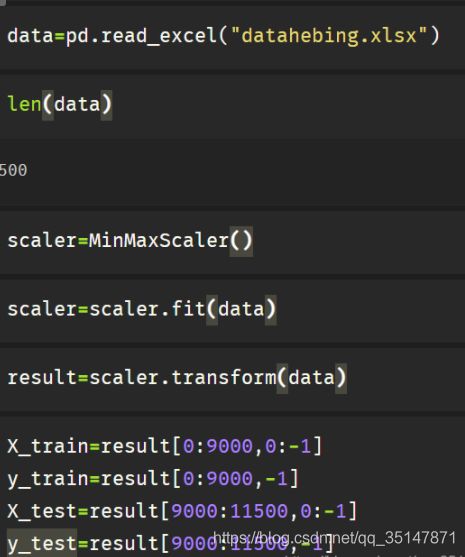
3.3.3 训练结果
训练过程与toy example相同
训练结果准确率与模型评估都较好。
由于是高维数据,无法可视化。
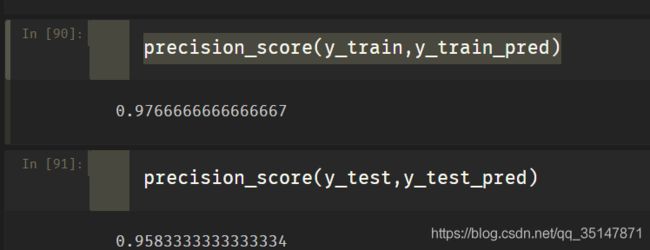
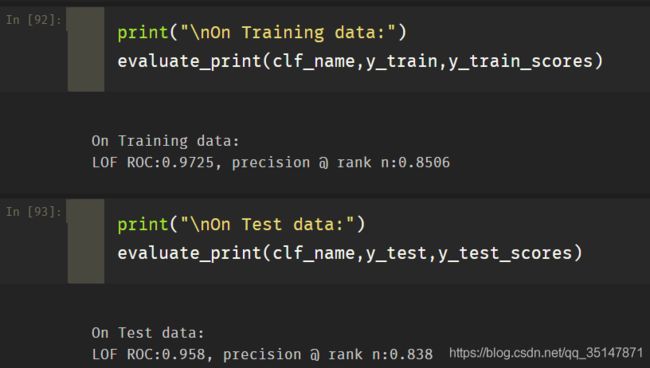
4.代码附件
- 代码的HTML展示
- 代码.ipython格式
- 数据文件hebing.7z(癫痫数据集)
Gitee 链接