pandas-task09-分类数据.md
文章目录
- 一、cat(category)对象
-
- 1. cat对象的属性
- 2. 类别的增加、删除和修改
-
- 1. 增加 add_categories
- 2.删除 remove_categories
- 3.改 rename_categories
- 二、有序分类
-
- 1. 序的建立
- 2. 排序和比较
- 三、区间类别
-
- 1. 利用cut和qcut进行区间构造
- 2. 一般区间的构造
- 3. 区间的属性与方法
- 四、练习
-
- Ex1:统计未出现的类别
- Ex2:钻石数据集
一、cat(category)对象
1. cat对象的属性
pandas中有一个dtype为category 类型,该类型是为了使用户能够处理分类类型的变量。将一个普通序列转换成分类变量可以使用 astype 方法。
df = pd.read_csv('data/learn_pandas.csv',
usecols = ['Grade', 'Name', 'Gender', 'Height', 'Weight'])
df.head()
| Grade | Name | Gender | Height | Weight | |
|---|---|---|---|---|---|
| 0 | Freshman | Gaopeng Yang | Female | 158.9 | 46 |
| 1 | Freshman | Changqiang You | Male | 166.5 | 70 |
| 2 | Senior | Mei Sun | Male | 188.9 | 89 |
| 3 | Sophomore | Xiaojuan Sun | Female | nan | 41 |
| 4 | Sophomore | Gaojuan You | Male | 174 | 74 |
s = df.Grade.astype('category')
s.head()
# 0 Freshman
# 1 Freshman
# 2 Senior
# 3 Sophomore
# 4 Sophomore
# Name: Grade, dtype: category
# Categories (4, object): ['Freshman', 'Junior', 'Senior', 'Sophomore']
在一个分类类型的 Series 中定义了 cat 对象,它和上一章中介绍的 str 对象类似,定义了一些属性和方法来进行分类类别的操作。
s.cat
对于一个具体的分类,有两个组成部分,其一为类别的本身,它以 Index 类型存储,其二为是否有序,它们都可以通过 cat 的属性被访问:
s.cat.categories
# Index(['Freshman', 'Junior', 'Senior', 'Sophomore'], dtype='object')
s.cat.ordered# 是否有序
# False
另外,每一个序列的类别会被赋予唯一的整数编号,它们的编号取决于 cat.categories 中的顺序,该属性可以通过 codes 访问。
讲明白点,比如上面例子年级这列有四种值,大一到大四,分别对应编号0123,那么年级这条Series的每个值其实都对应着自己的编号。0号是大一,对应0。2号是大三,对应2.
s.cat.codes.head()
# 0 0
# 1 0
# 2 2
# 3 3
# 4 3
# dtype: int8
2. 类别的增加、删除和修改
通过 cat 对象的 categories 属性能够完成对类别的查询,那么应该如何进行“增改查删”的其他三个操作呢?
类别不得直接修改。
在第三章中曾提到,索引 Index 类型是无法用 index_obj[0] = item 来修改的,而 categories 被存储在 Index 中,因此 pandas 在 cat 属性上定义了若干方法来达到相同的目的。
1. 增加 add_categories
s = s.cat.add_categories('Graduate') # 增加一个毕业生类别
s.cat.categories
# Index(['Freshman', 'Junior', 'Senior', 'Sophomore', 'Graduate'], dtype='object')
2.删除 remove_categories
所有原来序列中的该类会被设置为缺失。例如,删除大一的类别:
s.cat.remove_categories('Freshman').head()
# 0 NaN
# 1 NaN
# 2 Senior
# 3 Sophomore
# 4 Sophomore
# Name: Grade, dtype: category
# Categories (4, object): ['Junior', 'Senior', 'Sophomore', 'Graduate']
此外可以使用 set_categories 直接设置序列的新类别,原来的类别中如果存在元素不属于新类别,那么会被设置为缺失。
s.cat.set_categories(['Freshman','PHD'])
# 0 Freshman
# 1 Freshman
# 2 NaN
# 3 NaN
# 4 NaN
# Name: Grade, dtype: category
# Categories (2, object): ['Freshman', 'PHD']
直接set_categories([ ‘Junior’, ‘Senior’, ‘Sophomore’, ‘Graduate’])也可以达到remove_categories(‘Freshman’)的效果。
如果想要删除未出现在序列中的类别,可以使用 remove_unused_categories 来实现:
s = s.cat.remove_unused_categories() # 移除了未出现的博士生类别
s.cat.categories
# Index(['Freshman', 'Junior', 'Senior', 'Sophomore'], dtype='object')
3.改 rename_categories
该方法会对原序列的对应值也进行相应修改。例如,现在把 Sophomore 改成中文的 本科二年级学生 :
s = s.cat.rename_categories({
'Sophomore':'本科二年级学生'})
s.head()
# 0 Freshman
# 1 Freshman
# 2 Senior
# 3 本科二年级学生
# 4 本科二年级学生
# Name: Grade, dtype: category
# Categories (4, object): ['Freshman', 'Junior', 'Senior', '本科二年级学生']
二、有序分类
1. 序的建立
有序类别和无序类别可以通过 as_unordered 和 as_ordered 互相转化,如果想指定顺序,可以使用reorder_categories,传入的参数必须是由当前序列的无需类别构成的列表,不能够增加新的类别,也不能缺少原来的类别,并且必须指定参数 ordered=True ,否则方法无效。
例如,对年级高低进行相对大小的类别划分,然后再恢复无序状态:
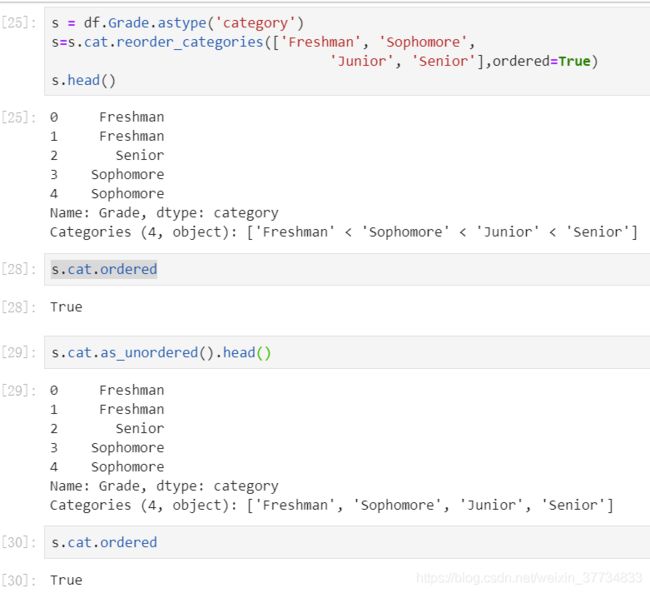
2. 排序和比较
在第二章中,曾提到了字符串和数值类型序列的排序,此时就要说明分类变量的排序:只需把列的类型修改为 category 后,再赋予相应的大小关系,就能正常地使用 sort_index 和 sort_values 。例如,对年级进行排序:
df.Grade = df.Grade.astype('category')
df.Grade = df.Grade.cat.reorder_categories(['Freshman',
'Sophomore',
'Junior',
'Senior'],ordered=True)
df.sort_values('Grade').head() # 值排序
| Grade | Name | Gender | Height | Weight | |
|---|---|---|---|---|---|
| 0 | Freshman | Gaopeng Yang | Female | 158.9 | 46 |
| 105 | Freshman | Qiang Shi | Female | 164.5 | 52 |
| 96 | Freshman | Changmei Feng | Female | 163.8 | 56 |
| 88 | Freshman | Xiaopeng Han | Female | 164.1 | 53 |
| 81 | Freshman | Yanli Zhang | Female | 165.1 | 52 |
df.set_index('Grade').sort_index().head()# 索引排序
| Grade | Name | Gender | Height | Weight |
|---|---|---|---|---|
| Freshman | Gaopeng Yang | Female | 158.9 | 46 |
| Freshman | Qiang Shi | Female | 164.5 | 52 |
| Freshman | Changmei Feng | Female | 163.8 | 56 |
| Freshman | Xiaopeng Han | Female | 164.1 | 53 |
| Freshman | Yanli Zhang | Female | 165.1 | 52 |
由于序的建立,因此就可以进行比较操作。分类变量的比较操作分为两类,第一种是 == 或 != 关系的比较,比较的对象可以是标量或者同长度的 Series (或 list ),第二种是 >,>=,<,<= 四类大小关系的比较,比较的对象和第一种类似,但是所有参与比较的元素必须属于原序列的 categories ,同时要和原序列具有相同的索引。
res1 = df.Grade == 'Sophomore'
res2 = df.Grade == ['Sophomore']*df.shape[0]
# res1和res2效果一致
# 0 False
# 1 False
# 2 False
# 3 True
# 4 True
# Name: Grade, Length: 200, dtype: bool
res3 = df.Grade <= 'Sophomore'
res3
# 0 True
# 1 True
# 2 False
# 3 True
# 4 True
res4 = df.Grade <= df.Grade.sample(
frac=1).reset_index(
drop=True) # 打乱后比较
res4.head()
# 0 True
# 1 True
# 2 False
# 3 True
# 4 True
# Name: Grade, dtype: bool
三、区间类别
1. 利用cut和qcut进行区间构造
区间是一种特殊的类别,在实际数据分析中,区间序列往往是通过 cut 和 qcut 方法进行构造的,这两个函数能够把原序列的数值特征进行装箱,即用区间位置来代替原来的具体数值。
首先介绍 cut 的常见用法:
其中,最重要的参数是 bin ,如果传入整数 n ,则代表把整个传入数组的按照最大和最小值等间距地分为 n 段。由于区间默认是左开右闭,需要进行调整把最小值包含进去,在 pandas 中的解决方案是在值最小的区间左端点再减去 0.001*(max-min) ,因此如果对序列 [1,2] 划分为2个箱子时,第一个箱子的范围 (0.999,1.5] ,第二个箱子的范围是 (1.5,2] 。如果需要指定左闭右开时,需要把 right 参数设置为 False ,相应的区间调整方法是在值最大的区间右端点再加上 0.001*(max-min) 。
s = pd.Series([1,2])
pd.cut(s,bins=2)# 分割成两份 区间默认左开右闭
# 0 (0.999, 1.5]
# 1 (1.5, 2.0]
# dtype: category
# Categories (2, interval[float64]): [(0.999, 1.5] < (1.5, 2.0]]
pd.cut(s,bins=2,right=False)#左闭右开
# 0 [1.0, 1.5)
# 1 [1.5, 2.001)
# dtype: category
# Categories (2, interval[float64]): [[1.0, 1.5) < [1.5, 2.001)]
这里用身高数据进行进一步实践,深入了解cut的过程。
首先获得min和max值分别作为左右端点,即(34,89],但默认左开右闭的形式导致min不被包含在其中,所以左端点34要减去0.001*(max-min)即左端点为34-0.055=33.945,使得34也在区间种。区间长度为(mx-min)/n=(89-34)/3=18.333,但是由于最左边区间减了0.55,为了简便,最左边的区间长度再加上0.55。
s = df.Weight
pd.cut(s, bins=3,right=True)
# 0 (33.945, 52.333]
# 1 (52.333, 70.667]
# 2 (70.667, 89.0]
# 3 (33.945, 52.333]
# 4 (70.667, 89.0]
# ...
# 195 (33.945, 52.333]
# 196 (33.945, 52.333]
# 197 (33.945, 52.333]
# 198 (70.667, 89.0]
# 199 (33.945, 52.333]
# Name: Weight, Length: 200, dtype: category
# Categories (3, interval[float64]): [(33.945, 52.333] < (52.333, 70.667] < (70.667, 89.0]]
bins 的另一个常见用法是指定区间分割点的列表(使用 np.infty 可以表示无穷大):
pd.cut(s, bins=[-np.infty, 1.2, 1.8, 2.2, np.infty])
# 0 (-inf, 1.2]
# 1 (1.8, 2.2]
# dtype: category
# Categories (4, interval[float64]): [(-inf, 1.2] < (1.2, 1.8] < (1.8, 2.2] < (2.2, inf]]
另外两个常用参数为 labels 和 retbins ,分别代表了区间的名字和是否返回分割点(默认不返回):
s = pd.Series([1,2])
res = pd.cut(s, bins=2, labels=['small', 'big'], retbins=True)
res[0]
# 0 small
# 1 big
# dtype: category
# Categories (2, object): ['small' < 'big']
res[1] # 该元素为返回的分割点
# array([0.999, 1.5 , 2. ])
从用法上来说, qcut 和 cut 几乎没有差别,只是把 bins 参数变成的 q 参数, qcut 中的 q 是指 quantile 。这里的 q 为整数 n 时,指按照 n 等分位数把数据分箱,还可以传入浮点列表指代相应的分位数分割点。
2. 一般区间的构造
对于某一个具体的区间而言,其具备三个要素,即左端点、右端点和端点的开闭状态,其中开闭状态可以指定 right, left, both, neither 中的一类:
my_interval = pd.Interval(0, 1, 'right')
my_interval
# Interval(0, 1, closed='right')
其属性包含了 mid, length, right, left, closed ,分别表示中点、长度、右端点、左端点和开闭状态。
使用 in 可以判断元素是否属于区间:
0 in my_interval
#False
使用 overlaps 可以判断两个区间是否有交集:
my_interval = pd.Interval(0, 1, 'right')#(0,1]
my_interval2=pd.Interval(1,2,'both')#[1,2]
my_interval2.overlaps(my_interval)#是否有交集
# True 有交集{1}
一般而言, pd.IntervalIndex 对象有四类方法生成,分别是 from_breaks, from_arrays, from_tuples, interval_range ,它们分别应用于不同的情况:
from_breaks 的功能类似于 cut 或 qcut 函数,只不过后两个是通过计算得到的风格点,而前者是直接传入自定义的分割点
pd.IntervalIndex.from_breaks([1,3,6,10], closed='both')
# IntervalIndex([[1, 3], [3, 6], [6, 10]],
# closed='both',
# dtype='interval[int64]')
from_arrays 是分别传入左端点和右端点的列表,适用于有交集并且知道起点和终点的情况
pd.IntervalIndex.from_arrays(left = [1,3,6,10],
right = [5,4,9,11],
closed = 'neither')
# IntervalIndex([(1, 5), (3, 4), (6, 9), (10, 11)],
# closed='neither',
# dtype='interval[int64]')
这个不能用cut替代,cut传入的端点必须递增,而from_arrays任意,只需要左端点小于等于右端点就行。
from_tuples 传入的是起点和终点元组构成的列表
pd.IntervalIndex.from_tuples([(1,5),(3,4),(6,9),(10,11)],
closed='neither')
# IntervalIndex([(1, 5), (3, 4), (6, 9), (10, 11)],
# closed='neither',
# dtype='interval[int64]')
一个等差的区间序列由起点、终点、区间个数和区间长度决定,其中三个量确定的情况下,剩下一个量就确定了, interval_range 中的 start, end, periods, freq 参数就对应了这四个量,从而就能构造出相应的区间:
pd.interval_range(start=1,freq=0.5,periods=4)
# IntervalIndex([(1.0, 1.5], (1.5, 2.0], (2.0, 2.5], (2.5, 3.0]],
# closed='right',
# dtype='interval[float64]')
3. 区间的属性与方法
IntervalIndex 上也定义了一些有用的属性和方法。同时,如果想要具体利用 cut 或者 qcut 的结果进行分析,那么需要先将其转为该种索引类型。
与单个 Interval 类型相似, IntervalIndex 有若干常用属性: left, right, mid, length ,分别表示左右端点、两端点均值和区间长度。
s = df.Weight
id_interval = pd.IntervalIndex(pd.cut(s, 3))
id_demo = id_interval[:5] # 选出前5个展示
id_demo
# IntervalIndex([(33.945, 52.333], (52.333, 70.667], (70.667, 89.0], (33.945, 52.333], (70.667, 89.0]],
# closed='right',
# name='Weight',
# dtype='interval[float64]')
id_demo.left
# Float64Index([33.945, 52.333, 70.667, 33.945, 70.667], dtype='float64')
id_demo.right
# Float64Index([52.333, 70.667, 89.0, 52.333, 89.0], dtype='float64')
id_demo.mid
# Float64Index([43.138999999999996, 61.5, 79.8335, 43.138999999999996, 79.8335], dtype='float64')
id_demo.length
# Float64Index([18.387999999999998, 18.334000000000003, 18.333,
# 18.387999999999998, 18.333],
# dtype='float64')
IntervalIndex 还有两个常用方法,包括 contains 和 overlaps ,分别指逐个判断每个区间是否包含某元素,以及是否和一个 pd.Interval 对象有交集。
id_demo.contains(34)
# array([ True, False, False, True, False])
id_demo.overlaps(pd.Interval(40,60))
# array([ True, True, False, True, False])
四、练习
Ex1:统计未出现的类别
在第五章中介绍了 crosstab 函数,在默认参数下它能够对两个列的组合出现的频数进行统计汇总:
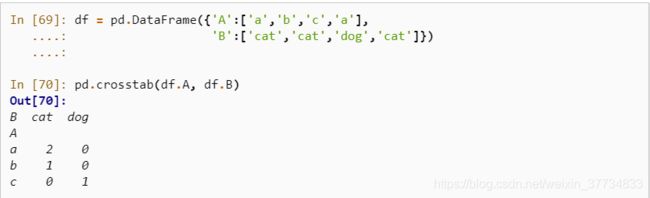
但事实上有些列存储的是分类变量,列中并不一定包含所有的类别,此时如果想要对这些未出现的类别在 crosstab 结果中也进行汇总,则可以指定 dropna 参数为 False :

请实现一个带有 dropna 参数的 my_crosstab 函数来完成上面的功能。
思考过程:
下面是看完答案后按照印象自己敲了一遍,之前一直在想用pivot_table怎么用,因为上次学到crosstab好像说都可以用pivot_table替换。cat获取类别很熟了。主要是之前的一些操作还不太数量,敲完代码熟悉了下df.at的操作,获取指定位置的值。
def my_crosstab(s1,s2,dropna=True):
index1=(s1.cat.categories if s1.dtype.name=='category'
and not drop
else s1.unique())
index2=(s2.cat.categories if s2.dtype.name=='category'
and not drop
else s2.unique())
res = pd.DataFrame(np.zeros((index1.shape[0],
index2.shape[0]))
,index=index1,columns=index2)
for i,j in zip(s1,s2):
res.at[i,j]+=1
res=res.rename_axis(index=s1.name,columns=s2.name).astype('int')
return res
res=res.rename_axis(index=s1.name,columns=s2.name).astype('int')
return res
my_crosstab(df.A,df.B,False)
Ex2:钻石数据集
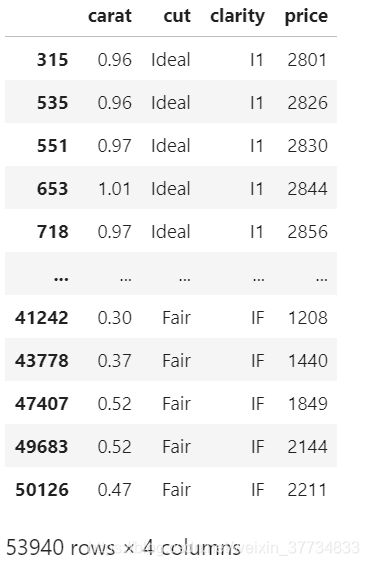
现有一份关于钻石的数据集,其中 carat, cut, clarity, price 分别表示克拉重量、切割质量、纯净度和价格,样例如下:

- 分别对 df.cut 在 object 类型和 category 类型下使用 nunique 函数,并比较它们的性能。
%timeit -n 30 df['cut'].nunique()
#2.02 ms ± 61.1 µs per loop (mean ± std. dev. of 7 runs, 30 loops each)
%timeit -n 30 df['cut'].astype('category').nunique()
#3.24 ms ± 230 µs per loop (mean ± std. dev. of 7 runs, 30 loops each)
这里我跟答案不一致,我的实验结果是object类型的更快,猜测是因为我每次都要单独转换下类型,如果一开始全部转换成category可能会快点。
验证猜想:
x1=df['cut']
x2=df['cut'].astype('category')
%timeit -n 30 x1.nunique()
#2.11 ms ± 151 µs per loop (mean ± std. dev. of 7 runs, 30 loops each)
%timeit -n 30 x2.nunique()
#577 µs ± 14.5 µs per loop (mean ± std. dev. of 7 runs, 30 loops each)
果然一开始直接全部转换成category后速度飞快,类型转换还是比较耗时的。
- 钻石的切割质量可以分为五个等级,由次到好分别是 Fair, Good, Very Good, Premium, Ideal ,纯净度有八个等级,由次到好分别是 I1, SI2, SI1, VS2, VS1, VVS2, VVS1, IF ,请对切割质量按照 由好到次 的顺序排序,相同切割质量的钻石,按照纯净度进行 由次到好 的排序。
# 切割质量次到好分别是 Fair, Good, Very Good, Premium, Ideal ,
# 纯净度有八个等级,由次到好分别是 I1, SI2, SI1, VS2, VS1, VVS2, VVS1, IF ,
# 请对切割质量按照 由好到次 的顺序排序,相同切割质量的钻石,按照纯净度进行 由次到好 的排序
cut_level=['Fair', 'Good', 'Very Good', 'Premium', 'Ideal']
clarity_level=['I1', 'SI2', 'SI1', 'VS2', 'VS1', 'VVS2', 'VVS1', 'IF']
df['cut']=df['cut'].astype('category').cat.reorder_categories(cut_level,ordered=True)
df['clarity']=df['clarity'].astype('category').cat.reorder_categories(clarity_level,ordered=True)
df.sort_values(['cut', 'clarity'], ascending=[False, True])
- 分别采用两种不同的方法,把 cut, clarity 这两列按照 由好到次 的顺序,映射到从0到n-1的整数,其中n表示类别的个数。
我使用和给的答案不一样,比较质朴。
方法一:cat.rename
cut_dict={
'Fair':4, 'Good':3, 'Very Good':2, 'Premium':1, 'Ideal':0}
clarity_dict={
'I1':7, 'SI2':6, 'SI1':5, 'VS2':4, 'VS1':3, 'VVS2':2, 'VVS1':1, 'IF':0}
df['cut'] = df['cut'].astype('category').cat.rename_categories(cut_dict)
df['clarity'] = df['clarity'].astype('category').cat.rename_categories(clarity_dict)
df.head()
| carat | cut | clarity | price | |
|---|---|---|---|---|
| 0 | 0.23 | 0 | 6 | 326 |
| 1 | 0.21 | 1 | 5 | 326 |
| 2 | 0.23 | 3 | 3 | 327 |
| 3 | 0.29 | 1 | 4 | 334 |
| 4 | 0.31 | 3 | 6 | 335 |
方法二:利用reorder_categories和cat.codes,我是相当用codes了,可是没想到逆序怎么办。下面附上答案过程:
df.cut = df.cut.cat.reorder_categories(
df.cut.cat.categories[::-1])
df.clarity = df.clarity.cat.reorder_categories(
df.clarity.cat.categories[::-1])
df.cut = df.cut.cat.codes # 方法一:利用cat.codes
df.clarity = df.clarity.cat.codes
df.head()
| carat | cut | clarity | price | |
|---|---|---|---|---|
| 0 | 0.23 | 0 | 6 | 326 |
| 1 | 0.21 | 1 | 5 | 326 |
| 2 | 0.23 | 3 | 3 | 327 |
| 3 | 0.29 | 1 | 4 | 334 |
| 4 | 0.31 | 3 | 6 | 335 |
方法三:使用replace映射
df.clarity = df.clarity.replace(dict(zip(
clarity_cat, np.arange(
len(clarity_cat)))))
df.head()
| carat | cut | clarity | price | |
|---|---|---|---|---|
| 0 | 0.23 | 0 | 6 | 326 |
| 1 | 0.21 | 1 | 5 | 326 |
| 2 | 0.23 | 3 | 3 | 327 |
| 3 | 0.29 | 1 | 4 | 334 |
| 4 | 0.31 | 3 | 6 | 335 |
- 对每克拉的价格按照分别按照分位数(q=[0.2, 0.4, 0.6, 0.8])与[1000, 3500, 5500, 18000]割点进行分箱得到五个类别 Very Low, Low, Mid, High, Very High ,并把按这两种分箱方法得到的 category 序列依次添加到原表中。
q = [0, 0.2, 0.4, 0.6, 0.8, 1]
point = [-np.infty, 1000, 3500, 5500, 18000, np.infty]
avg = df.price / df.carat
df['avg_cut'] = pd.cut(avg, bins=point, labels=[
'Very Low', 'Low', 'Mid', 'High', 'Very High'])
df['avg_qcut'] = pd.qcut(avg, q=q, labels=[
'Very Low', 'Low', 'Mid', 'High', 'Very High'])
df.head()
| carat | cut | clarity | price | avg_cut | avg_qcut | |
|---|---|---|---|---|---|---|
| 0 | 0.23 | 0 | 6 | 326 | Low | Very Low |
| 1 | 0.21 | 1 | 5 | 326 | Low | Very Low |
| 2 | 0.23 | 3 | 3 | 327 | Low | Very Low |
| 3 | 0.29 | 1 | 4 | 334 | Low | Very Low |
| 4 | 0.31 | 3 | 6 | 335 | Low | Very Low |
- 第4问中按照整数分箱得到的序列中,是否出现了所有的类别?如果存在没有出现的类别请把该类别删除。
df.avg_cut.unique()
df.avg_cut.cat.categories
df.avg_cut = df.avg_cut.cat.remove_categories([
'Very Low', 'Very High'])
df.avg_cut.head(3)
# 0 Low
# 1 Low
# 2 Low
# Name: avg_cut, dtype: category
# Categories (3, object): ['Low' < 'Mid' < 'High']
- 对第4问中按照分位数分箱得到的序列,求每个样本对应所在区间的左右端点值和长度。
interval_avg = pd.IntervalIndex(pd.qcut(avg, q=q))
interval_avg.right.to_series().reset_index(drop=True).head(3)
# 0 2295.0
# 1 2295.0
# 2 2295.0
# dtype: float64
interval_avg.left.to_series().reset_index(drop=True).head(3)
# 0 1051.162
# 1 1051.162
# 2 1051.162
# dtype: float64
interval_avg.length.to_series().reset_index(drop=True).head(3)
# 0 1243.838
# 1 1243.838
# 2 1243.838
# dtype: float64