生信小白的工作笔记
生信小白的工作笔记
目录
- 生信小白的工作笔记
- 前言
- 一、技术栈
-
- 1. GIT
-
- 1.1 download
- 1.2 upload
- 1.3 Q&A
-
- 1. conflict
- 2. fatal: remote origin already exists.
- 3. fatal: unable to access ****git; Connection refused
- 二、 基因
-
- 1. 基因的定义
- 2. 基因特性
- 三、遗传学与医学
-
- 1. 人类基因和基因组
-
- 1.1 基因组的组成
- 1.2 DNA序列的分类
-
- 1.单拷贝序列
- 2.重复多拷贝序列
- 1.3 突变
-
- 1.基因突变
- 2.基因突变的特征
- 3.基因突变的诱发因素
- 4.基因突变的形式
- 2. 单基因病的遗传
-
- 2.1. 常染色体显性遗传病 AD
- 2.2 常染色体隐形遗传 AR
- 2.3 X连锁显性遗传病 XD
- 2.4 X连锁隐性遗传病 XR
- 2.5 Y连锁遗传病
- 2.6 影响单基因遗传病的因素
- 3. 多基因病的遗传
-
- 3.1 质量性状与数量性状
- 3.2 多基因遗传特点
- 3.3 遗传度
- 3.4 遗传多基因遗传病再发风险估计因素
- 4. 人类染色体与染色体病
-
- 4.1 染色质与染色体
- 4.2 染色体畸变
- 4.3 染色体结构畸变及产生机制
- 4.4 常染色体病
- 4.5 性染色体病
- 5. 线粒体病的遗传
-
- 5.1 线粒体
- 5.2 线粒体的遗传特征
- 5.3 线粒体突变与疾病
- 6. 群体遗传学
-
- 6.1 概念
- 6.2 遗传平衡定律
- 6.3 影响遗传平衡的因素
- 四、 泛基因组学
-
- 1. 单一“参考基因组”分析模式的局限
- 2. 泛基因组学”概念的由来和定义
- 3. 构建物种泛基因组的意义
- 4. 人类泛基因组的构建
- 5. 如何记录和表示泛基因组序列
- 五、 生信数据格式
-
- 1. FASTA格式
- 2. FASTQ格式
- 3. BED格式
-
- 3.1 BED格式文件的组成
- 3.2 BED文件的处理
-
- 4. SRA格式
- 5. SAM格式
-
- 5.1 SAM格式文件的组成
- 5.2 如何处理SAM格式数据
- 5.6 BAM格式
- 5.7 CRAM格式
- 5.8 GFF格式
- 5.9 GTF格式
- 5.10 VCF格式
- 六、 WDL
-
- 1. WDL的组成
-
- 1.1 workflow
- 1.2 task
- 2. 任务调度
- 3. 运行WDL脚本
- 七、全基因组测序WGS分析流程
-
- 1. DNA测序技术
-
- 1.1 第一代测序技术——sanger法
- 1.2 第二代测序技术——illumina
- 1.3 第三代测序技术——PacBio SMRT技术(纳米孔单分子技术)
- 1.4 第三代测序技术——Oxford Nanopore
- 2. 四大测序技术的优缺点
- 2. FASTA&FASTQ
-
- 2.1 FASTA
- 2.2 FASTQ
- 2.3 碱基质量值
- 2.4 质量体系
- 3. WGS主流程
-
- 3.1 原始数据质控
- 3.2 数据预处理
-
- 1. 序列比对
- 2. 排序
- 3. 去除重复序列(或者标记重复序列)
- 4. 局部重比对
- 5. 重新校正碱基质量值(BQSR)
- 3.3 变异检测
-
- 1. 变异检测
- 2. 变异检测质控和过滤(VQSR)
- 八、转录组数据分析处理
-
- 1. 转录组的建库与质量控制
-
- 1.1 人类基因组与转录组的组成
- 1.2 转录组的2种建库策略
-
- 1.正选择——polyA positive
- 2. 负选择——rRNA minus
- 1.3 两种建库方法比较
- 九、python并发编程
-
- 1. 基础知识
前言
作为一名生信小白,在工作中需要补充学习的知识很多。那么我将会把工作中学习的知识记录下来。
和我一起学习吧,加油!
一、技术栈
1. GIT
1.1 download
$ git clone http://172.18.0.1:5004/zhanganqi/nebula_pipelines.git
1.2 upload
upload
$git init 建立git仓库
$git add . 将当前路径下的文件 添加到待上传文件列表中
$git commit -m "xxx" 将add的文件commit到仓库
$git remote add origin http://172.18.0.1:5004/zhanganqi/nebula_pipelines.git 将本地的仓库关联到github上
$git fetch origin develop
$git pull origin develop
$git push origin develop
1.3 Q&A
1. conflict
内容冲突
两个用户修改了同一个文件的同一块区域,git会报告内容冲突。常见的都是这种冲突。
一般来讲,出现冲突时都会有“CONFLICT”字样:
$ git pull
Auto-merging pipelines/dnaseq/wgs_ch1k.py
Removing gatk4.py
Auto-merging bfx/gatk4.py
CONFLICT (content): Merge conflict in bfx/gatk4.py
Automatic merge failed; fix conflicts and then commit the result.
merge/patch的冲突解决
先编辑冲突,然后git commit提交。
注:对于git来讲,编辑冲突跟平时的修改代码没什么差异。修改完成后,都是要把修改添加到缓存,然后commit。
最简单的编辑冲突的办法,就是直接编辑冲突了的文件(test.txt),把冲突标记删掉,把冲突解决正确。
冲突产生后,文件系统中冲突了的文件(这里是test.txt)里面的内容会显示为类似下面这样:
a123
<< << << < HEAD
b789
== == == =
b45678910
>> >> >> > 6853e5ff961e684d3a6c02d4d06183b5ff330dcc
c
其中:冲突标记<<<<<<< (7个<)与=======之间的内容是我的修改,=======与>>>>>>>之间的内容是别人的修改。
此时,还没有任何其它垃圾文件产生
但是,也有例外,repo sync的报错,可能并不是直接提示冲突,而是下面这样:
error : project mini /sample
注:无论是否存在冲突,只要本地修改不是基于服务器最新的,它都可能报告这个错误,解决方法都是一样。
这个时候,需要进入报错的项目(git库)目录,然后执行git rebase解决:
$git rebase remote -branch -name
rebase的冲突解决
rebase的冲突解决过程,就是解决每个应用补丁冲突的过程。
解决完一个补丁应用的冲突后,执行下面命令标记冲突已解决(也就是把修改内容加入缓存):
$git add -u
注:-u 表示把所有已track的文件的新的修改加入缓存,但不加入新的文件。
然后执行下面命令继续rebase:
$git rebase -- continue
有冲突继续解决,重复这这些步骤,直到rebase完成。
如果中间遇到某个补丁不需要应用,可以用下面命令忽略:
$git rebase --skip
如果想回到rebase执行之前的状态,可以执行:
git rebase --abort
注:rebase之后,不需要执行commit,也不存在新的修改需要提交,都是git自动完成。
树冲突
文件名修改造成的冲突,称为树冲突。
比如,a用户把文件改名为a.c,b用户把同一个文件改名为b.c,那么b将这两个commit合并时,会产生冲突。
$ git status
added by us : b.c
both deleted : origin -name.c
added by them : a.c
如果最终确定用b.c,那么解决办法如下:
$git rm a.c
$git rm origin -name.c
$git add b.c
$git commit
树冲突也可以用git mergetool来解决,但整个解决过程是在交互式问答中完成的,用d 删除不要的文件,用c保留需要的文件。
最后执行git commit提交即可。
2. fatal: remote origin already exists.
解决方法:
1. 先输入 git remote rm origin
2. 再输入 git remote add origin**************
3. fatal: unable to access ****git; Connection refused
git config --list
git config --global --unset http.proxy
二、 基因
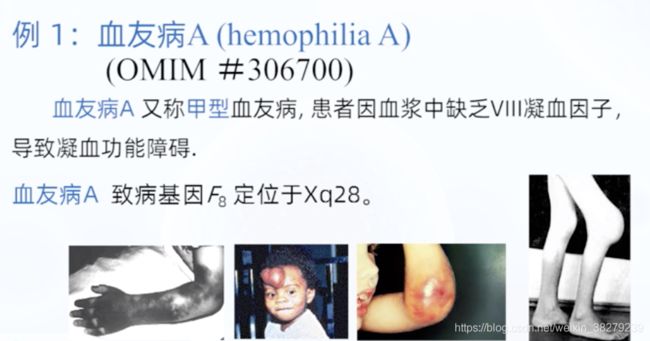
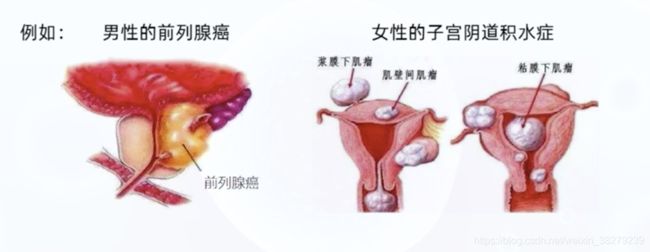
基因是控制性状的基本遗传单位,决定着人的生老病死,是健康、靓丽、长寿之因,是生命的操纵者和调控者。人类约有两万至两万五千个基因。
首先,我们应该了解一下染色体,DNA,RNA,基因之间的关系。
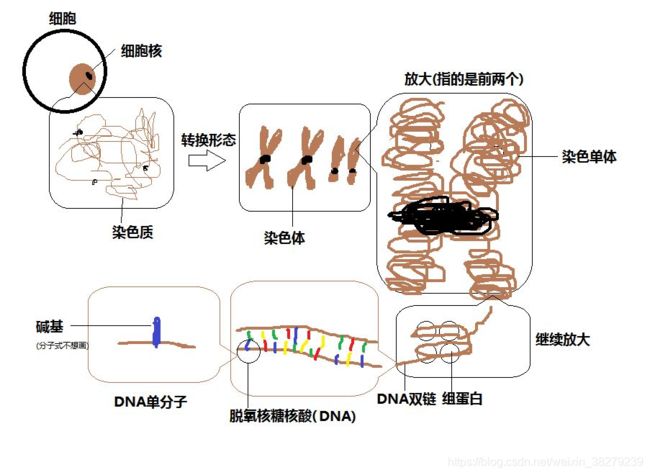
1. 基因的定义
基因是控制生物性状的基本遗传单位。
基因(遗传因子)是产生一条多肽链或功能RNA所需的全部核苷酸序列。
也就是说,基因只是DNA链中的一个个片段。每一片段会控制不同的性状。
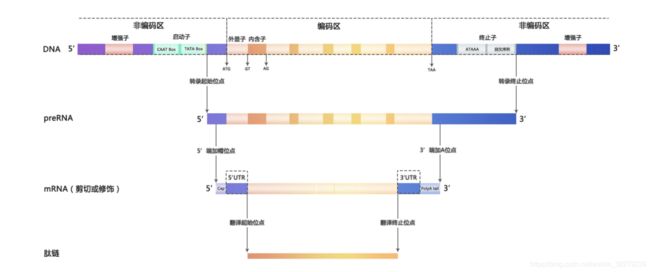
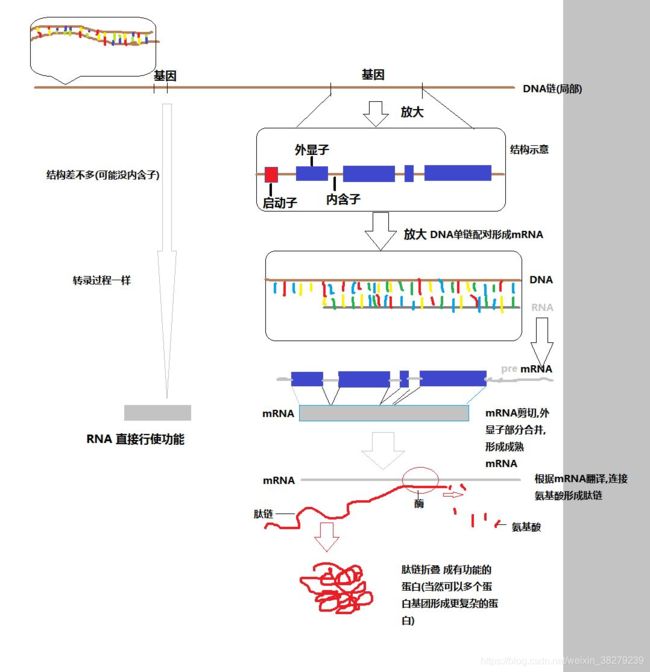
按照基因结构,可分为编码区和非编码区。
编码区
真核生物的编码区是不连续的,分为外显子和内含子,在转录过程中会修剪内含子,并拼合外显子来形成转录产物。在原核生物中,基因是连续的,也就是说无外显子和内含子之分。
编码区就是可以转录为mRNA,合成肽链形成蛋白质的那部分。
外显子 Exon
外显子是在 preRNA 经过剪切或修饰后,被保留的DNA部分,并最终出现在成熟RNA的基因序列中。
内含子 Intron
在真核生物中,内含子作为阻断基因的线性表达的一段DNA序列,是在 preRNA 经过剪切或修饰后,被切除的DNA序列。
真核生物基因的编码区是不连续的,内含子和外显子是交替穿插的,所以转录形成的原始的RNA需进行剪切,将内含子部分剪切掉进行拼接形成成熟的mRNA。
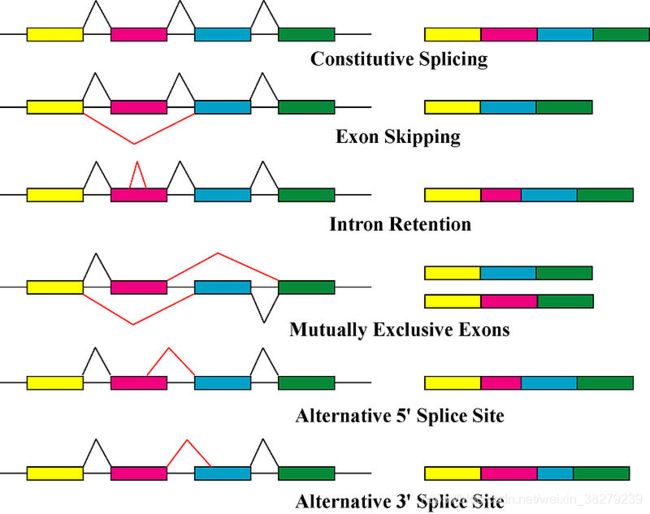
通常,是要将内含子剪切掉。但是,还会有其他的剪切方式,选择性的保留内含子,或者并不按照线性次序进行剪切,也就是可变剪切。
通过外显子的不同组合方式,可以产生一系列不同的转录本,最终使得生物体内的转录组和蛋白质组呈现多样性。在这个过程中,可变剪接起着关键的作用。在人类中,绝大多数具有蛋白编码能力的基因都能受到可变剪接的调控,产生多种转录本,最终产生多种不同功能的蛋白亚型。而异常的可变剪接也广泛涉及到了多种疾病,例如癌症等。
可变剪切
指的是在mRNA前体到成熟mRNA的过程当中,不同的剪切方式使得同一个基因可以产生多个不同的成熟mRNA, 最终产生不同的蛋白质。
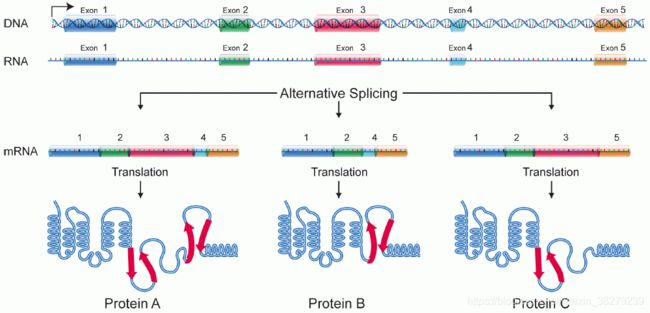
可变剪切在真核生物体内广泛存在,有研究指出,对于人类基因组中包含多个exon的基因而言,其中有95%的基因都存在可变剪切现象。可变剪切导致了转录本和蛋白质结构与功能的多态性,是一种重要的转录调控机制。
变剪切
变剪切依据外显子之间连接位置的不同,又可以具体细分为如下几种剪切方式:
1. 组成型拼接
2. 外显子跳跃拼接
3. 内含子保留拼接
4. 相互排斥的外显子拼接
5. 替代5’端剪切
6. 替代3'端剪切
非编码区
非编码区在对基因的表达调控中发挥重要作用,如启动子,增强子,终止子等都位于该区域,在人类基因中非编码区的占比超过90%。
它们中的一部分可以转录为功能性RNA,比如tRNA(transfer RNA), rRNA(ribosomal RNA)等;可以作为DNA复制,转录起始来对复制,转录和翻译起到调控作用;也可能是着丝粒与端粒的重要组成部分。
1.启动子-Promoter
启动子是基因的重要组成部分,它的主要功能是控制基因表达(转录)的起始时间和表达的程度。启动子就像“开关”,决定基因的活动。但启动子本身并不控制基因活动,而是通过与转录因子结合而控制基因活动。
启动子活性的异常,则可能导致基因表达的调节障碍,从而有可能导致疾病的发生
找到组织特异性启动子,为靶向治疗提供可能
找到某些疾病关键基因异常表达与启动子的关系,为基因治疗提供可能
启动子是特定基因转录的DNA区域,启动子一般位于基因的转录起始位点,5‘端上游,启动子长约100-1000bp。在转录过程中,RNA聚合酶与转录因子可以识别并特异性结合到启动子特有的DNA序列(一般为保守序列),从而启动转录。启动子本身并不转录而且也不控制基因活动,而是通过与转录因子结合来调控转录过程。在细胞核中,似乎启动子优先分布在染色体区域的边缘,可能是在不同染色体上共同表达基因。 此外,在人类中,启动子显示出每个染色体特有的某些结构特征。
CCAAT
CCAAT box(有时也缩写为CAAT box或CAT box):具有GGCCAATCT 共有序列的不同核苷酸序列 ,是真核生物基因常有的调节区,位于转录起始点上游约-80bp处,可能也是RNA聚合酶的一个结合处,控制着转录起始的频率。与之相似的是,在原核生物启动子上-35bp处的TTGACA区,又称-35区。
CAAT框是最早被人们描述的常见启动子元件之一,常位于接近-80的位置,但是它可以在离起始点较远的距离仍能起作用,且在两种取向均可发挥作用。CAAT框的突变敏感性提示了它在决定转录效率上有很强的作用,但是突变对启动子的特异性没有影响。
TATA 框
TATA(TATA box / Goldberg-Hogness box),存在于古细菌和真核生物的核心启动子区域的一段DNA序列,TATA 框的原核同源物称为Pribnow 框(Pribnow box),其具有较短的共有序列TATAATAAT。 它约在多数真核生物基因转录起始点上游约-30bp(-25~-32bp)处,基本上由A-T碱基对组成,是决定基因转录始的选择,为RNA聚合酶的结合处之一,RNA聚合酶与TATA框牢固结合之后才能起始转录。
2.增强子-Enhancer
增强子是位于转录起始位点或下游基因1Mbp的位置,长度50-1500bp的序列,其可以被转录激活因子结合从而增加特定基因转录发生的可能性,广泛的存在于原核与真核生物基因结构中。
增强子能大大增强启动子的活性。
增强子有别于启动子处有两点:增强子对于启动子的位置不固定,而能有很大的变动;它能在两个方向产生相互作用。一个增强子并不限于促进某一特殊启动子的转录,它能刺激在它附近的任一启动子。
3.终止子-Terminator
终止子处于基因或操纵子的末端,给RNA聚合酶提供转录终止信号的DNA序列。
ATAAA
ATAAA 是 preRNA 在通过修剪后形成成熟mRNA 时在3’UTR产生ployA 是的加尾信号。但是这段序列并不是绝对保守,也可能为其他A富集的序列,比如AATAAA等。
回文序列是双链DNA中的一段倒置重复序列,这段序列有个特点,它的碱基序列与其互补链之间正读和反读都相同。
5’ GGTACC 3’
3’ CCATGG 5
当该序列的双链被打开后,如果这段序列较短,有可能是限制性内切酶的识别序列,限制性内切酶就是一个基因剪刀,于是就可以剪断啦。
但是如果比较长,有可能形成发卡结构,这种结构的形成有助于DNA与特异性DNA蛋白质的结合。
preRNA
前体RNA,需进行可变剪切形成成熟的mRNA
转录起始位点 Transcription start sites (TSS)
转录起始位点是指与新生RNA链第一个核苷酸相对应的DNA链上的碱基,通常为一个嘌呤(A 或G),即5’UTR的上游第一个碱基。
5’末端的序列称为上游,而把其后面即3‘末端的序列称为下游.
转录终止位点 Transcription termination sites (TTS)
转录终止位点是指新生RNA链最后一个核苷酸相对应的DNA链上的碱基。
当RNA链延伸到转录终止位点时,RNA聚合酶不再形成新的磷酸二酯键,RNA-DNA杂合物分离,转录泡瓦解,DNA恢复成双链状态,而RNA聚合酶和RNA链都被从模板上释放出来。
开放阅读框 Open reading frame(ORF)
ORF 是连续的一段密码子,其含有起始密码子(通常是AUG)和终止密码子(通常是UAA,UAG或UGA)。
在真核基因中,ORF跨越内含子/外显子区域,其可以在 ORF 转录后拼接在一起形成成熟的mRNA。 由于读写位置不同(对应不同的起始位点),ORF 可能翻译为不同的多肽链。在没有其它信息的前提下,DNA序列可以按六种框架阅读和翻译(每条链三种,对应三种不同的起始密码子)。
如果遗传密码是不重叠的三联体,那么会有三种可能的方式将核苷酸翻译成蛋白质, 这三种可能的读码(Reading frame ) 方式称为读码框架。
比如序列:ACGACGACGACGACGACG,可能的读码框架就有以下三种:
ACG ACG ACG ACG ACG ACG ACG ACG
CGA CGA CGA CGA CGA CGA CGA CGA
GAC GAC GAC GAC GAC GAC GAC GAC
一段翻译成蛋白质的序列有一个阅读框架,它有一个特殊的起始密码子,从此延伸出一系列代表氨基酸的三联体,一直到在三种类型的终止密码子上结束。如果终止密码子频繁出现,就会阻止阅读框被翻译成蛋白质。一个序列的三个阅读框全部被阻断,那么它就会失去翻译成蛋白质的功能。 当获得一个未知的DNA 序列后,就可分析其三个读码框是被阻断的还是开放的。在任何一段DNA 中,通常不会超过一个读码框是开放的 ,因为替换的读码框被频繁出现的终止密码子阻断。证明序列是开放框是确定该框架能翻译为蛋白质的首要证据。一个不能表达蛋白质的开放读框被称为不确定读框(URF) 。
一个DNA顺序可能有3种阅读框,但只有一种具有编码的作用称为开放阅读框(open reading frame or ORF)。有的阅读框因终止密码出现频繁故不能生成蛋白,这种阅读框称为封闭阅读框(block reading frame)。若一个顺序所有的三个阅读框都是封闭的,则它无编码蛋白的功能。一个翻译成蛋白的顺序有一个阅读框,开始于AUG起始密码子,通过一系列有义密码子,直到终止密码子结束。通常3个阅读框中总有封闭阅读框的存在。
例如一段5’-UCUAAAGGUCCA-3’序列。此序列共有3种读取法:
UCU AAA GGU CCA
CUA AAG GUC
UAA AGG UCA
由于UAA为终止编码,因此第三种读取法不具编译出蛋白质的潜力,故只有前两者为开放阅读框架。
ORF识别包括检测这六个阅读框架并决定哪一个包含以启动子和终止子为界限的DNA序列而其内部不包含启动子或密码子,符合这些条件的序列有可能对应一个真正的单一的基因产物。ORF的识别是证明一个新的DNA序列为特定的蛋白质编码基因的部分或全部的先决条 件。当获得了一个未知功能的DNA区域的顺序,要通过分析来确定阅读框是开放的还是封闭的。在任何一个DNA顺序中往往只有一个开放阅读框。
mRNA
最终得到成熟的mRNA,可翻译成肽链合成蛋白质。
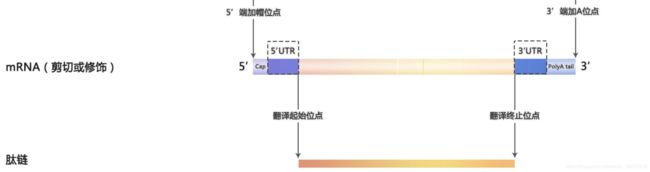
5’UTR 与 3’UTR
这里需要注意的是外显子包含UTR区,也就是说外显子不只有可编码的序列,而且包含非编码序列
UTR (Untranslated Region ),如果这段序列位于5’端,就称作5’UTR(5‘-untranslated region),也叫前导序列(leader)。相反若位于3’端,我们就叫它3’UTR(3‘-untranslated region),也叫尾随序列(trailer)。
5’UTR 位于从mRNA起点的甲基化鸟嘌呤核苷酸帽延伸至起始密码子AUG,3’UTR从编码区末端的终止密码子延伸至多聚A尾巴(Poly-A)的前端。
原核生物和真核生物都可以看到UTR,但它们的长度和组成都有所不同。原核生物中,5′非翻译区通常为3至10个核苷酸的长度。但在真核生物中,5′非翻译区有成百上千个核苷酸的长度。与原核生物相比,真核生物的基因组的复杂性更高,3′非翻译区的长度也不同。虽然5′非翻译区和3′非翻译区在长度上有差异,但5′非翻译区的长度在演化过程中比3′非翻译区显得更保守2。
5‘Cap
5‘Cap也被称为7-甲基鸟苷酸帽,缩写为m7G。
这种结构在RNA进出细胞核起到识别作用;可以抗5’-核酸外切酶的截切;促进5’端内含子的切除;在翻译过程中有助于核糖体对mRNA的识别和结合。
3’ PolyA tail
Poly A tail 由多个腺苷一磷酸组成 ,也就是说它是一段仅含有腺嘌呤碱基的RNA 。这种结构可以避免细胞质中的酶促降解,并有助于转录终止,mRNA从细胞核中的输出和翻译。
CDS (coding dna sequence)
CDS 是基因中DNA或RNA为蛋白质编码区域
该区域通常开始于5‘末端的起始密码子并结束于3’端的终止密码子。生物体基因组编码区的总和称为外显子组。
2. 基因特性
基因座
基因在染色体上的位置称为座位,每个基因都有自己特定的座位。
一个基因座可以是一个基因,一个基因的一部分,或具有某种调控作用的DNA序列。
二倍体与多倍体细胞的某些染色体上,在同一基因座上有相同的等位基因,这类细胞称为同型合子(纯合子)。若是相同基因座上含有不同的等位基因,则称作异型合子(杂合子)。
等位基因
位于一对同源染色体相同位置上控制同一性状不同形态的基因。
等位基因之间存在相互作用。当一个等位基因决定生物性状的作用强于另一等位基因并使生物只表现出其自身的性状时,就出现了显隐性关系。作用强的是显性,作用被掩盖而不能表现的为隐性。
一对呈显隐性关系的等位基因,显性完全掩盖隐性的是完全显性(complete dominance),两者相互作用而出现了介于两者之间的中间性状,如红花基因和白花基因的杂合体的花是粉红色,这是不完全显性(incomplete dominance)。有些情况下,一对等位基因的作用相等,互不相让,杂合子就表现出两个等位基因各自决定的性状,这称为共显性(codominance)。
野生型vs突变型
在自然群体中往往有一种占多数的(因此常被视为正常的)等位基因,称为野生型基因。同一座位上的其他等位基因一般都直接或间接地由野生型基因通过突变产生,相对于野生型基因,称它们为突变型基因。
野生型(wild type)用来描述自然界中常见的基因型和表现型。野生型等位基因都产生有功能的蛋白质。
突变型等位基因最常见的是丧失功能型(loss-of-function),绝大多数产生改变了的蛋白质,极少数根本不产生蛋白质。
所以,野生型对突变型而言是显性。但是,如果突变型等位基因是获得功能型(gain-of-function),产生的蛋白质赋予生物体以新的性状,此时突变型等位基因则为显性。
复等位基因
在二倍体的生物群体中等位基因往往不止两个,两个以上的等位基因称为复等位基因。
拟等位基因
一部分早期认为是属于复等位基因的基因,实际上并不是真正的等位,而是在功能上密切相关、在位置上又邻接的几个基因,所以把它们另称为拟等位基因。
同等位基因
有某些表型效应差异极少的复等位基因的存在很容易被忽视,通过特殊的遗传学分析可以分辨出存在于野生群体中的几个等位基因。这种从性状上难以区分的复等位基因称为同等位基因。
许多编码同工酶的基因也是同等位基因。
非等位基因
非等位基因之间也存在相互作用。位于同一染色体的不同基因座,或位于不同染色体上的非等位基因,都可能影响到同一性状。
某些性状只有同时存在若干个非等位基因时才会出现,当其中任何一个非等位基因发生改变时,都会导致产生同一种突变性状。这些非等位基因称为互补基因(complementary gene)。又如,有些基因本身没有可观察到的表型效应,但可以抑制其他非等位基因的活性,这就是抑制基因(inhibitor)。
三、遗传学与医学
医学遗传学是目前医学中最前沿的学科,也是新兴学科。是研究出生缺陷生命科学主要的课题。医学遗传学主要利用DNA技术来研究疾病与基因的关系。开展新型的诊断技术和治疗方法。可以从分子水平为疾病的早期诊断,预防出生缺陷、以及疑难杂症的诊断和治疗提供更高效的新型医学服务。包括基因诊断,基因治疗等。
像目前有些功能性疾病如耳聋、弱视、斜视、色盲、狐臭,智能发育低下,都可以通过产前诊断进行产前干预或宫内基因治疗。
1. 人类基因和基因组
基因及其结构
基因是细胞内遗传物质的结构和功能单位
以脱氧核糖核酸(DNA)的化学形式存在于染色体上
DNA分子组成
基本单位为 脱糖核苷酸
脱氧核苷酸的组成为 一分子脱氧核糖 一分子磷酸 一分子含氮碱基
碱基有四种 == A腺嘌呤T胸腺嘧啶C胞嘧啶G鸟嘌呤==
4种脱氧核苷酸按一定顺序首尾相接构成脱氧多核苷酸单链
每条脱氧核苷酸单链都有3’端 (-OH)和 5‘端 (磷酸基团)
相邻脱氧核苷酸通过磷酸二酯键相连
DNA分子双螺旋结构
DNA由两条碱基互补反向平行排列 脱氧多核苷酸单链组成
一条5’-3’ 方向 一条 3’-5’方向
AT之间2个H键相连 CG之间3个H键相连
两条互补链围绕一个“主轴”向右盘旋双螺旋结构
双螺旋表面形成两条凹槽 分为大沟小沟
两条沟 对有特定功能蛋白质或酶识别调节DNA上遗传信息非常重要
DNA上碱基排列组合方式无限
基因结构
绝大多数真核生物编码蛋白质的基因为 割裂基因
割裂基因由 编码序列(外显子exon)和非编码序列(内含子intron)组成 两者相间排列
侧翼序列
每个割裂基因在第一个和最后一个外显子的外侧 都有一段不被转录的非编码区 为侧翼序列
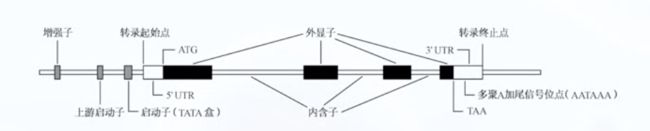
侧翼序列包含 启动子 增强子 以及 终止子, 它们对基因的有效表达起调控作用
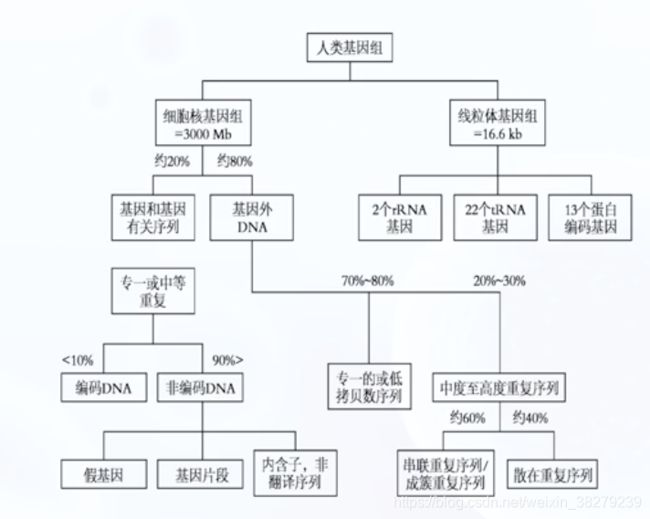
1.1 基因组的组成
人类基因组
人体所有遗传信息的总和,包括相对独立又互相关联的 核基因组和线粒体基因组。若不特殊说明 通常指核基因组。
人类基因组约有2w-2.2w个基因 ,与蛋白质合成有关的为1.1% ;4%为基因调控序列 和 RNA基因序列
1.2 DNA序列的分类
根据DNA序列在基因组出现的次数多少可划分为 单拷贝序列 和 重复多拷贝序列
1.单拷贝序列
在基因组中只出现一次或少数几次
大多数编码蛋白及酶类基因及结构基因为单一序列,即非重复序列。单拷贝或低拷贝序列可占人类基因组大约45%
2.重复多拷贝序列
在基因组中有很多拷贝的DNA序列
根据重复序列来源分布特点 可分为 串联重复序列 和 散在重复序列
串联重复序列
以5bp 10bp 20bp 或 200bp为一个重复单位 重复很多次 约占整个基因组10%
- 卫星DNA
重复次数多,长度达几百kbp
存在于异染色质区 - 小卫星DNA 可变数目串联重复 VNTR
15bp-100bp的组成单位,重复20-50次
形成0.1-20kb的短DNA
多数VNTR用于标记,少数与疾病相关 - 微卫星DNA 短串联重复 STR
基因组的间隔序列 与 非编码区
重复序列1-6bp ,长度小于100bp
表现多态性 可拥于基因定位 群体进化 基因诊断的研究
散在重复序列
以分散方式分布于整个基因组内的重复序列 约占45%
- 短散在核元件 SINES 100-400bp
人类基因组含量最丰富的散在重复序列,平均每3kb发生一次以上重复
占基因组总DNA含量11% 长达300bp 重复30w-50w次
典型 :Alu 重复
含有限制性内切酶Alu I的特异性识别位点 AGCT 可被Alu I酶解为170bp和130bp的两个片段
- 长散在核元件 LINES 5k-7kbp
典型:Kpn I家族
可由限制性内切酶Kpn I切割
1.3 突变
突变指遗传物质发生可遗传变异
突变类别可分为
- 染色体畸变:染色体数目和结构的改变
- 基因突变:DNA碱基对组成与序列的变化
1.基因突变
- 生殖细胞突变 可通过有性生殖传递后代个体
- 体细胞突变 不会传递后代个体 可通过突变细胞分裂增殖形成突变细胞克隆
2.基因突变的特征
- 多向性
任何基因座上的基因都有可能独立的发生多次不同的突变 而形成新的等位基因
如在不同条件下 位于染色体某一基因座上的基因A可突变为其等位基因a1 a2 a3等 从而形成复等位基因
群体中存在于同一基因座上 决定同一类相对性状 具有三种或三种以上不同形式的等位基因成为复等位基因
如ABO血型系统 由位于9号染色体长臂三区四带这一区域同一基因座上的Ia Ib 和 i 三种等位基因形式所构成的一组复等位基因所决定的
- 可重复性
对于任何一个基因位点 并不是只发生一次或少数几次突变 突变总是以一定的频率重复出现
- 随机性
突变的发生对于不同个体 细胞或不同基因来说都是随机的
- 稀有性
自然状态下 各种生物的突变率都是很低的
如人类自发基因突变率 约为每1w-100w个生殖细胞中 有一个基因发生突变
- 可逆性
基因突变的方向是可逆的
任何一种野生型基因A都能够通过突变而形成其等位的突变型基因a 为正向突变
a也可突变为A 为回复突变
一般情况下 正向突变率远远高于回复突变率
- 有害性
通常基因突变会对个体产生不利影响 但也是相对的并非所有基因突变都会有害
3.基因突变的诱发因素
- 自发突变
自然条件下 没有人为干涉未经任何人工处理而发生的突变
- 诱发突变
在人为干涉下 经过特殊的人工处理所产生的突变
无论是自发突变还是诱发突变 都有内外环境因素的影响
诱变剂
凡是能够诱发基因突变的各种内外环境因素
诱变剂分类
- 物理因素
辐射:紫外线辐射 电离辐射
紫外线辐射引起的损失 DNA分子相邻嘧啶碱的二聚体化 最常见的是胸腺嘧啶二聚体造成碱基互补配对的错误 影响新合成链中碱基的组成
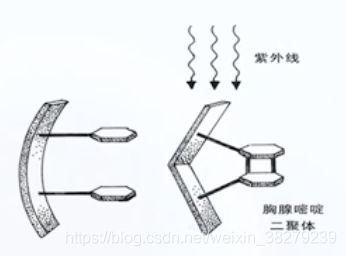
电离辐射包括电磁辐射 如X-射线 y射线 特殊辐射 粒子辐射
电离辐射 引发遗传物质内部的辐射化学反应 导致染色体和DNA分子的断裂性损伤
射线的诱变作用 不仅与其一次的照射强度或剂量相关 还具有照射强度和剂量的累积效应
- 化学因素
碱基类似物 亚硝酸类化合物 羟胺类 烷化剂类物质 芳香族化合物
碱基类似物 掺入DNA分子中而取代某些正常碱基 引起突变发生
亚硝酸类物质 可引起碱基脱氨基作用 造成原有碱基分子结构和化学性质的改变
羟胺类 引起DNA分子中胞嘧啶发生化学组分的改变 不能与其互补碱基鸟嘌呤正常配对 转而与腺嘌呤配对结合 两次复制后 原本的C-G 突变为 A-T
- 生物因素
病毒 风疹 麻疹 流感 疱疹
细菌和真菌 产生毒素或代谢物
如生活于花生玉米中的黄曲霉菌产生的黄曲霉素 是肝癌发生的重要诱发因素之一
4.基因突变的形式
点突变
又称碱基替换
碱基替换的方式有转换和颠换两种
同类碱基中的替换为转换 即嘌呤->嘌呤 嘧啶->嘧啶
异类碱基的替换为颠换 即嘌呤->嘧啶 嘧啶->嘌呤
密码子区突变
若突变发生在基因的编码序列 mRNA的密码子相应改变
-
同义突变
新旧密码子编码的氨基酸种类不变 不产生突变效应 -
错义突变
碱基替换导致编码氨基酸变为另一种氨基酸 多肽链发生改变 影响蛋白质功能
人类许多分子病和代谢病由此造成 -
无义突变
使编码氨基酸的密码变成终止密码子 UAA UAG UGA 多肽链提前终止 导致蛋白质功能异常或缺失
导致遗传表型改变的致病效应 -
终止密码突变
终止密码子突变 成为氨基酸的密码子 形成延长的异常多肽链
非密码子区突变
引起的可能是调控序列或内含子与外显子的剪接位点的突变
内含子与外显子剪接位点突变
GT-AG中任一碱基发生置换 导致剪辑加工异常 不能形成正确的mRNA
移码突变
DNA链上插入或丢失1个 2个 甚至多个碱基 (但不是三联体密码子及其倍数)
导致读码时 密码子移位 导致插入或丢失点以后的编码都发生改变
引发的遗传学效应往往比较严重,导致一条或多条多肽链的合成障碍或功能缺陷甚至完全丧失
整码突变
又称密码子的插入或缺失,指在DNA链中增加或减少的碱基对为一个或几个密码子,此时基因产物多肽链中会增加或减少一个或几个氨基酸,而此部位之后的氨基酸序列无改变。
密码子插入或丢失,插入或丢失前后的氨基酸种类和序列不变
动态突变
基因编码序列及基因侧翼序列中的短串联重复的核苷酸序列重复扩增 随世代传递而出现累加突变效应 为动态突变
由此突变引起的疾病统称为三核苷酸重复扩增疾病
三核苷酸扩增的显著特点是其突变的动态性 即 不稳定性
正常人类基因组中 已发现的三核苷酸扩增的致病位点大多具有40个以下重复顺序,异常扩增超过一定范围则导致疾病
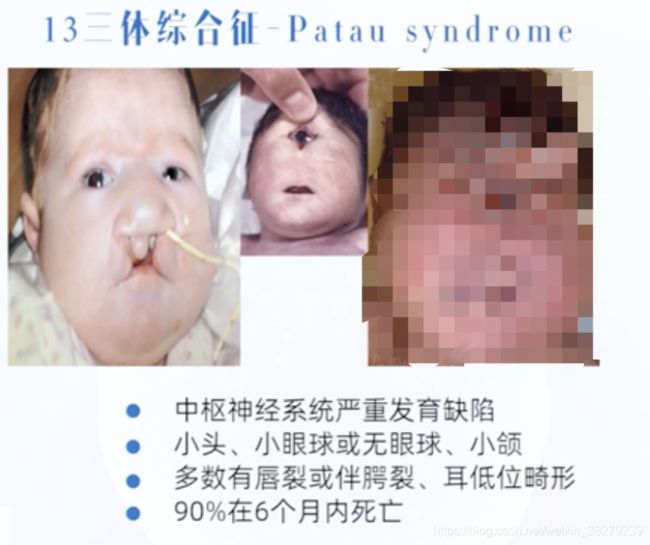
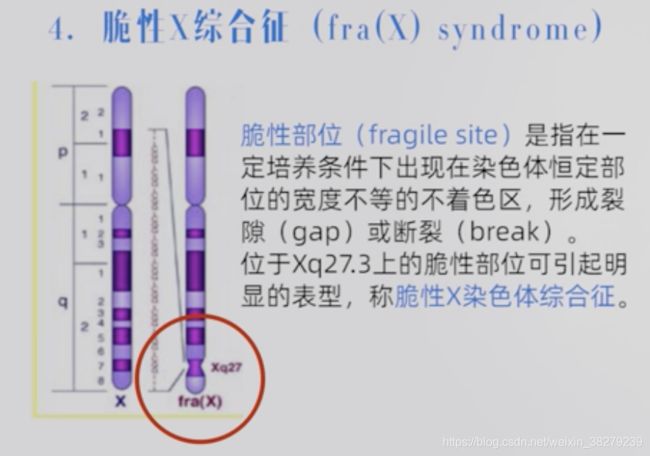
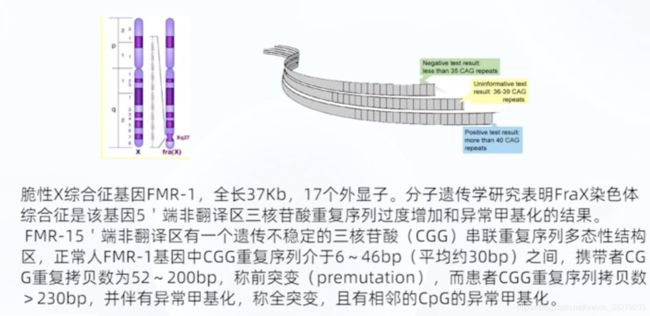
- 脆性X综合症
Xq27.3, FMR-1 基因
5’端非翻译区(CGG)n重复数:
正常 6-50 最常见29-30个重复
前突变 60-200 无临床表现的携带者
全突变 230-1000 将出现智力低下及其他一些脆性X综合症的特征
重复序列异常扩增导致疾病发生 其不稳定的序列长度n越长 症状越严重
- 强直性肌营养不良
19q13.3 19号染色体长臂1区3带3亚带
可扩增三核苷酸重复顺序CTG 位于基因3’端非翻译区
正常人群中 (CTG)n重复长度在5-37之间 扩增后致病突变 其长度通常大于50个
临床症状严重程度和扩增突变的长度有较好的相关性
若扩增重复顺序长度在150以下 通常只有轻微症状
150-800 在成年后逐渐出现临床症状
超过800 可为先天性病患 临床症状严重 而且在婴儿期有很高死亡率
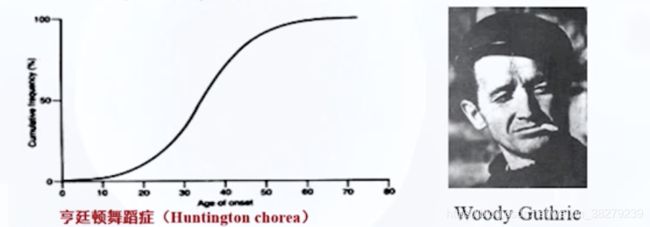
- 亨廷顿舞蹈症 脊髓肌萎缩症等
目前已知近20种疾病的发生是动态突变所导致的
2. 单基因病的遗传
定义
由一对等位基因控制而发生的遗传性疾病,这对等位基因成为主基因
单基因遗传病的遗传 可分为核基因遗传病 和 线粒体基因遗传病
核基因遗传病 上下代之间传递遵循孟德尔定律
基因定位表示
染色体序号:
1~22号染色体或XY;
染色体的臂号:
长臂q或短臂p;
区的序号:
“区”为位于染色体臂上两相邻界标(“界标”是染色体上恒定的、有显著形态特征的带,包括染色体两臂的末端、着丝粒以及臂上某些显著的带)之间的区域.区的序号是从着丝粒部位向两臂远端依次编号;-
带的序号:
“带”是染色体上宽窄各异、明暗相间的横纹.带的序号从着丝粒侧向臂的远端依次编号,作为界标的带属于该界标以远区的第1条带;
亚带和次亚带的序号:
亚带和次亚带是在带的基础上逐级细分出来的.亚带写在带号的后面,以小数点相隔,编号原则也是从着丝粒的近侧向远侧依次编号;次亚带直接写在亚带后,不加标点.
例如,1q42.13表示1号染色体长臂4区2带1亚带3次亚带.
等位基因
人群中某一基因发生结构改变而与原DNA结构组成有所不同
这一新的基因结构为原基因的等位基因
用大写字母表示显性基因
用小写字母表示隐性基因
基因型
一个个体的遗传结构和组成 ,一般指特定基因座上的等位基因的构成
一个基因座上的基因一般是成对存在的 两个基因分别来自于父母
AA 显性纯合子 aa 隐形纯合子 统称纯合子 Aa 杂合子
同源染色体
形态结构和功能相似的一对染色体
一条来自父亲 一条来自母亲
表型/ 表现型
基因型所表现的或所表达 能够显示出的遗传性状
除表现的性状 检测的结果 如细胞形态 酶的活性 临床化验结果等
显性性状
在杂合状态下 受某等位基因所控制的性状能表现出来时 该性状为显性性状 Aa
隐性性状
在杂合状态下 受某等位基因所控制的性状不能表现出来时 该性状为隐性性状 aa
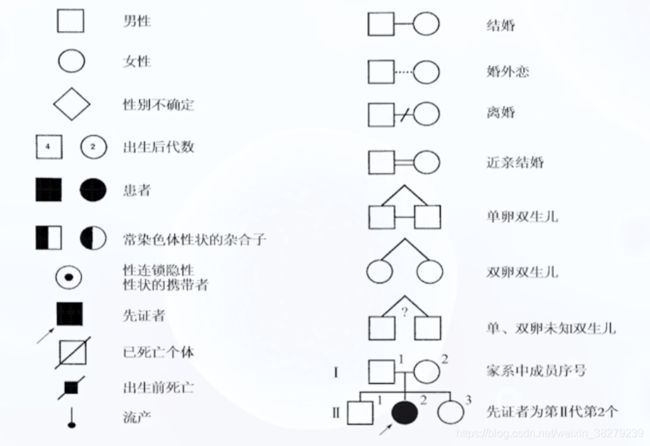
系谱
从先证者入手 追溯调查所有家族成员的数目 亲属关系 以及某种遗传病或性状的分布资料,并按一定格式将资料绘制成的图解
先证者为该家族第一个被诊断患有某种遗传病的患者 或具有某种性状的成员
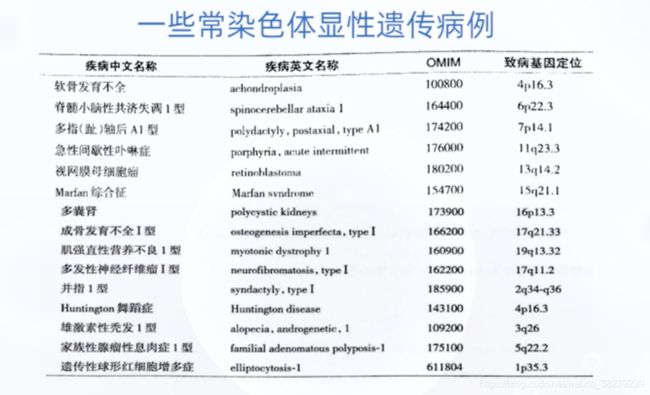
2.1. 常染色体显性遗传病 AD
遗传病致病基因位于1~22号常染色体上
常染色体显性遗传病可以有完全显性、不完全显性、共显性、延迟显性等
完全显性
杂合子表现型与显性纯合子完全相同时为完全显性
如 短指症A1型 基因定位 2q33-q35 患者基因型 Aa、AA
在线人类孟德尔遗传网址
常染色体完全显性遗传的特征
- 致病基因在常染色体上 与性别无关 男女发病均等
- 患者双亲之一必是本病患者 患者同胞1/2发病 双亲无病 子女一般不会发病
- 患者子代中有1/2发病可能
- 系谱中连续几代都可见患者 存在连续传递现象
婚配类型不同,子代发病风险不同
不完全显性遗传(半显性遗传)
杂合体Aa 表现型介于 纯合显性 AA 与 纯合隐性aa 之间

临床研究发现 味盲者 易患结节性甲状腺肿
软骨发育不全 AA纯合显性个体不能完成胚胎发育或早夭 存活下来都是 Aa
共显性遗传
在杂合子个体中 两种基因的作用都能表现出来
如 人类ABO血型 MN血型 组织相容性抗原的基因等
ABO血型
遗传受一组复等位基因控制(IA IB i 三种基因)定位与9q34

复等位基因
在群体中 一对特定的基因座上的基因有三种或三种以上成员 但对每个个体来说只能具有其中任何两个
延迟显性遗传
一些带有显性致病基因的杂合子 幼年时期致病基因A并不表达 或 表达不足 只有达到一定年龄后才表现出相应的临床症状
脊髓小脑共济失调I型
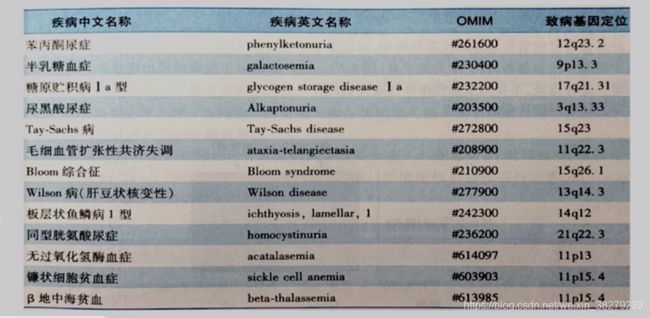
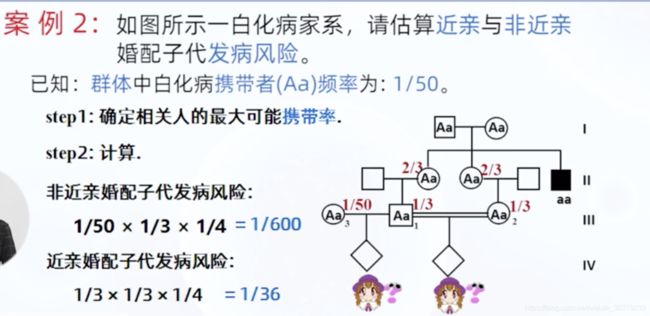
2.2 常染色体隐形遗传 AR
遗传病的致病基因位于常染色体上,遗传方式是隐形的,只有隐性致病基因纯合时才发病
携带者带有隐性致病基因的杂合子本身不发病 但可将隐性致病基因遗传给和后代 导致后代发病
眼皮肤白化病IA型
基因定位 11q14.3
代谢病 酪氨酸酶基因突变
其他常染色体隐性遗传病例
类显性遗传
隐性遗传中,杂合子与患者婚配造成子代有一半为患者。
家系中连续两代出现患者,子代比例模拟,类似显性遗传格局
常染色体隐性遗传特征
基因位于常染色体男女发病几率均等
患者双亲表型往往正常 但都是携带者出生患儿可能性为1/4。患儿正常同胞中有2/3可能性为携带者
系谱中患者散发,通常不连续传递
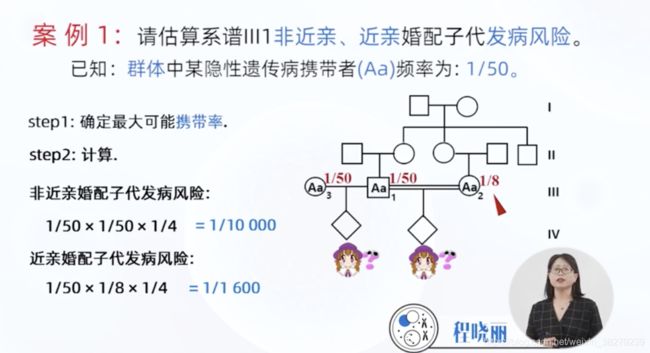
近亲婚配时 子女发病风险高
近亲婚配
3~4代之内有共同祖先的个体之间的婚配
亲缘系数
有共同祖先的两个人,在某一位点上具有同一基因的概率
![]()
2.3 X连锁显性遗传病 XD
致病基因是显性基因 且位于X染色体上,随X染色体连锁遗传
特点
女性XAXA 或 XAXa 基因型都将发病 纯合子患者极少见
女性为男性发病率2倍
通常男患者病情较重,女患者较轻
男性只有一条X染色体,其X染色体上的基因在Y染色体上缺乏等位基因。
因此男性只有成对基因中的一个成员,为半合子
交叉遗传
母亲的X染色体可传给儿子和女儿
父亲的X染色体只能传给女儿 不存在男男传递
实际病例
女性患者多于男性
患者双亲中 必有一方是本病患者
男性患者后代中 女儿都发病 儿子都正常
女性患者后代中 子女各有1/2可能发病
连续传递
2.4 X连锁隐性遗传病 XR
致病基因为隐性基因 位于X染色体
实际病例
XR遗传病的系谱特点
男患者多于女患者 系谱往往只有男患者
男患者双亲都无病 致病基因来自携带者母亲
交叉遗传 男患者同胞、外祖父、舅父、外甥、外孙等也可能是患者
如女性是患者,其父亲一定是患者,母亲一定是携带者或患者
男患者子女都正常 所以代与代之间可见明显不连续,隔代遗传
2.5 Y连锁遗传病
致病基因在Y染色体上,由男性向男性传递。
限雄遗传
Y染色体包含约78个编码蛋白质的基因
实际病例
2.6 影响单基因遗传病的因素
遗传异质性
一种遗传性状可由多个不同遗传改变所引起
基因座异质性
由不同基因座的基因突变所致

等位基因异质性
遗传病由同一基因座上的不同突变所引起的
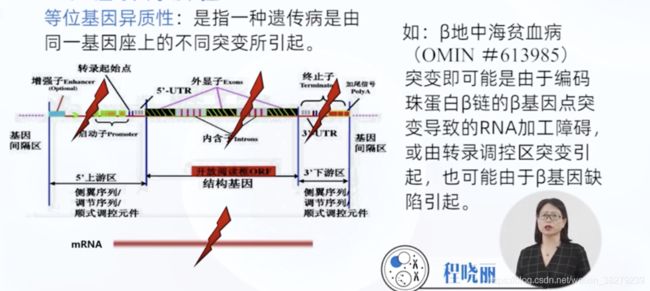
基因的多效性
外显率与表现度
外显率
指在一定环境条件下 群体中某一基因型个体表现出相应表型的百分率
100% 为完全外显
低于100% 为 不完全外显 或 外显不全
带有显性基因却未表现相应性状的个体为 钝挫型
此现象可导致系谱隔代遗传现象
表现度
在不同遗传背景和环境因素影响下 相同基因型的个体在性状或疾病的表现程度上产生的差异
“外显率”和“表现度”的根本区别
在于前者阐述了基因表达与否,是个“质”的问题;而后者要说明的是在表达前提下的表现程度如何,是个“量”的问题。
成骨发育不全综合症
临床表现不同
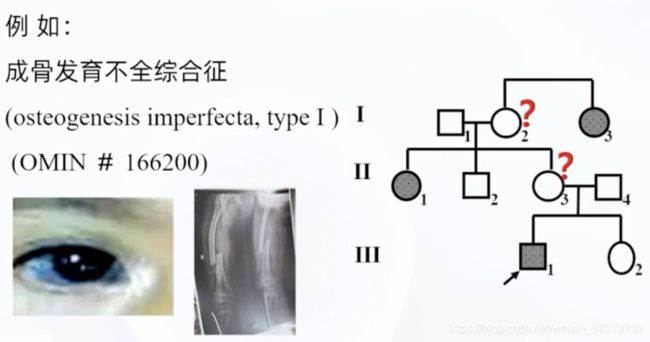
从性遗传
男女遗传都有临床表现 只是程度不同
患者发病年龄逐代提前 或 病情逐代加重
3. 多基因病的遗传
人类的一些性状或遗传病不是决定于一对主基因 而是由多对基因协同决定
这些基因对表型影响小,为微效基因
多对微效基因有累加效用, 共同作用形成明显表型为加性效应, 这些基因为加性基因。
多基因遗传除受微效基因作用外,还收环境因素的影响。因此也称为多因子遗传
3.1 质量性状与数量性状
质量性状
质量性状的分布是不连续的,存在着全或无的关系,没有中间的类型
数量性状
不同个体之间存在着程度或数量上的差别,没有质的分界线。如身高、体重、血压

示例
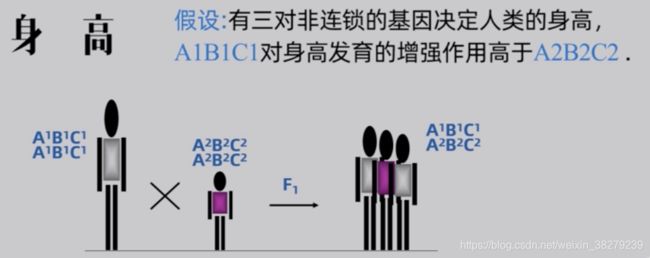
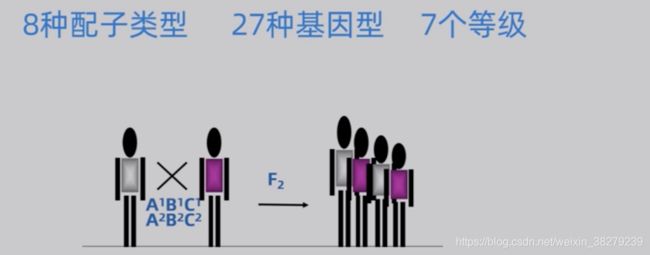
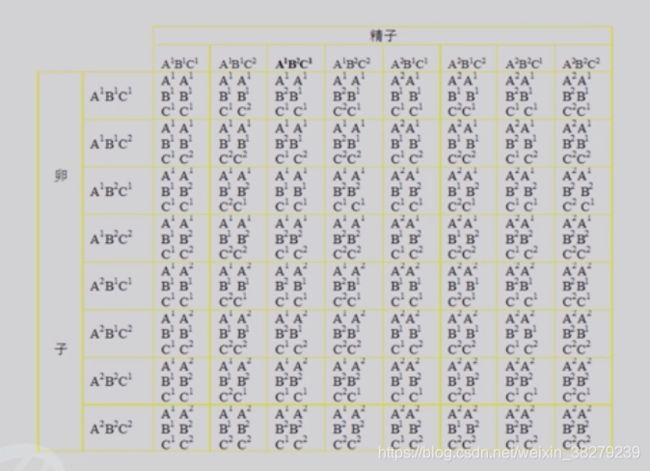
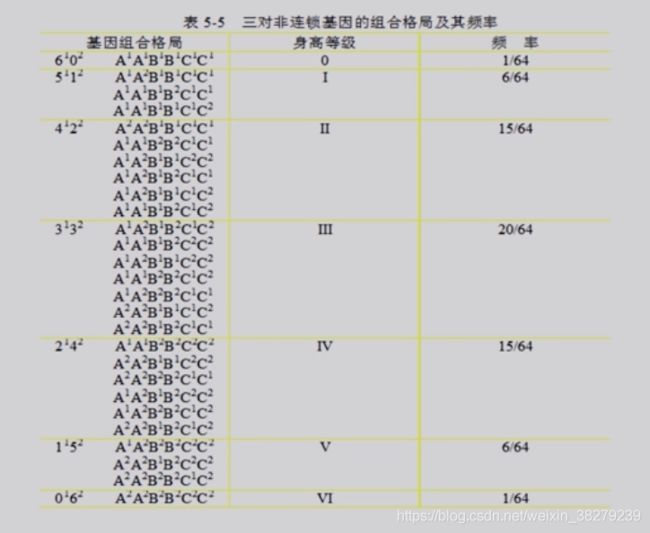
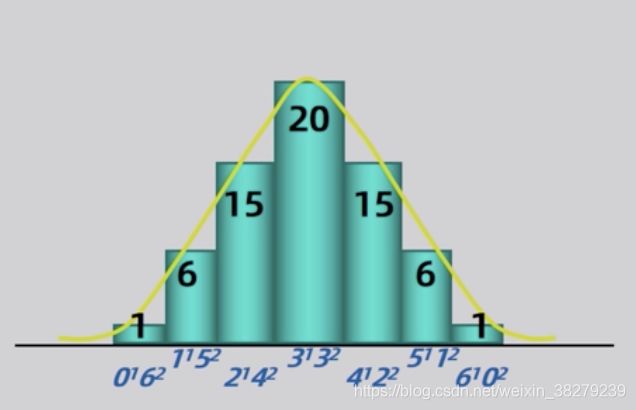
基因组组合型,呈正态分布

3.2 多基因遗传特点
两个极端变异纯合子的个体婚配,其子一代都是中间类型。但环境因素的影响会使其发生一定范围的变异
两个中间类型的子一代个体婚配所产生的子二代个体,大都属于中间类型的个体。但其变异范围比子一代更广泛
在一个随机婚配的群体中,变异范围十分广泛,但大多数个体仍接近中间类型。极端变异个体很少
多基因疾病的遗传
在评估多基因病因时 不仅要分析遗传因素 也要考虑环境因素
易患性与发病阈值
易感性
在多基因病中 由多基因基础决定的发生某多基因病的风险高低
仅强调疾病发生过程中遗传因素的影响
易患性
多基因发病中 遗传因素和环境因素共同作用 决定个体是否患病
比易感性较科学的评判标准
易患性越高 发病可能性越高
群体易患性 呈正态分布
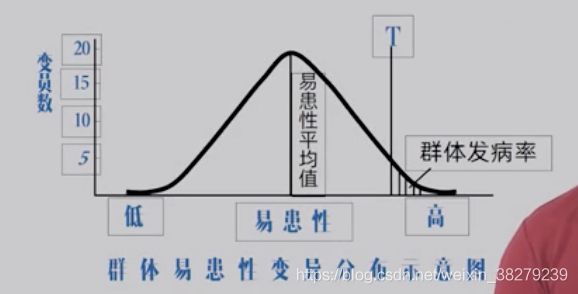
阈值 T
当个体易患性高达一定限度 即引发疾病 该限度为阈值
在一定环境条件下 阈值代表发病所必需的最低致病基因数量
阈值作用
将易患性连续变异 呈正态分布的群体分为 正常群体 和 患病群体 两部分

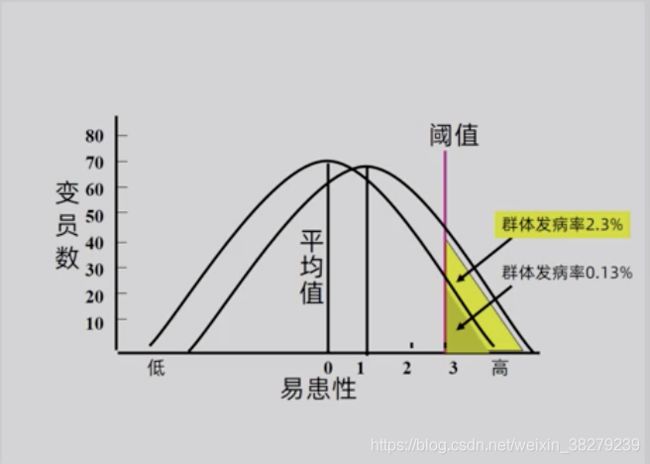
3.3 遗传度
表型是基因型和环境共同作用的结果
一个数量性状的变异包括遗传变异和环境变异
表型方差 = 遗传方差 + 环境方差
遗传度为遗传方差占表型方差的比值
遗传度的计算
通过一般群体与患者一级亲属发病率计算
通过患者一级亲属和对照组亲属发病率计算
通过双生子发病一致率计算
双生子发病一致率计算


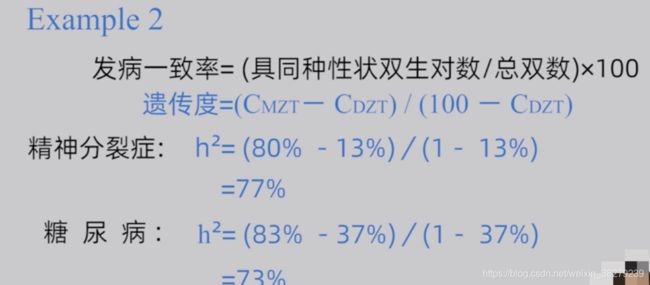
遗传度的意义
仅表明多基因遗传病中遗传因素的作用相对大小
是群体概念,对个人无意义
针对特定人群,特定环境,对其他人群和环境无意义
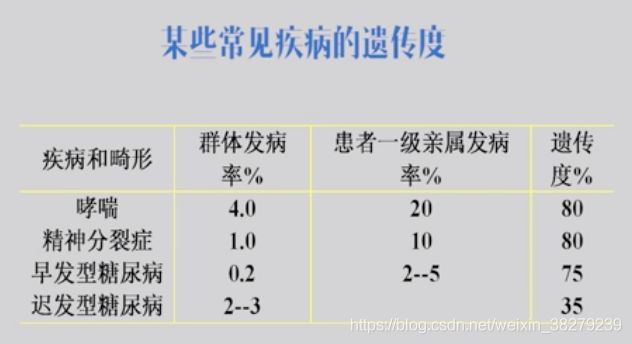
3.4 遗传多基因遗传病再发风险估计因素
患病率与亲属级别有关
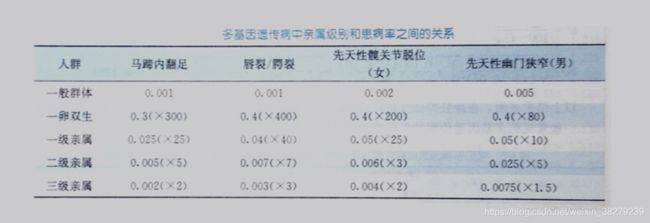
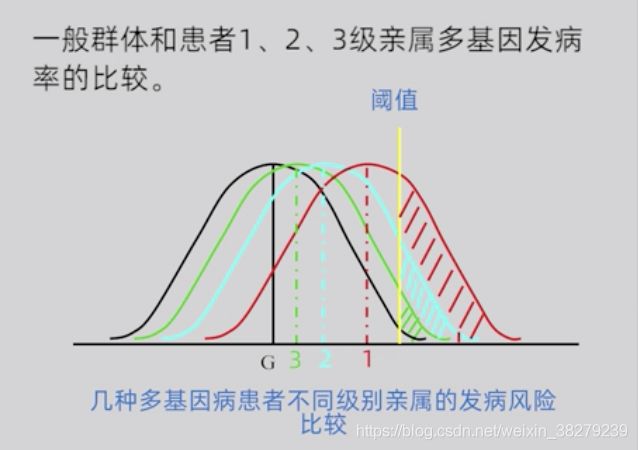
群体发病率与患者一级亲属发病率的关系
多基因病患者一级亲属的发病率与遗传度及群体发病率的大小相关
当群体发病率在0.1%-1%,遗传度在70%-80%时,患者一级亲属的发病风险f等于一般群体发病率P的平方根

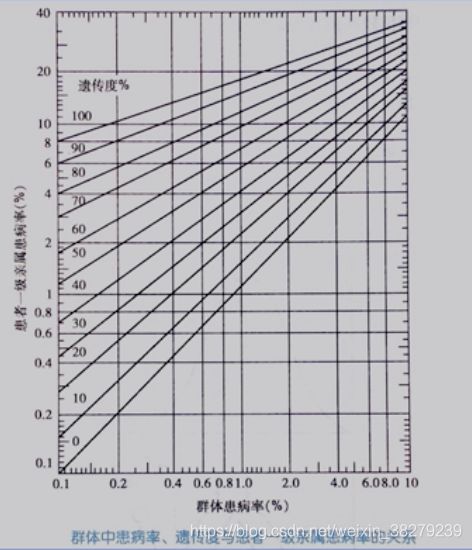
患者亲属再发风险与亲属中受累人数有关
家庭中发病人数越多 亲属发病风险越高

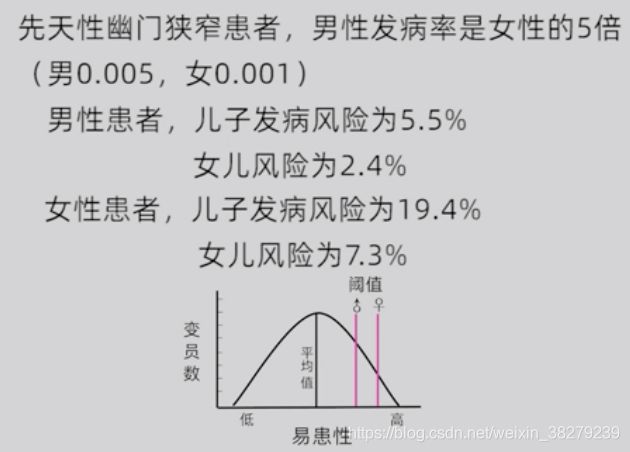
患者亲属再发风险与患者畸形或疾病严重程度有关

多基因遗传病的群体患病率存在性别差异时,亲属再发风险与性别有关
发病率低的性别后代发病风险相对较高,尤其是与其性别不同的后代再发风险更高
发病率高的性别 -> 子代发病率低
发病率低的性别 -> 子代发病率高
4. 人类染色体与染色体病
4.1 染色质与染色体
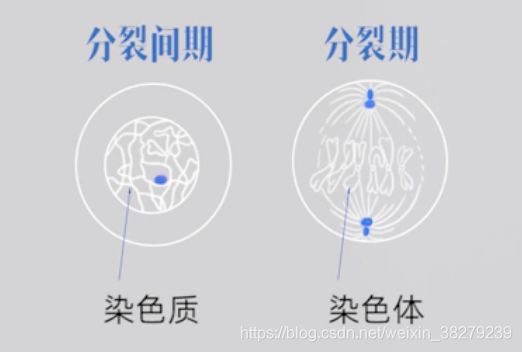
染色质为细丝状 。
当细胞分裂时,染色质复制反复盘绕高度压缩,凝集形成兴泰特定的条状染色体,以保证DNA能准确分配到两个子细胞中

染色质和染色体的化学组成
DNA和组蛋白占染色质化学总量98%以上

染色质的种类
染色质由于其折叠及压缩程度的不同,在形态、染色性能和功能上呈现出差异
可将染色质分为两大类 常染色质、异染色质

常染色质
折叠压缩程度较低,处于伸展状态
碱性染料染色时着色较浅
分散度较大,常位于细胞核中央
异染色质
折叠压缩程度较高,处于凝集状态
碱性染料染色师着色较深
主要分布于间期核的边缘
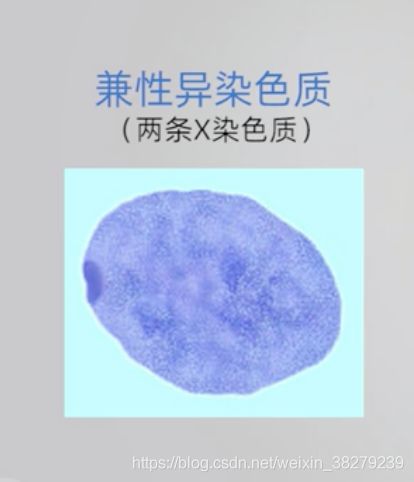
异染色质可分为结构异染色质和兼性异染色质二种类型
结构异染色质
在所有细胞类型的全部发育阶段均处于永久凝集状态
兼性异染色质
某些细胞类型,特殊的发育阶段呈凝集状态的异染色质

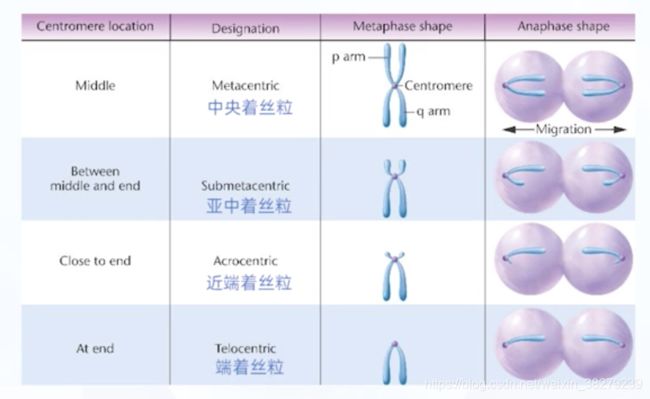
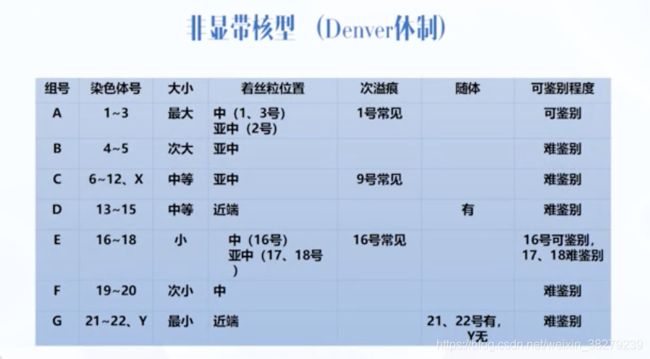
根据着丝粒在染色体上的位置 将染色体分为4种类型
人类染色体只有前三种类型 没有端着丝粒染色体
染色体核型与显带技术
核型
指一个体细胞中的全部中期染色体按大小、形态特征排列构成的图像
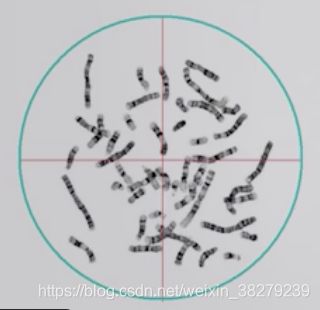
核型分析
将一个体细胞中的全部中期染色体按染色体的形态特点和大小依次配对,分组排列进行分析的过程
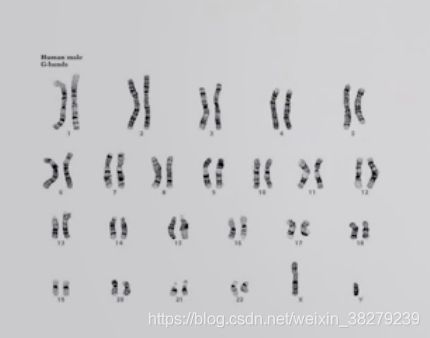
人类13、14、15、21、22染色体具有随体,其余染色体无随体
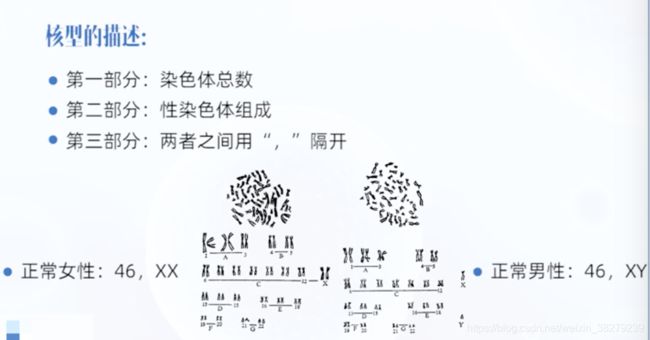
核型的描述
染色体显带技术

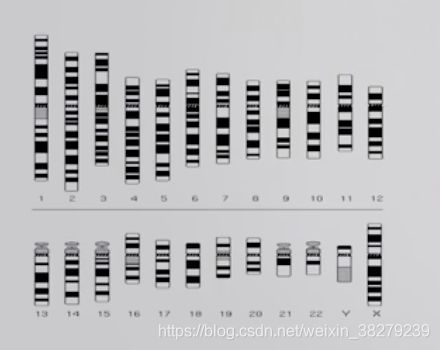

![]()
富含A-T碱基片段为亮带 C-G为暗带

最常用G显带
染色亮暗与Q带相反
标本易于保存,普通显微镜就可观察



染色体命名

Xq28
X染色体长臂第2区的,第8条带
1p31.32
1号染色体短臂第3区第1带第3亚带第2次亚带
性染色体
性别决定
XX 女孩
XY 男孩

Y染色体的有无是人类性别决定的关键因素

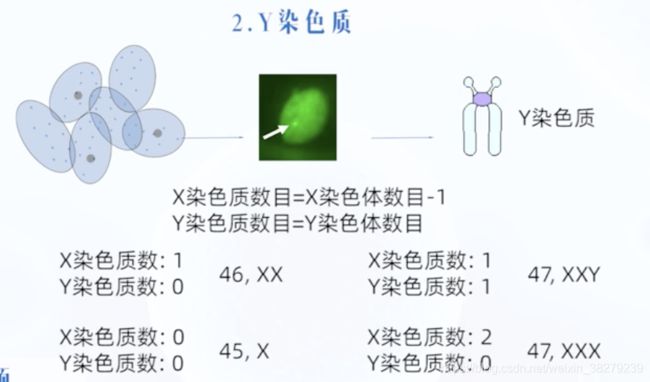
性染色质

X染色质

正常女性的X染色体,只有一条有转录活性
另一条无转录活性,螺旋化呈异固缩状态,形成X染色质
因此男女体细胞中,X染色体的基因产物是相等的
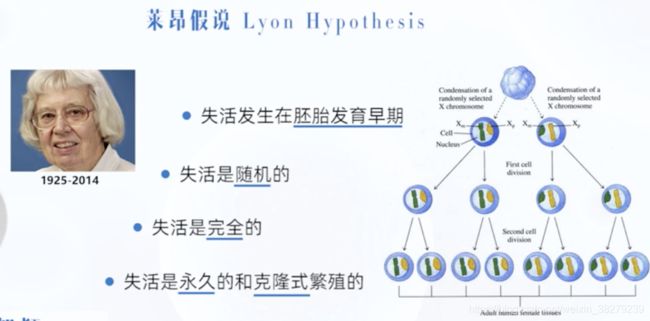
莱昂假说
正常女性的X染色体 只有一条有转录活性
其余都无转录活性 螺旋化呈异固缩状态 形成X染色质
4.2 染色体畸变

染色体畸变的诱因

染色体数目异常及产生机制
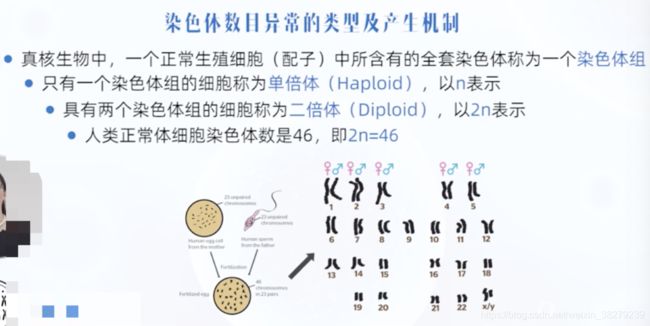

整倍体畸变

三倍体3n发生的机制
1.双雄受精
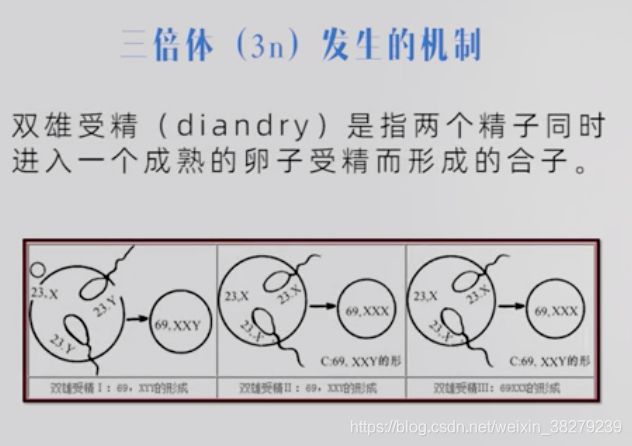
可形成三种类型的三倍体
69,XXX / 69,XXY / 69,XYY
2.双雌受精
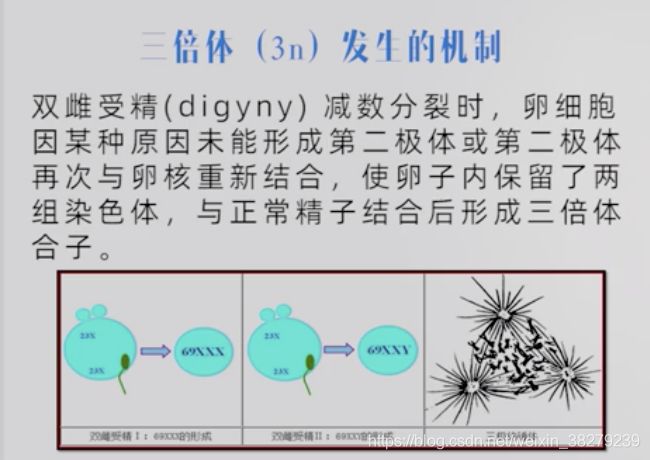
可形成二种类型的三倍体
69,XXX / 69,XXY
四倍体4n发生的机制
1.核内复制

2.细胞分裂时异常

非整倍体

如仅减少一条染色体 称为单体型
如仅增加一条染色体 称为三体型
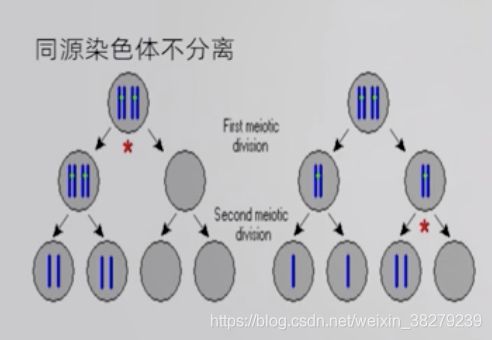
非整倍体的形成机制
1.减数分裂中染色体不分离

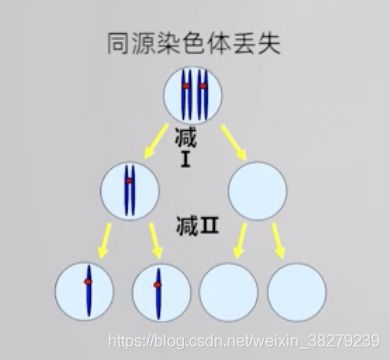
2.减数分裂中染色体丢失

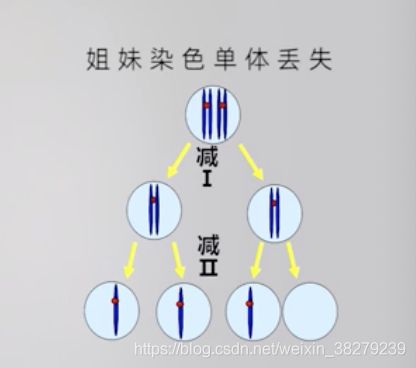
嵌合体
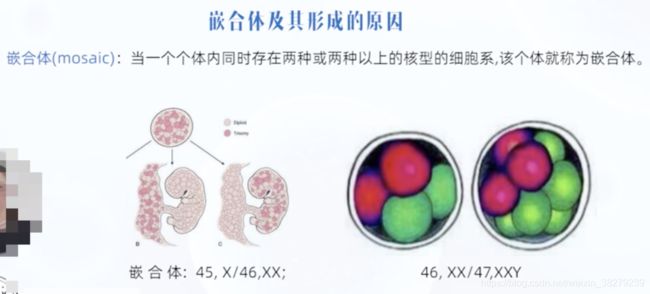
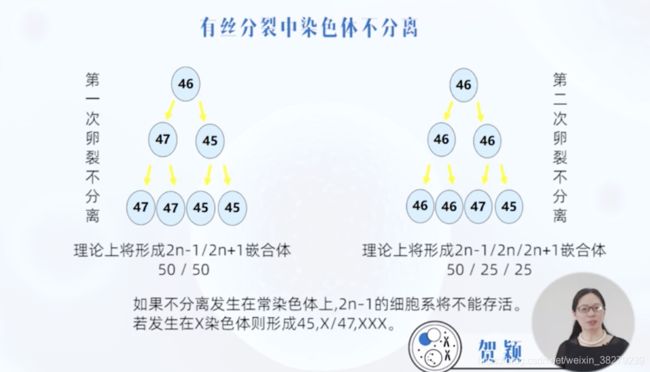
1.有丝分裂中染色体不分离
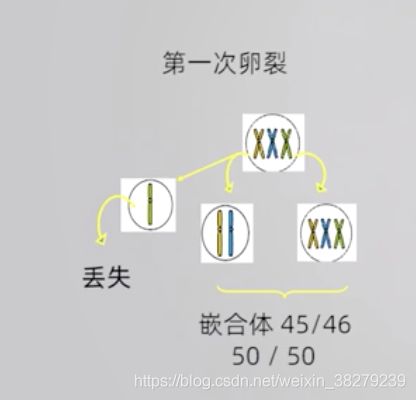
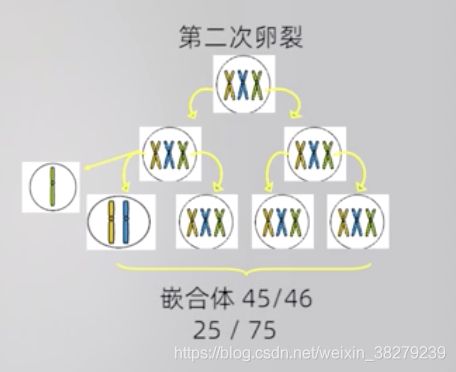

4.3 染色体结构畸变及产生机制
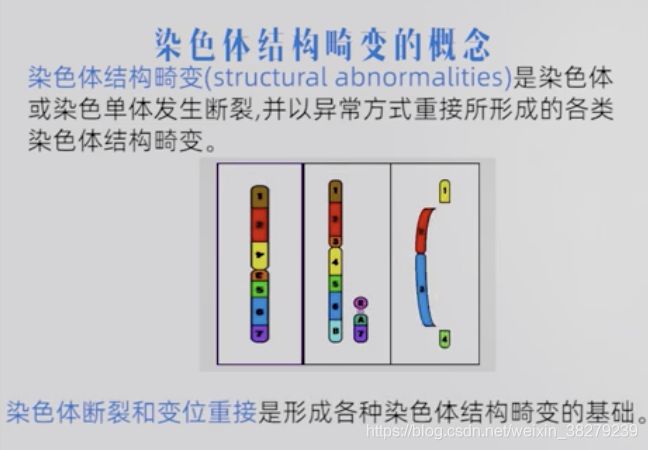

缺失 del
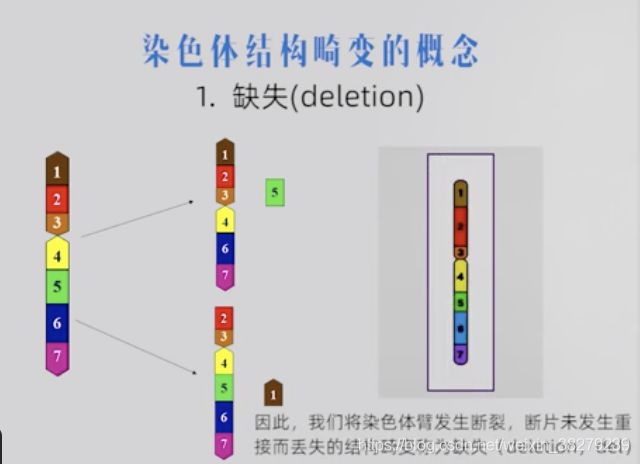
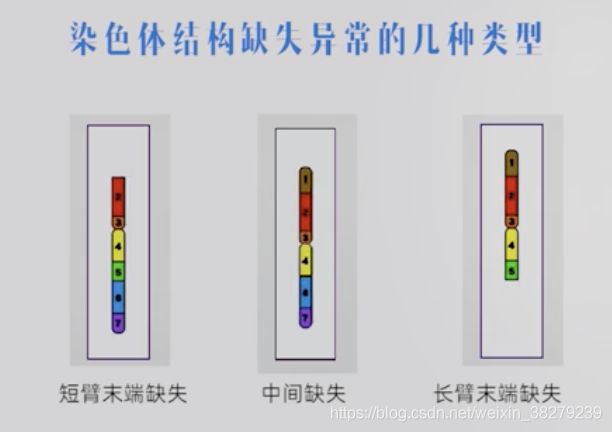
末端缺失,发生一次断裂
中间缺失,发生二次断裂
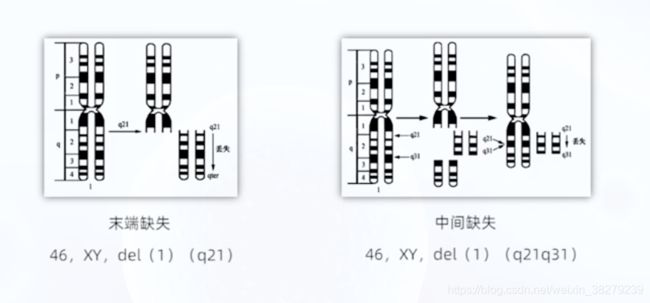
猫叫综合症 5p-综合症
5号染色体短臂缺失
倒位 inv

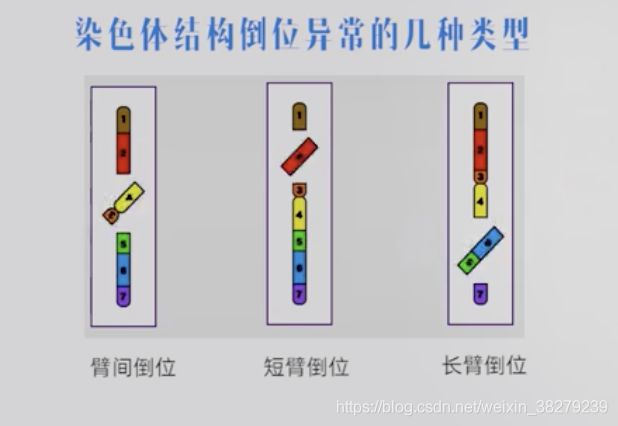
短臂倒位 和 长臂倒位 统称为 臂内倒位
可根据倒位片段是否包含着丝粒区分臂间倒位 和臂内倒位
倒位一般无遗传物质丢失 个体无明显异常表现 但对后代将产生不良影响
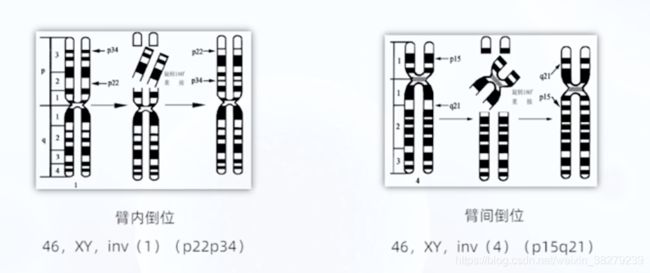
 该女性4号染色体发生臂间倒位
该女性4号染色体发生臂间倒位
将具有倒位染色体而表型正常的个体称为倒位携带者
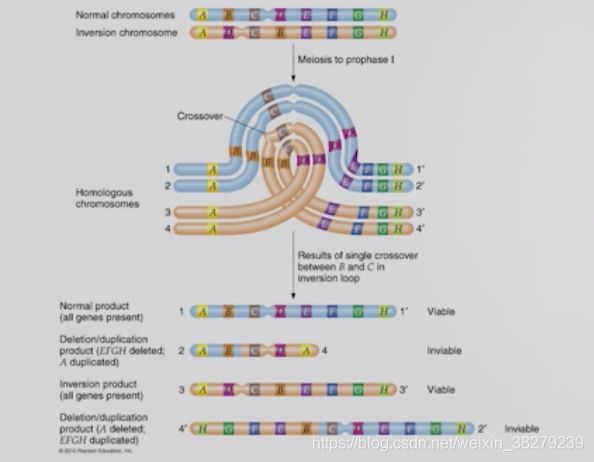
在形成生殖细胞的减数分裂过程中 因同源染色体只能在相同的部分紧密联会
即发生倒位的染色体只有形成其特有的倒位环,才能与其正常的同源染色体配对
着丝粒在倒位环内 经过倒位环内的一次交换
形成4种不同的生殖细胞:1种正常细胞 1种具有倒位 1种部分重复 1种部分缺失的染色体
后两种异常的生殖细胞受精后 受精卵或胚胎 致死致畸 发生流产或死胎
易位 t
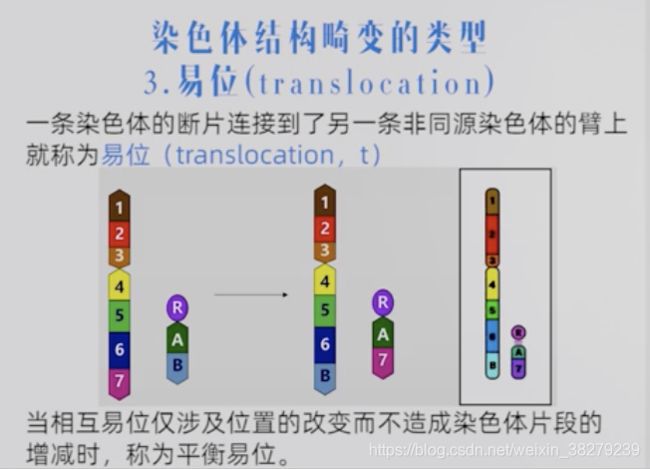
相互易位
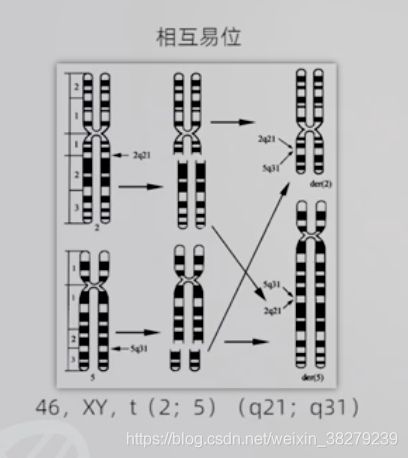
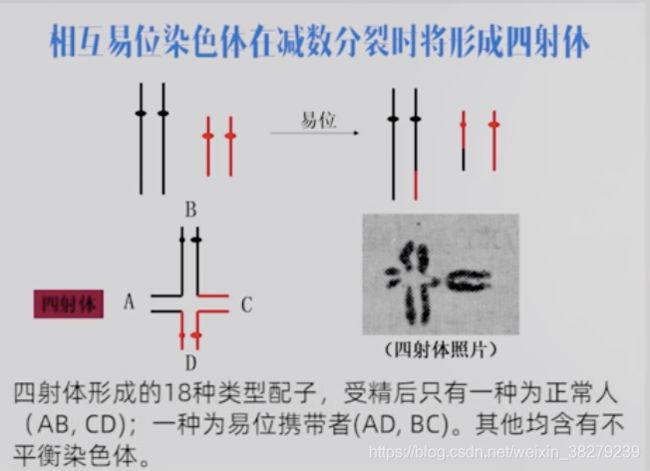

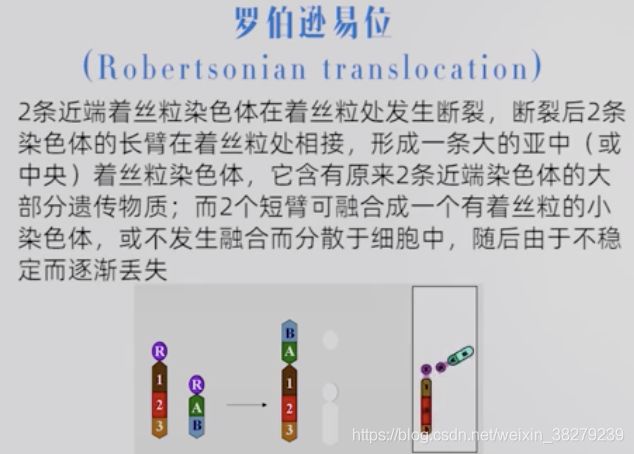
罗伯逊易位
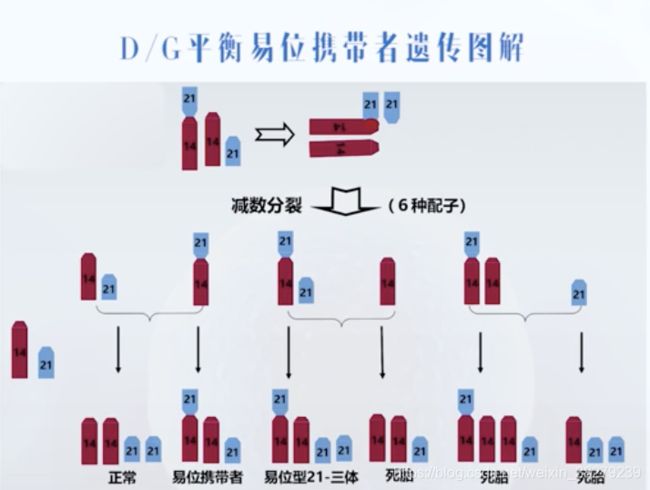
成活的胚胎有1/3可能性为21-三体
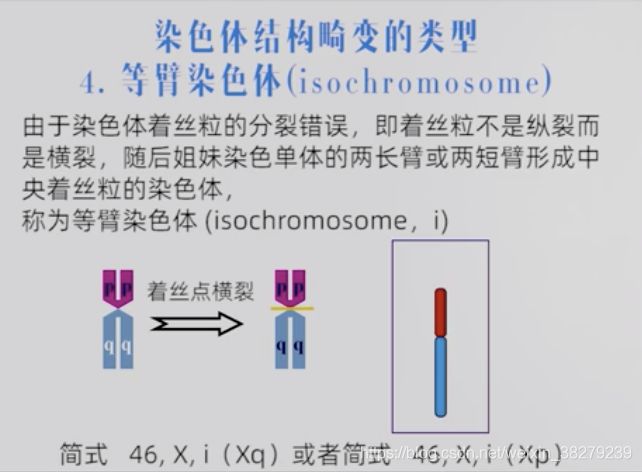
等臂染色体 i
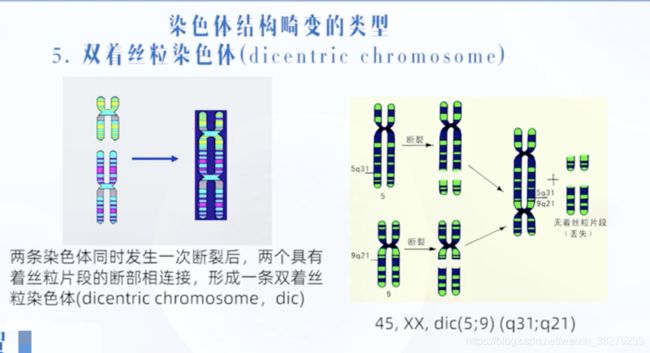
双着丝粒染色体 dic
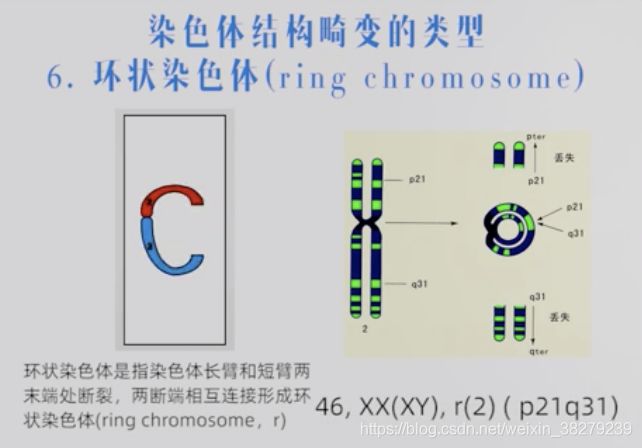
环状染色体
染色体病


外表正常的染色体易位携带者所携带的异常染色体 是造成子代流产 死胎 新生儿死亡 先天畸形的重要原因
4.4 常染色体病
DOWN综合症
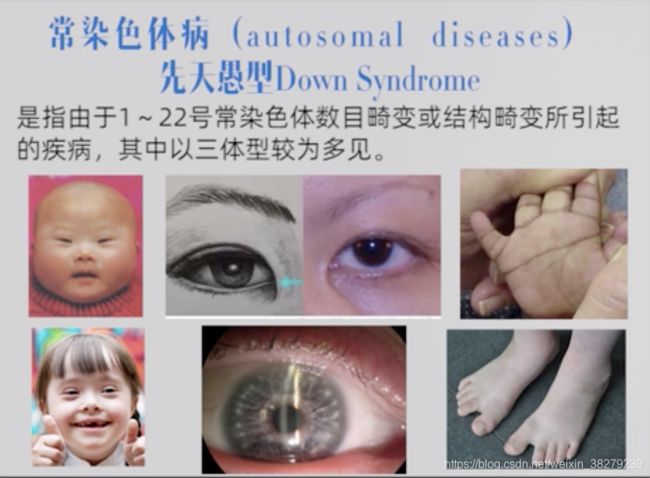
先天愚型根据核型不同可分3种类型
游离型(21三体型)、易位型和嵌合型
游离型
92.5%的先天愚型患者属游离型21三体
核型:47,XX(XY), +21
与母亲年龄有关 母亲年龄增高 发生率增高
易位型
5%的先天愚型患者属易位型21三体
30岁以下母亲所生患儿

染色体平衡易位携带者外表正常
常有流产或死胎
所生子女 1/3为正常 1/3易位携带者 1/3易位型先天愚型患儿
嵌合型
症状比游离型轻
正常受精卵在早期卵裂过程中发生21号染色体不分离
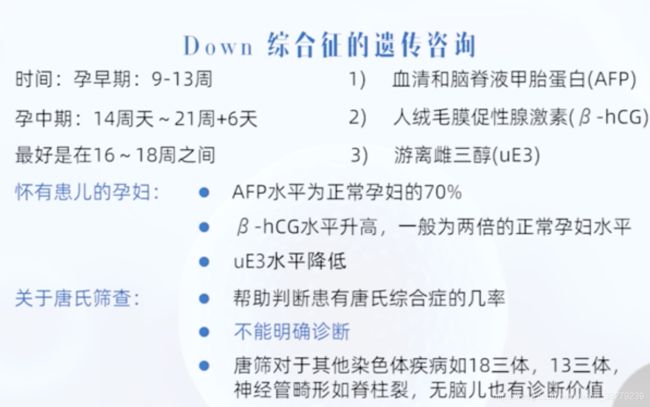
18三体综合症

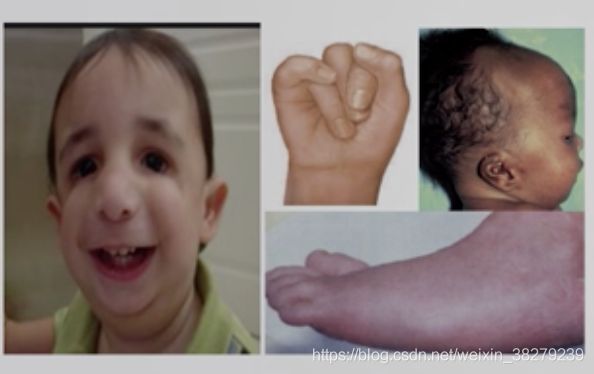
多数在出生后半年内死亡
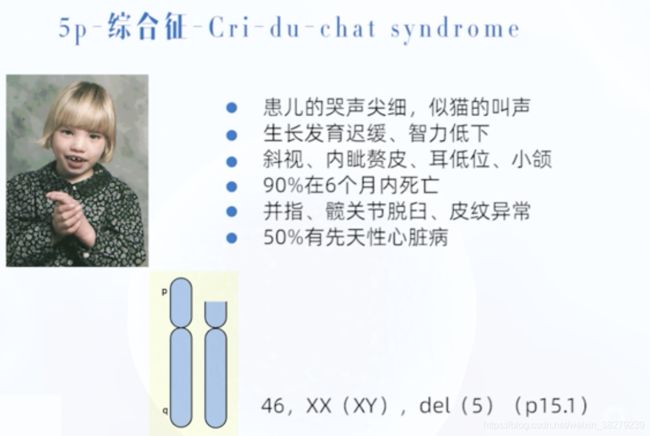
5p综合症
常染色体结构异常-缺失
发生率1/5000 女性多于男性
4.5 性染色体病
5. 线粒体病的遗传
5.1 线粒体
线粒体是真核细胞的能量代谢中心。细胞呼吸作用中的氧化还原反应是在线粒体中进行,并在此过程中产生大量的能量,供给整个机体利用。
线粒体是细胞质中独立的细胞器,也是动物细胞核外唯一的含有DNA(mtDNA) 的细胞器
线粒体内含有DNA分子,被称为人类第25号染色体,是细胞核以外含有遗传信息和表达系统的细胞器。其遗传特点表现为非孟德尔遗传方式,又称核外遗传
- mtDNA具有半自主性
mtDNA是一个长为16568bp的双链闭合环状分子。
外环含G较多,称重链(H链)。内环含C较多,称轻链(L链)
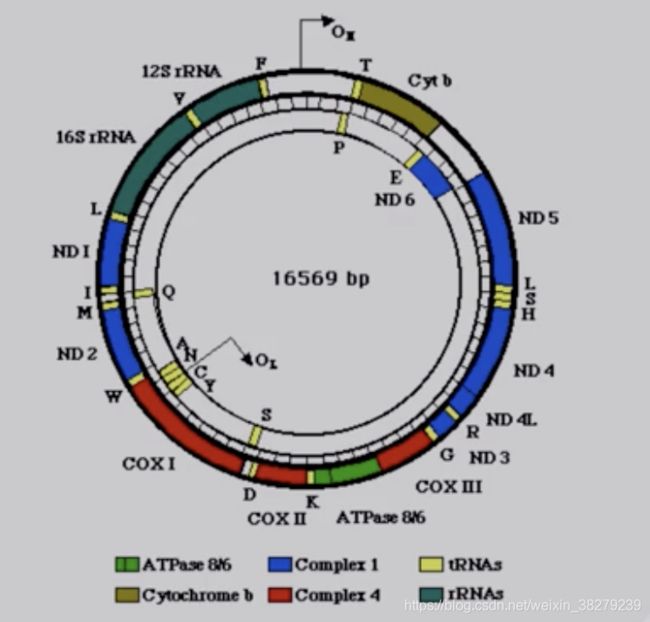
mtDNA结构紧凑,部分区域出现重叠,即功能重叠。
某个区域既是前一个基因的一部分,又是后一个基因的一部分。
无启动子和内含子,缺少终止密码子,仅以U或UA结尾
唯一的非编码区是D环区,是重要的调节区域
非编码区,由1122bp组成。包括mtDNA重链复制起始点,重轻链转录的启动子
人类线粒体编码基因37个
mtDNA编码13条多肽链,22种ZtRNA,2种rRNA,13种蛋白质均是呼吸链酶复合物的亚单位
线粒体的H链,12条多肽链,12S rRNA ,16S rRNA ,14中tRNA的转录模板
L链是1条多肽链,8种tRNA转录的模板
线粒体DNA是裸露的,缺乏组蛋白的保护,缺乏DNA损伤修复体系,所以线粒体DNA的突变率较高。
5.2 线粒体的遗传特征
mtDNA具有半自主性
线粒体虽具有完整的基因表达翻译体系 但其基因不足以支持完成完全的功能 所以mtDNA具有半自主性
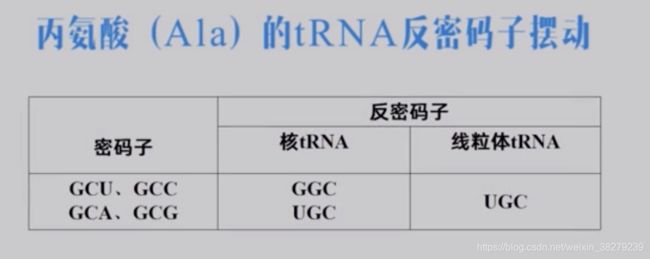
线粒体中,所有的tRNA由线粒体基因来编码,一共22种。而三联体密码子组成由64个,所以在线粒体中 反密码子对密码子的识别作用,特异性有所降低。
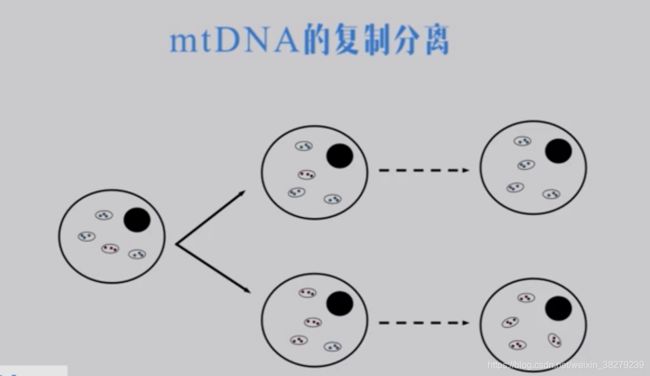
mtDNA在有丝分裂和减数分裂间都要经过复制分离
在发生复制时,线粒体进入子代细胞是随机进入的。如在子代细胞中,大多数都是野生型的线粒体DNA 。则该细胞为正常健康细胞,如大多为突变型线粒体进入,则细胞为不健康。
遗传瓶颈效应
卵子形成过程中,由含有几w个线粒体的卵母细胞发育成含几百个线粒体的卵细胞。
大多数的线粒体被丢弃掉,而丢弃过程也是随机的。
若保留的都是野生型线粒体,则是正常健康的个体。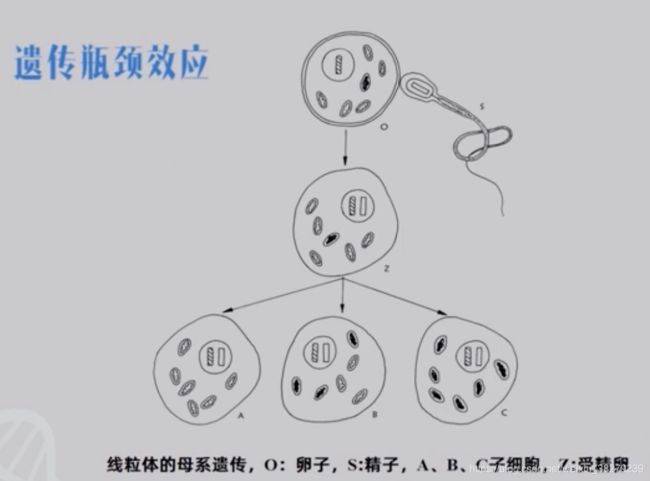
mtDNA的异质性与阈值效应
异质性
线粒体中存在野生型和突变型DNA

阈值效应
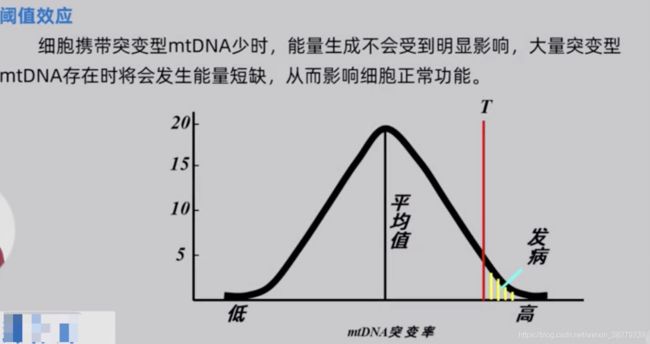
mtDNA的突变率和进化率极高
![]()
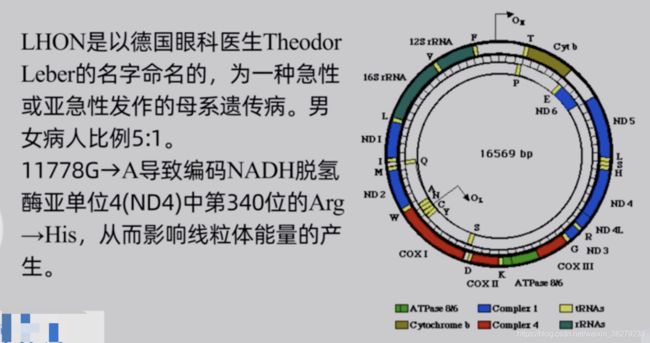
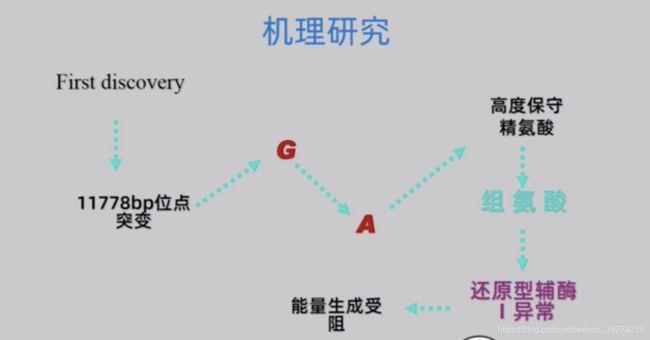
5.3 线粒体突变与疾病
碱基突变
错义突变
错义突变,引发氨基酸突变。
造成的疾病主要与脑脊髓性以及神经性疾病有关
常见的有Leber遗传性视神经病 和 神经肌病



蛋白质生物合成基因突变

大片段重组(缺失、插入突变)

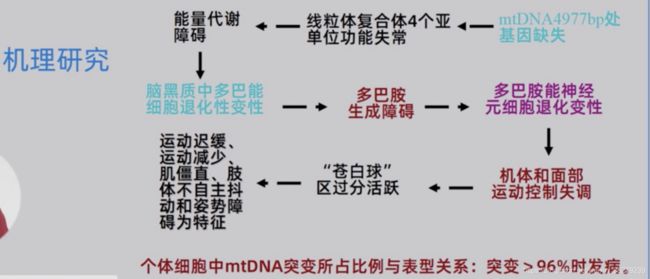
帕金森病
是在脑细胞中的线粒体发生了4977bp的缺失


6. 群体遗传学
群体遗传学 即以群体为对象来研究群体的遗传和变异的学科
6.1 概念
群体
指同一物种生活在某一地区内的,能相互杂交的个体群,也称为孟德尔式群体
遗传学将一个群体中所有个体含有的全部遗传信息(或基因)称为 基因库
医学群体遗传
主要探讨遗传病的发病频率 遗传方式及致病基因频率和变化的规律
基因频率
指某一基因在群体中出现的频率
也是某一种等位基因的占该位点上全部等位基因的比率

基因型频率
指群体中一对 / 一组等位基因组成的各种基因型个体的数量与该群体中个体总数的比率


当已知一个群体中某一特定座位的基因所组成的各种基因型频率时 基因频率可直接推算出来
 当证明一个群体中每个基因型的相对比例保持不变,即p2/2pq/q2的比例,那么这个群体可以说处于 Hardy-Weinberg平衡
当证明一个群体中每个基因型的相对比例保持不变,即p2/2pq/q2的比例,那么这个群体可以说处于 Hardy-Weinberg平衡
6.2 遗传平衡定律
Hardy-Weinberg平衡定律的应用
前提该群体处于 Hardy-Weinberg平衡时
最重要的应用就是通过某一性状(疾病)频率在群体中的分布情况,确定等位基因频率和杂合子频率,并以此为依据进行遗传咨询和制定遗传筛查项目
Hardy-Weinberg平衡的判定
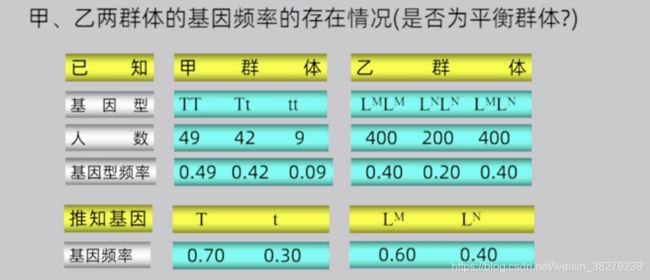
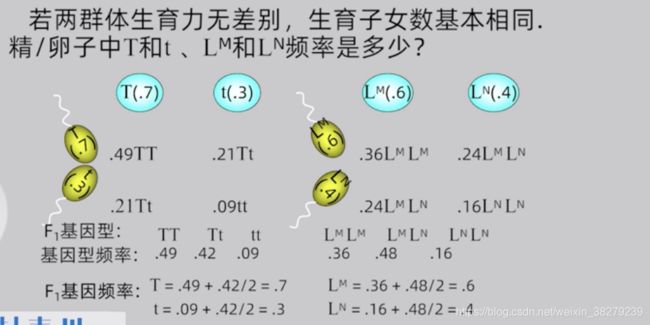
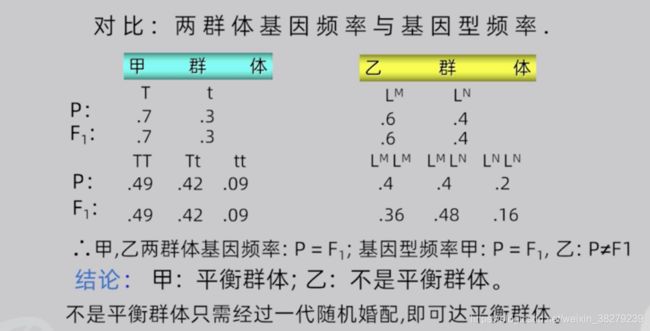
非遗传平衡群体只需要一代的随机婚配,就能够达到平衡。

Hardy-Weinberg平衡在遗传咨询中的用途
常染色体隐性遗传病基因频率计算


![]()
常染色体显性遗传病基因频率计算

X连锁遗传基因频率计算
女性有两条X染色体,基因频率和基因型频率的遗传行为和常染色体遗传相同
男性只有一条X染色体,X染色体上的致病基因只要有就会表现出来,基因型即为表现型,
基因频率= 基因型频率 
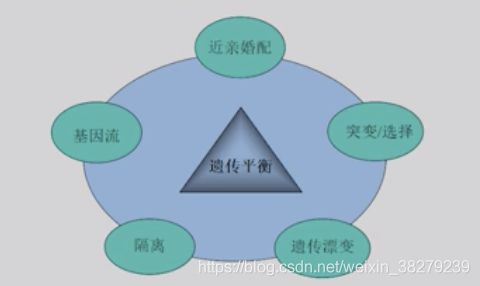
6.3 影响遗传平衡的因素
选型婚配
门当户对 郎才女貌 政治联姻 皇族联姻
如发生在AR患者中 将会增加纯合患者的相对频率
如 维多利亚女王 败血症
近亲婚配
表面上不改变等位基因频率,但可增加纯合子比例
由于不利的隐性表型面临被选择,因此最终改变后代等位基因频率 

选择
反映环境因素对特定表型或基因型的作用
正性选择或负性选择 对于特定缺陷往往为负性选择
适合度
指在一定环境条件下 某种基因型个体能生存并将其基因传给后代的能力
一般用相对生育率 即患者的生育率与他们正常同胞的生育率之比来衡量
S表示在选择作用下被降低了相对生育率
S = 1-f

 选择的作用不会使有害遗传病消失
选择的作用不会使有害遗传病消失
其他影响遗传平衡的因素
四、 泛基因组学
1. 单一“参考基因组”分析模式的局限
参考基因组
这里的“典型”泛指一个或者多个特定个体(这些个体是研究者所能获得样本,它们未必是最最具代表性的,这个要注意)。我们熟知的 “人类基因组计划(HGP)” 测序得到的基因组序列其实只是人类这个物种少量个体的基因组序列,我们将它称之为“人类基因组参考序列”,它由若干个高加索人的基因组序列合并而成。
基因组学的研究都需要围绕 “参考基因组” 来开展,因此它的作用和意义非凡。以人类基因组学研究为例,通常情况下我们要将新测序的人类基因组数据和参考基因组进行比对,才能够获得个体、人群之间所存在的序列差异,然后再进行后续研究,比如寻找疾病的遗传起源、肿瘤的易感因子、药物开发的靶点、特定目的基因芯片的设计等,参考序列就是这个过程中最重要的标准参照物——它是一个基因组学的“参考坐标系”。
但是,随着群体基因组研究规模的增大,这种单一参考基因组的模式就逐渐暴露出了它的不足之处。其中最重要的是,它无法包含(也不能表达)群体中所有个体的基因序列变化情况。因为,它的序列仅仅由一个或者多个特定个体的基因组信息合并而成,这些个体还不一定是种群中最具代表性的样本,所以它实际上是高度特异的,通常只能较好地代表一部分个体。因此,它不可避免地遗漏了很多在群体中存在的DNA序列,这在群体遗传学、特别是疾病基因组学上就会带来问题,比如可能由于完全丢失了和疾病相关的序列信息,导致无法对某类疾病进行完整的研究。
因此,学者们就提出了构建一个能够包含物种中所有 DNA 序列信息的理想基因组集合,这个“序列集合” 就称之为泛基因组(Pan-genome)
但泛基因组其实并不是要大家为每个不同地区的人群去组装一个所谓“特定地区人群参考序列”。我有时候会看到国内外有些团队和机构在干这个事,我觉得这完全不对(关于这一点我也欢迎大家发表自己的看法)。参考序列是一个参考坐标系,就像度量衡一样,它是需要统一的,它和测量长度的标准单位,测量质量标准单位之间并没有本质的差别,否则大家各用各的坐标体系,那么彼此的研究成果就说不到一块去了,结果与结果之间也无法共享,更无法高效复用现有的数据集和研究结论,就连基因你都得重新注释,完全就是一个事倍功半(可能连“半”都达不到)的活。
2. 泛基因组学”概念的由来和定义
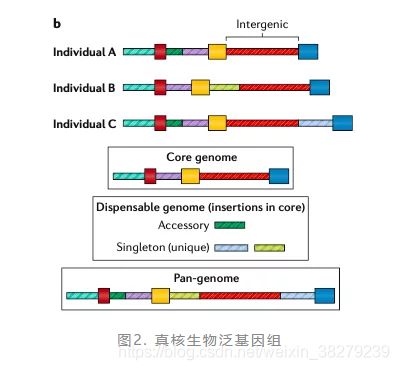
泛基因组(并不是人类泛基因组),这个概念最初是美国马里兰大学的 Tetterlin 教授,在 2005 年研究无乳链球菌的多种不同致病菌株时提出的。不过他当时的定义和现在已经有些差别了,他当时描述为,共享于所有菌株的 “核心基因组(core genome)” 和分散在单个或者部分菌株基因组中的 “非必需基因组(Dispensable genome)” 的总和,如图1。
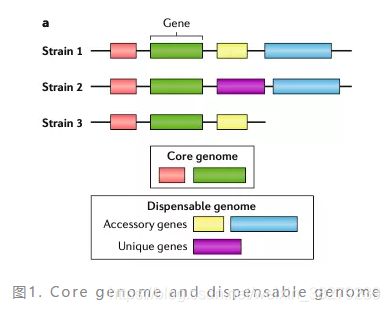
从他的定义中我们知道,泛基因组包含了两个部分,一个是共享于物种的公有序列(即,core genome)和分散在部分个体里的差异序列(即,dispensable genome)。构建泛基因组的关键是汇集第二部分的差异序列,这是物种中差异化最大、最为多样化的序列信息。
泛基因组在不同类型的物种里面,定义上还是有些区别的。
在 Tetterlin 刚提出这个概念时,他主要还是针对细菌这一类原核生物的。因此,那时研究人员所说的泛基因组,通常是指细菌的基因(gene)序列而不是全部的基因组(genome)序列。当时这样定义有其合理之处,因为,在原核生物中,绝大部分的 DNA 序列其实就是有功能的基因序列,而且不同菌株间基因的差异还比较大。在某些已经发现的细菌基因组中,菌株间基因的差异甚至可以达到20%-40%,而这些差异通常就和菌株它们在人群中的致病性、耐药性等密切相关。
真核生物泛基因组的定义和所包含的内容就与原核生物不同了,它不能仅围绕基因本身,因为在真核生物的基因组中,有很大一部分(超过50%)并不是编码基因的序列(外显子),而是基因间区(intergenic)、内含子(Intron)这一类。例如,在人类基因组中,基因编码序列仅占到了全基因组的 ~2%,如果仅用这些序列来描述人类的泛基因组那肯定是不对的。
因此,真核生物的泛基因组定义的是物种全体的 DNA序列总集合。如果我们非要将其中的编码序列拎出来造一个编码序列的泛基因组,那么可以叫:“基因泛基因组(genic pan-genome)”,图2 是关于真核生物泛基因组的示意图。
另外,除了泛基因组之外,最近还有一种新的泛分析,叫做:“泛转录组(Pan-stanscriptome)”。顾名思义,它是物种全体转录出来的 RNA 序列总集合,用来特指基因表达和转录上的泛分析。
这类分析在高等植物中比较常见,有两个方面的原因:第一,泛转录组的研究成本较低,研究起来也要更方便;第二,植物基因组通常都比较巨大而且序列复杂(通常都有高杂合、高重复的特点),以目前的技术来说,并不容易构建它们的泛基因组。目前已经拥有泛转录组的植物主要是一些重要的农作物,如玉米、小麦这一类。
3. 构建物种泛基因组的意义
对于细菌的泛基因组来说,就如上面提到的,细菌泛基因组有助于我们研究菌株内的遗传多样性,深入了解菌株致病性、毒力和耐药性差异的根本原因,而且还可以帮助我们预测新菌株的危害性。这对于人类健康来说有实际的意义。
对于真核生物的泛基因组特别是“植物泛基因组”研究来说意义也很大。现在已经有多个农作物的物种泛基因组被构建出来了,这其中包括水稻、西红柿、大豆、白菜、西兰花和向日葵等。
构建农作物泛基因组的目的通常都是为了了解作物基因组上的哪些突变会影响农作物的表型,从而改善育种。
尽管人类自8000年前开始驯化农作物以来,我们就已经对作物进行了多次选择性育种,但是我们通常并不了解所选定作物表型背后关联到的基因都是什么,有时甚至会留下具有不良表型的基因。比如,有些能够产出更大果实的品种可能缺乏抗病基因。通过泛基因组分析,可以用来揭示农作物表型和特定基因型的关联,反过来就可以帮助我们定向地进行作物育种和基因改良,然后创造出更高产、更具抗病性、更长保质期和口感更好的作物。
4. 人类泛基因组的构建
自 2015 年以来,大规模的人类基因组项目变多了(图3)。但迄今为止,也没有哪一个项目可以做到真正囊括全球各个不同的人类群体,并产生出一个包含全部人类群体变异序列并且可分析的泛基因组。
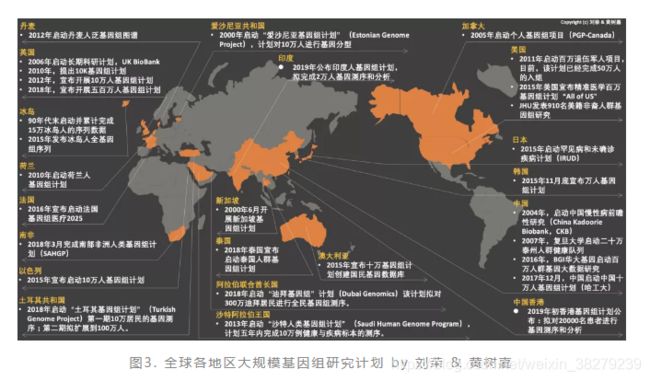
实现全人群的泛基因组,目前看起来还比较遥远,但针对特定地区或者国家的人群泛基因组(也就是Population-specific pan-genome)却已经有眉目了。这其中比较有代表性的是美国国家人类基因组研究所发起和资助的项目。而且,他们有一个项目计划收集350个具有不同遗传背景的个体构建全人群的泛基因组序列——意图实现全人群泛基因组。
构建人类泛基因组需要以人类基因组变异数据为基础。这些变异不仅要包括SNPs、短长度的Indels,也要包含基因组结构性变异(SVs)、拷贝数变异(CNV)等。
自人类基因组计划以来,我们代表性的变异数据集上已经有了一些进展,比如dbSNPs、ClinVar,以及孟德尔遗传病变异数据库(OMIM)。其中,ClinVar 和 OMIM 属于已知与临床性状相关联的变异集合。但这些变异集合都只是一些小长度的变异,对于基因组上长度超过 50bp 的高质量结构性变异来说,数量还很有限,这其中 dbVar、DGV和DGVa,已经算是人类基因组上一些比较大的结构性变异集合了——但其实都很小。
为了进一步扩大变异集合,国际上有多个人类基因组变异检测的项目在进行,这其中包括高深度千人基因组计划、TOPMed、Simons基因组多样性计划以及 gnomAD。特别是 gnomAD,今年成果很显著,6月份的时候以专刊的形式在Nature期刊上发了4篇相关的文章,其中也包括结构性变异的成果。
不过,在以上项目中,千人基因组项目无疑是最早开展的大型全球性人类基因组测序计划,它分为三期,从2009年开始,一共测序了世界上26个不同的群体、2504个人的基因组,它所产出的人类基因组变异数据库也是目前最成熟和用的最广的一个。但千人基因组项目发起时使用的是低深度测序技术,这导致在基因组变异检测方面存在局限性——特别是难以检测结构性变异。因此在2019年的时候,千人基因组启动了一个高深度重测序计划,目的就是为了弥补前期的局限并提升现有变异检测的质量,不过目前尚未有关于这一块的新进展。
除了要获得基因组完整的结构性变异之外,构建人类泛基因组还面临着另一个挑战,那就是Novel sequence 的检测。Novel sequence与前面的序列变异不同,序列变异指的都是那些和参考基因组相比存在差别的序列信息,大多数情况下这些序列还是可以通过比对,然后在参考基因组上找到信号的。但 Novel sequence 是参考基因组上完全丢失的序列内容,它不在参考基因组上,因此这是无法通过直接比对获得的,而是要进行高深度测序和序列组装(或者采用单分子测序技术获得长序列)才能发现。
Simons基因组多样性项目是寻找人类 Novel sequence 方面的一个代表。它收了全球各地 142 个不同群体、一共300个样本的基因组数据,然后高深度(30x-40x)测序,并进行序列组装,最后发现平均每个个体有约 5.2M 的新序列(Novel sequence)无法比对到人类参考基因组上。
除了 Simons 项目之外,TOPMed 项目也在这方面有所发现。TOPMed 很大!有 53,831 个样本,使用的测序方法也是短序列测序方法,但它平均每个样本只发现了2.2M 的 Novel sequence,之所以比 Simons 项目少,可能是因为这个项目的质控条件比较严格。
另外,虽然说现在全球大型人类基因组项目也不少,但用这些项目来构建泛基因组帮助是有的,但也都有一些很明显的局限性,原因就在于:
首先,它们基本上都是短读长测序数据,这本身就不利于基因组完整变异的检测。而且短序列也并不利于组装,常常会由于读长不足的关系丢失一部分基因组上的大长度插入序列。还有就是这些项目的首要目标其实并不是为了 Novel sequence 和泛基因组,泛基因组分析通常都只是这些项目一个次要的、补充性的边角料分析。最后,这些项目即使发现了相关的变异信息和 Novel sequence,也没有一个统一的、标准化的方式来将这些新变异、新序列进行整合、或者纳入到一个可被使用的所谓”人类泛基因组序列集合“中。
另外,我们目前甚至也没有人类染色体从端至端的完整序列拼接结果,这也会导致我们难以分辨一些 DNA 序列到底应该落在哪。不同的人群基因组项目对的 Novel sequence 数量和长度也存在着差异,有时候差异还十分巨大。比如 1.5万冰岛人的基因组项目和910个非洲裔基因组项目所获得的 Novel sequence 分别是0.33Mb和295.5Mb。
非洲裔的 Novel sequence 比起冰岛人的要多得多,这很可能是由于人类参考基因组是由欧洲人所构成的有关。现代欧洲人其实只是非洲人中的一个子集,在走出非洲之后逐渐演变而来,这个子集自然无法包含更多属于非洲裔的特有序列。
如果要比较有效地解决结构性变异和Novel sequence的检测问题,我们还是得通过长读长测序技术和序列组装技术。尽可能将每个个体的基因组(甚至单倍体基因组)组装出来,这样得到的差异序列才能更好地用于泛基因组序列的构建。
5. 如何记录和表示泛基因组序列
泛基因组序列有两个部分:核心基因组和分散在个别群体中的DNA序列,如何将它们有效地记录起来,并用在实际项目中,目前来看有三种可能的方式。
- 线性记录
第一个是线性记录,就是将这些额外的序列直接作为 Alternate 序列收录在原来的人类参考基因组之中,如同另一条染色体序列一般,GRCh38 参考基因组中大量的 ALT 序列就是这一类。使用的时候,它们相当于被看成是另外的染色体序列。这样的表示方式好处是简单,但是却丢失了一些属于这些新序列的信息,比如它们原本应该是在某一条染色体上的特定位置的信息。
- 图表示
第二个方式,称之为图表示(graphical representation)。这是一个在逻辑上更加合适的表达方式,如图4 所示。
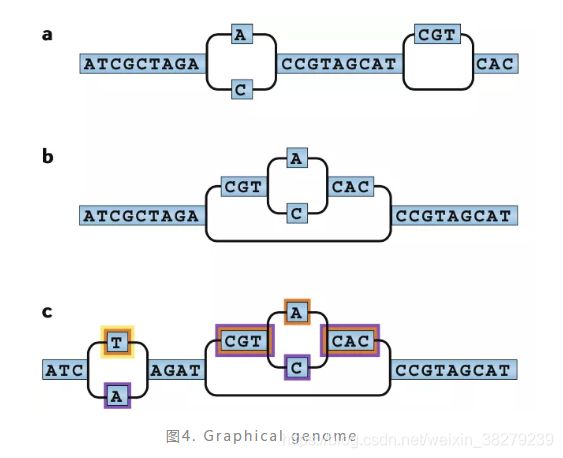
它以原来的参考基因组为框架,以一个个的碱基作为图的节点,碱基之间的前后关系作为图的边,然后在存在序列差异的地方自然形成不同的分支,从而逐渐构建出一个图基因组。这个图基因组可以依据新序列的加入不断扩展变化,最终它将会成为一个符合全人群的泛基因组图谱。这和上面的线性基因组是不同的,图基因组是一个二维序列图谱。
但要使用这样的一个图基因组来进行分析,就无法沿用当前适合于线性参考基因组的算法了,而是必须开发一系列全新的生物信息学方法。目前适合用在图基因组方面的算法有VG、SevenBridges、aSGAL和GraphAligner。
图基因组虽然逻辑合理,表示方式也很形象,但遗憾的是缺点也很多。首先,它很容易产生更多的歧义比对结果(如下图5)。
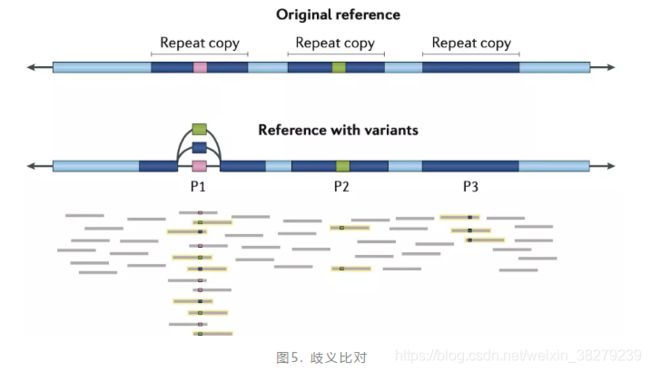
在图5中,本来测序 read 只会比对在一个地方的,但现在却会同时比对到图中三个不同的分支序列上,而且由于发生差异的情况都是相同的,这就导致这个 read 在这三个位置上看起来都是合理的,从而形成了歧义,这样的歧义结果会明显增加变异检测的难度——而且难以排除。
另外,序列错误比对情况增多,这随着而来的就是更多假阳性变异结果的检出。
以上这两个问题是应用图基因组来做研究的明显挑战。至于是否还有其它方面的挑战也欢迎大家在留言区一起来讨论。
- 混合记录
第三个方法是混合记录。将人类泛基因组同时以图基因组和线性基因组的方式进行存储,如图6所示。
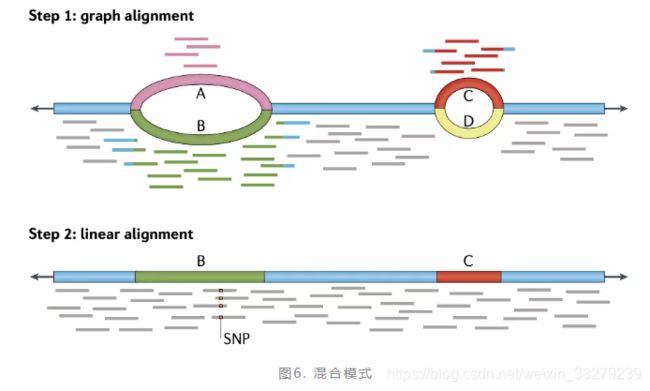
这样可以同时发挥这两个方式的优点,目的其实是为了减少由图基因组所带来的歧义情况。不过这在进行数据分析的时候就需要分两个步骤了,要分别对图基因组和线性基因组进行比对,然后再结合这两个比对结果完成基因组上的序列差异分析。
五、 生信数据格式
了解不同的生信数据格式,对日常科研、分析数据是非常有必要的。
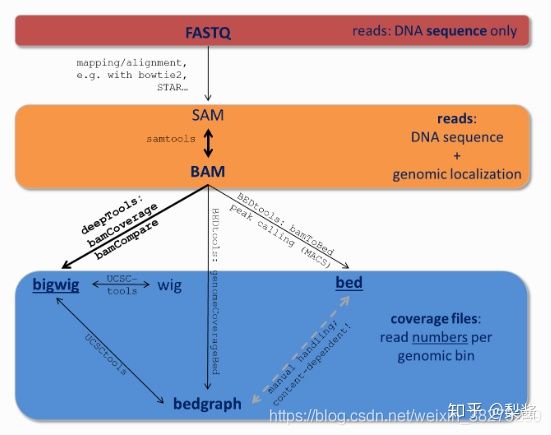
不同的软件,针对某方向的某生物学问题,都会创建或使用一种或者多种格式标准。比如储存数据常用sra、比对数据常用bam,基因组注释信息常用bam或gff。
先介绍下FASTA和FASTQ。
因为要便于存储生物序列信息,比如ACGT组成的DNA序列、蛋白质序列,生物信息学家们便基于 txt 文本格式定义了有一定规范的FASTA和FASTQ格式。
1. FASTA格式
FASTA文件格式,来自一款古老的名为“FASTA”的比对软件。
该软件1988年问世,发表于PNAS。
之后,生物学家们就沿用这个软件名作为后缀:.fasta,.fa,或其压缩格式.fasta.gz,.fa.gz。
以下为一个包含单个序列的FASTA文件示例:
>gi|31563518|ref|NP_852610.1| microtubule-associated proteins 1A/1B light chain 3A isoform b [Homo sapiens]
MKMRFFSSPCGKAAVDPADRCKEVQQIRDQHPSKIPVIIERYKGEKQLPVLDKTKFLVPDHVNMSELVKI
IRRRLQLNPTQAFFLLVNQHSMVSVSTPIADIYEQEKDEDGFLYMVYASQETFGFIRENE
第一行为“>”开头的标题行,包含序列的名称和/或唯一标识符等信息。
在标题行和注释之后,则是由一行或多行构成的蛋白质或核酸序列数据,其中每行的长度应短于80字符。
2. FASTQ格式
有了FASTA,为什么还要有FASTQ呢?
因为要把测序数据和其质量得分放在一个文件里储存,方便。
FASTQ格式是一种保存生物序列(通常为核酸序列)及其测序质量得分信息的文本格式。序列与质量得分皆由单个ASCII字符表示。
该格式最初由维尔康姆基金会桑格研究所开发,旨在将FASTA格式序列及其质量数据集成在一起。而当前,FASTQ格式已经成为了保存高通量测序结果的事实标准。
质量得分:
一个碱基的错误概率的对数值。最早在Phred软件中被定义和使用,后来逐渐推广开来。
以下为一个包含单个序列的FASTQ文件示例:
@SRR001339.3 FC12160_04JAN08_s_3.tar:3:1:230:474 length=36
GTTAGTCGGGAACTAAGGCCTGTAGGCTCTTTCCAT
+SRR001339.3 FC12160_04JAN08_s_3.tar:3:1:230:474 length=36
IIIIBIII*II,III$I′I9IDI%II … 5′E%%(H
可以看到,在FASTQ文件中,一个序列通常由四行组成:
- 第一行以@开头,之后为序列的标识符以及描述信息(与FASTA格式的描述行类似)
- 第二行为序列信息
- 第三行以+开头,之后可以再次加上序列的标识及描述信息(可选)
- 第四行为质量得分信息,与第二行的序列相对应,长度必须与第二行相同
3. BED格式
BED (Browser Extensible Data)是一种灵活的储存数据的格式,主要用来储存基因组特征(genomic features)或注释信息。
3.1 BED格式文件的组成
必选信息(3行)
- chrom - 染色体名称
e.g. chr3, chrY, chr2_random 或 scaffold e.g. scaffold10671.
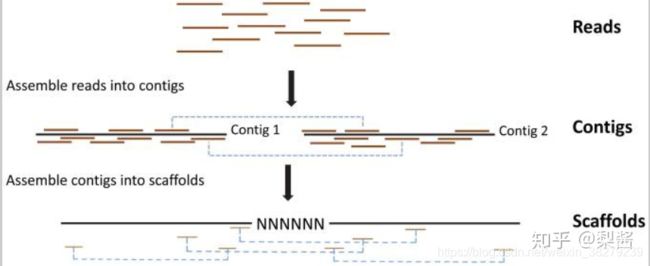
补充下什么是scaffold?
二代基因组测序的原始结果为reads,reads可以拼接为序列contig,contig进一步可以组装成更长的序列scaffold。
- chromStart - 染色体或scaffold起始位点
注意!是从0开始计数!是从0开始计数!是从0开始计数! - chromEnd - 染色体或scaffold终止位点
注意!结束位点的碱基不包含在内。比如chromStart=0, chromEnd=100, 那么实际上表示为0-99.
以下为最简单的BED3文件示例:
(有几列就叫BED几,下面数据有3列,则是BED3)
chrom chromStart chromEnd
chr1 213941196 213942363
chr1 213942363 213943530
chr1 213943530 213944697
chr2 158364697 158365864
chr2 158365864 158367031
chr3 127477031 127478198
chr3 127478198 127479365
chr3 127479365 127480532
chr3 127480532 127481699
备选信息(9行)
- name - BED的名称.
- score - 分数为0 到1000。颜色越深,分数越高。
如图所示

- strand - 链方向.
“.” (=no strand) 或"+" 或 “-”. - thickStart - 绘制特征的起始位置(例如起始密码子)。
当没有thick部分时,thickStart和thickEnd通常设置为chromStart位置。 - thickEnd - 绘制特征的终止位置(例如终止密码子)。
- itemRgb - RGB值
e.g. 255,0,0
如果track 行 itemRgb 参数设置为 “On”, RBG值会决定BED文件中该行数据的颜色. - blockCount - blocks (exons)在BED line的数量.
- blockSizes - 用逗号分隔的列表,用来表示block的的大小。
列表中数字的数目应和blockCount 一致。 - blockStarts - 用逗号分隔的列表,用来表示block的起始。
所有blockStart 的位点应该和chromStart 相关。 列表中数字的数目应和blockCount 一致。
在有block等相关列的BED文件中,blockStart 值必须是 0, 因此第一个block是从chromStart 开始。同样的,最后一个blockStart 位点加上最后一个blockSize 值应该和chromEnd相等。Blocks不能重叠。
以下为BED9文件示例:
track name="ItemRGBDemo" description="Item RGB demonstration" itemRgb="On"
chr7 127471196 127472363 Pos1 0 + 127471196 127472363 255,0,0
chr7 127472363 127473530 Pos2 0 + 127472363 127473530 255,0,0
chr7 127473530 127474697 Pos3 0 + 127473530 127474697 255,0,0
chr7 127474697 127475864 Pos4 0 + 127474697 127475864 255,0,0
chr7 127475864 127477031 Neg1 0 - 127475864 127477031 0,0,255
chr7 127477031 127478198 Neg2 0 - 127477031 127478198 0,0,255
chr7 127478198 127479365 Neg3 0 - 127478198 127479365 0,0,255
chr7 127479365 127480532 Pos5 0 + 127479365 127480532 255,0,0
chr7 127480532 127481699 Neg4 0 - 127480532 127481699 0,0,255
3.2 BED文件的处理
bedtools,可对储存了基因组特征信息的BED文件进行比较、注释、转化等操作。
- 安装bedtools
curl http://bedtools.googlecode.com/files/BEDTools.<version>.tar.gz > BEDTools.tar.gz
tar -zxvf BEDTools.tar.gz
cd BEDTools
make
sudo cp bin/* /usr/local/bin/
#也可以使用conda
conda
- bedtools常见用法
#查看bedtools的说明文档
bedtools --help
#从两个BED文件中得到genome feature的交集
bedtools intersect -a reads.bed/gff/bam/vcf -b genes.bed/gff/bam/vcf
#-wa参数可以报告出原始的在A文件中的feature
#-wb参数可以报告出原始的在B文件中的feature
#-c参数可以报告出两个文件中的overlap的feature的数量
#-wo 返回overlap碱基数
#-v 返回非overlap区间
#-s 相同链上的feature
4. SRA格式
Sequence Read Archive (SRA) 数据库,用于储存来自不同高通量测序平台的原始序列数据和比对信息。
在提交测序数据到SRA数据库时,每一个样本会获得一个以SRR开头的数字编号:SRRxxx。
从SRA数据库下载公共的测试数据,原始格式即为 .sra。
如何下载.sra数据
可以用SRA数据库的SRA Toolkit下载sra数据。
如何分析.sra数据
使用fasterq_dump将sra转换为fastq,进行下一步处理。
注:fasterq-dump不支持gzip!
# 单个数据解压
fasterq-dump --split-3 -e 20 SRRxxx.sra -O fastq/
# 批量解压
for i in *sra
do
echo $i
fastqer-dump --split-3 -e 20 $i
done
使用SRA数据库时,可以看到有不同的ID前缀,比如SRA、SRR。
SRA数据库用6种不同的前缀区分不同的数据:
- SRA是提交数据时得到的数据号,包含下列5种前缀所组成的数据集。
- SRP表示 SRA Studies/projects;研究课题或项目的元数据;一个Study(SRP)可能包含多个experiments。
- SRX 表示 Experiments;包含元数据及其建库方式、测序平台、处理方式。
- SRR 表示 Runs;包含真实的测序数据;一个Experiment(SRX)可能有多个Run。
- SRS 表示 Samples;描述进行测序的物理样本的元数据。
- SRZ 表示 Analysis;包含序列数据分析的BAM文件和描述序列分析的元数据。
SRA中数据结构的层次关系为:Studies->Experiments->Samples->Runs。这样分类是以便更好的找到所需信息。
在挖掘生物数据信息时,会将未知的序列与已知的reference对比,从而找到未知序列中隐藏的信息。这就是序列比对。常见的序列比对的文件输出格式为sam和bam。
5. SAM格式
5.1 SAM格式文件的组成
Sequence Alignment Mapping (SAM) 格式包括两部分:
- 注释信息(header section)
- 比对结果(alignment section)
1.注释信息
注释信息可有可无,都是以@开头,用不同的tag表示不同的信息:
@HD,说明符合标准的版本、对比序列的排列顺序;
@SQ,参考序列说明;
@RG,比对上的序列(read)说明;
@PG,使用的程序说明;
@CO,任意的说明信息。
2.比对结果
比对结果必须通过11个tab间隔的字段来表示。注意,如果为空,则用*或0表示。
No. 名称 描述
1 QNAME Query NAME of the read or the read pair
2 FLAG Bitwise FLAG (pairing, strand, mate strand, etc.)
3 RNAME Reference sequence NAME
4 POS 1-Based leftmost POSition of clipped alignment
5 MAPQ MAPping Quality (Phred-scaled)
6 CIGAR Extended CIGAR string (operations: MIDNSHP)
7 MRNM Mate Reference NaMe (‘=’ if same as RNAME)
8 MPOS 1-Based leftmost Mate POSition
9 ISIZE Inferred Insert SIZE
10 SEQ Query SEQuence on the same strand as the reference
11 QUAL Query QUALity (ASCII-33=Phred base quality)
标准的CIGAR列描述的内容:
‘M’ for match/mismatch,
‘I’ for insertion compared with the reference
‘D’ for deletion.
为了更好的理解SAM格式,假设有下列原始数据。
1. Read r001/1 和 r001/2 组成一个 read pair
2. Read r003 是 chimeric read
3. Read r004 代表 split alignment
5.2 如何处理SAM格式数据
使用 SAMtools来处理SAM/BAM格式的数据:
# 安装
conda install samtools
# 常见用法
samtools view [options] <输入bam文件> # 将sam文件与bam文件互换,详细参数查看samtool --help
samtools sort [options] <输入bam文件> <输出bam文件名> # 排序
samtools merge [options] <输出bam文件> <输入bam文件1> <输入bam文件> # 合并文件
samtools flagstat <输入bam文件> # 统计bam文件中的比对flag信息,并输出比对统计结果
samtools index <排序后的bam文件> # 建立索引,输出bai文件,用于快速随机处理。
samtools faidx <输入fasta文件> # 建立索引,输出fai文件,用于快速随机处理。
samtools tview [options] [ref.fasta] # 显示reads比对基因组的情况,使用前要先建立index并sort
samtools mpileup # multi-way pileup,用于生成bcf文件,再使用samtool中附带的软件bcftools进行SNP和Indel的分析
5.6 BAM格式
Binary Alignment/Map (BAM)是SAM格式的二进制压缩格式,这两种格式是序列比对时软件常用的数据格式。
为什么有了SAM还要有BAM?
因为BAM占用的磁盘空间比SAM格式小,运算速度更快。
5.7 CRAM格式
有人习惯将其称为BAM的高压缩格式,因为它和BAM/SAM的格式基本相同。
可以通过samtools view -C -T hg19.fa N190446.sort.bam > N190446.sort.cram 命令进行bam和cram的转换,但是速度很慢,压缩后的文件更小了。
5.8 GFF格式
General Feature Format (GFF)格式,和BED格式一样,也可用于储存基因组或蛋白质特征。主要用来注释基因组。
目前主要用第三版GFF3,为更好的理解GFF3,例子如下:
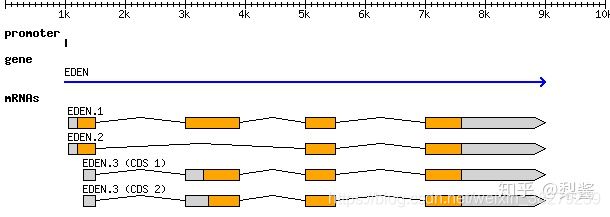
如果将上面这个基因用GFF3格式表示,则为:
0 ##gff-version 3.2.1
1 ##sequence-region ctg123 1 1497228
2 ctg123 . gene 1000 9000 . + . ID=gene00001;Name=EDEN
3 ctg123 . TF_binding_site 1000 1012 . + . ID=tfbs00001;Parent=gene00001
4 ctg123 . mRNA 1050 9000 . + . ID=mRNA00001;Parent=gene00001;Name=EDEN.1
5 ctg123 . mRNA 1050 9000 . + . ID=mRNA00002;Parent=gene00001;Name=EDEN.2
6 ctg123 . mRNA 1300 9000 . + . ID=mRNA00003;Parent=gene00001;Name=EDEN.3
7 ctg123 . exon 1300 1500 . + . ID=exon00001;Parent=mRNA00003
8 ctg123 . exon 1050 1500 . + . ID=exon00002;Parent=mRNA00001,mRNA00002
9 ctg123 . exon 3000 3902 . + . ID=exon00003;Parent=mRNA00001,mRNA00003
10 ctg123 . exon 5000 5500 . + . ID=exon00004;Parent=mRNA00001,mRNA00002,mRNA00003
11 ctg123 . exon 7000 9000 . + . ID=exon00005;Parent=mRNA00001,mRNA00002,mRNA00003
12 ctg123 . CDS 1201 1500 . + 0 ID=cds00001;Parent=mRNA00001;Name=edenprotein.1
13 ctg123 . CDS 3000 3902 . + 0 ID=cds00001;Parent=mRNA00001;Name=edenprotein.1
14 ctg123 . CDS 5000 5500 . + 0 ID=cds00001;Parent=mRNA00001;Name=edenprotein.1
15 ctg123 . CDS 7000 7600 . + 0 ID=cds00001;Parent=mRNA00001;Name=edenprotein.1
16 ctg123 . CDS 1201 1500 . + 0 ID=cds00002;Parent=mRNA00002;Name=edenprotein.2
17 ctg123 . CDS 5000 5500 . + 0 ID=cds00002;Parent=mRNA00002;Name=edenprotein.2
18 ctg123 . CDS 7000 7600 . + 0 ID=cds00002;Parent=mRNA00002;Name=edenprotein.2
19 ctg123 . CDS 3301 3902 . + 0 ID=cds00003;Parent=mRNA00003;Name=edenprotein.3
20 ctg123 . CDS 5000 5500 . + 1 ID=cds00003;Parent=mRNA00003;Name=edenprotein.3
21 ctg123 . CDS 7000 7600 . + 1 ID=cds00003;Parent=mRNA00003;Name=edenprotein.3
22 ctg123 . CDS 3391 3902 . + 0 ID=cds00004;Parent=mRNA00003;Name=edenprotein.4
23 ctg123 . CDS 5000 5500 . + 1 ID=cds00004;Parent=mRNA00003;Name=edenprotein.4
24 ctg123 . CDS 7000 7600 . + 1 ID=cds00004;Parent=mRNA00003;Name=edenprotein.4
5.9 GTF格式
Gene Transfer Format (GTF),和GFF类似,都有9列数据并用tab键分隔。
但GTF格式主要是用来对基因进行注释,目前广泛使用的是第二版GTF2。
9列分别为:seqname source feature start end score strand frame attributes
GFF和GTF格式的区别:
1.第3列feature/type不同;GFF可以为任意名称,而GTF则必须注明
2. 第9列attribures不同;GFF名称和值是以等号=隔开,GTF则是以空格隔开
可以使用cufflinks中的gffread转换GFF和GTF:
#gff2gtf
gffread file1.gff3 -T -o file1.gtf
#gtf2gff
gffread file2.gtf -o file2.gff3
5.10 VCF格式
VCF (Variant Call Format) version 4.0 是 1000 Genomes Project的标准数据格式之一,可用于储存SNP、INDEL和结构变异信息。
例子:
##fileformat=VCFv4.0
##fileDate=20090805
##source=myImputationProgramV3.1
##reference=1000GenomesPilot-NCBI36
##phasing=partial
##INFO=
##INFO=
##INFO=
##INFO=
##INFO=
##INFO=
##FILTER=
##FILTER=
##FORMAT=
##FORMAT=
##FORMAT=
##FORMAT=
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT NA00001 NA00002 NA00003
20 14370 rs6054257 G A 29 PASS NS=3;DP=14;AF=0.5;DB;H2 GT:GQ:DP:HQ 0|0:48:1:51,51 1|0:48:8:51,51 1/1:43:5:.,.
20 17330 . T A 3 q10 NS=3;DP=11;AF=0.017 GT:GQ:DP:HQ 0|0:49:3:58,50 0|1:3:5:65,3 0/0:41:3
20 1110696 rs6040355 A G,T 67 PASS NS=2;DP=10;AF=0.333,0.667;AA=T;DB GT:GQ:DP:HQ 1|2:21:6:23,27 2|1:2:0:18,2 2/2:35:4
20 1230237 . T . 47 PASS NS=3;DP=13;AA=T GT:GQ:DP:HQ 0|0:54:7:56,60 0|0:48:4:51,51 0/0:61:2
20 1234567 microsat1 GTCT G,GTACT 50 PASS NS=3;DP=9;AA=G GT:GQ:DP 0/1:35:4 0/2:17:2 1/1:40:3
六、 WDL
WDL是一种流程管理语言,内置的支持并行,适合编写pipeline
WDL (一个workflow description language)+ Cromwell(an execution engine that can run WDL scripts)是目前可以更好使用GATK的一套工具。
运行wdl脚本需要两步:第一步编辑参数列表对应的json文件,第二步直接运行Cromwell.jar
1. WDL的组成
workflow;task;call;command;output
1个WDL脚本包括1个workflow,1个workflow由多个task构成,并在workflow中通过call调用。其中每个task在workflow代码块外单独定义,且包含多个command。
变量
有2种不同层次的变量,一种存在于tasks,一种存在于整个workflow。变量也可从一个task传递到下一个task
1.1 workflow
workflow myWorkFlowName {
call task_A
call task_B
}
task task_A{
...}
task task_B{
...}
1.2 task
变量
变量需要定义其类型。主要有file,string,array[]。在command中引用需要${var}
command
定义运行的命令
output
定义输出的变量和文件
runtime
定义运行的环境
task task_A {
File ref
File in
String id
command {
do_stuff R=${ref} I=${in} O=${id}.ext
}
runtime{
docker: "ubuntu:latest"
memory: "2GB"
}
output {
File out="${id}.ext"
}
}
2. 任务调度
Parallelism即平行可以使得任务更快,而非顺序进行。
使用基于WDL standard library的scatter,会产生可平行的任务(成为一列Input,array),并且会输出结果(也是array)。
Scatter这个过程是外显的(explicit),而gather这个过程是不外显的(implicit)
Array[file] inputFiles
sactter (oneFile in inputFiles) {
call stepA {
input: in=oneFile}
}
call stepB {
input: files=stepA.out}
call stepA as firstSample {
input: in=firstInput}
call stepA as secondSample {
input: in=secondInput}
call stepB {
input: in=firstSample.out}
call stepB {
input: in=secondSample.out}
3. 运行WDL脚本
运行WDL脚本需要:(1) cromwell.jar (2) womtools.jar
step1
得到输入的参数列表,用json格式存储
java -jar womtools.jar inputs myWorkflow.wdl > myWorkflow_inputs.json
step2
运行脚本
java -jar Cromwell.jar run myWorkflow.wdl -inputs myWorkflow_inputs.json
七、全基因组测序WGS分析流程
1. DNA测序技术
1.1 第一代测序技术——sanger法
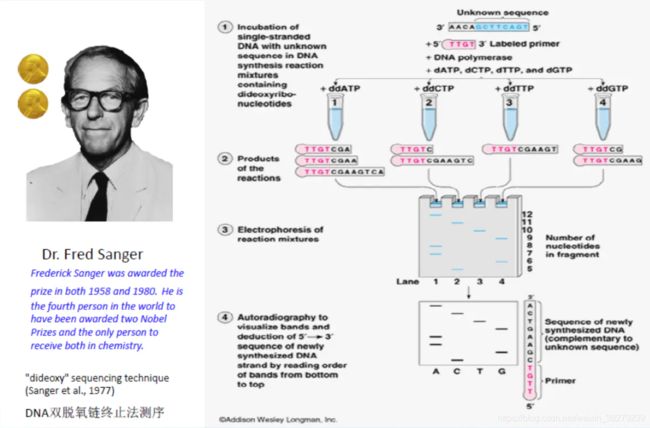
Sanger法是基于DNA合成反应的测序技术,又称为SBS法、末端终止法。1975年由Sanger提出,并于1977发表第一个完整的生物体基因组序列。
原理和特点
由于ddNTP(4种带有荧光标记的A,C,G,T碱基)的2’和3’都不含羟基,其在DNA的合成过程中不能形成磷酸二酯键,因此可以用来中断DNA的合成反应,在4个DNA合成反应体系中分别加入一定比例带有放射性同位素标记的ddNTP(分别为:ddATP,ddCTP,ddGTP和ddTTP),然后利用凝胶电泳和放射自显影后可以根据电泳带的位置确定待测分子的DNA序列。
在每个反应体系中,ddNTP相对于dNTP是很少的,所以只有部分新链在不同的位置特异性终止,最终就会得到一系列长度不一的序列。
然后对这些DNA片段的混合物进行纯化,也就是去除掉游离的核苷酸和上面游离ddNTP的对这些混合物进行跑胶电泳分析,不同质量的会在电泳上排列在不同的位置,质量小的在上面,重的在下面,最后按顺序对每个序列检测尾部的荧光颜色序列读取每一个的颜色,就可以得到整个DNA片段序列信息了。
第一代测序技术的主要特点为,测序读长可达1000bp,准确性高达99% 。但是费用高,通量低,是先合成,再测序的方法。
1.2 第二代测序技术——illumina
第二代测序技术大大降低了测序成本的同时,还大幅提高了测序速度,并且保持了高准确性。第二代测序技术的核心思想是边合成边测序(Sequencing by Synthesis)。
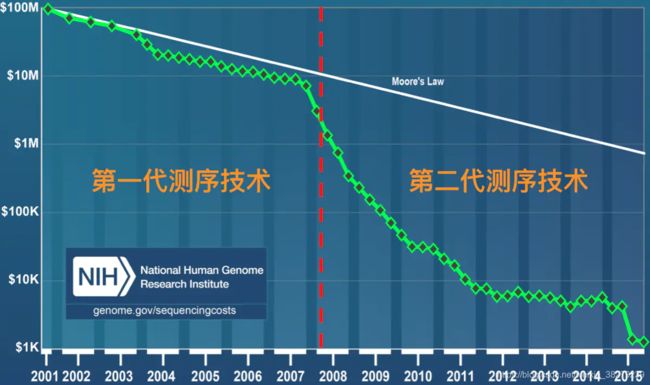
第二代测序技术在大幅提高了测序速度的同时,还大大地降低了测序成本,并且保持了高准确性,以前完成一个人类基因组的测序需要3年时间,而使用二代测序技术则仅仅需要1周,但其序列读长则短很多,大多只有100bp-150bp。
第一代和第二代测序技术测序成本作了一个简单的比较,可以看出自第二代测序技术发展出来之后,历史开始发生根本性的改变,测序的成本开始快速实现断崖式下降,也就是业内经常提到的超摩尔定律现象。
Illumina循环SBS法(cycle SBS)即SBRT(Sequencing By Reversible Termination,可逆终止)的核心技术是DNA合成的可逆性末端循环,即3’-OH可逆性的修饰和去修饰。
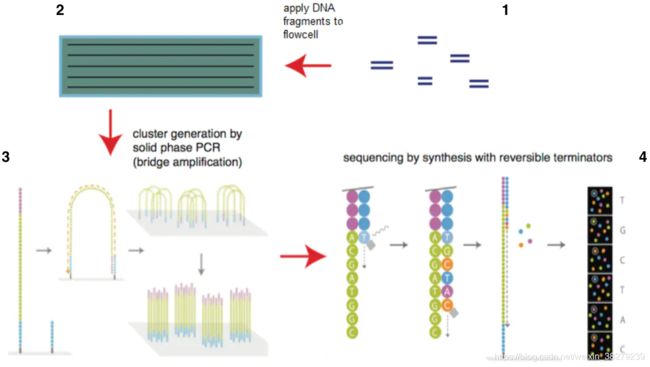
目前illumina的测序仪占全球75%以上,以HiSeq系列为主。它的机器采用的都是边合成边测序的方法,主要分为以下4个步骤:
- 构建DNA测序文库
简单来说就是把一堆乱糟糟的DNA分子用超声波打断成一定长度范围的小片段。目前除了一些特殊的需求之外,基本都是打断为300bp-800bp长的序列片段,并在这些小片段的两端添加上不同的接头,构建出单链DNA文库,以备测序之用。
纯化后的连接产物构成了测序文库,测序文库是测序是否成功的关键步骤。
【注】接头在illumina中一般分为P5和P7接头,其中一个带有和flowcell上的探针反向互补的序列,以完成待测序列和探针结合的作用,另外一个接头带有barcord序列以区分不同的样本。
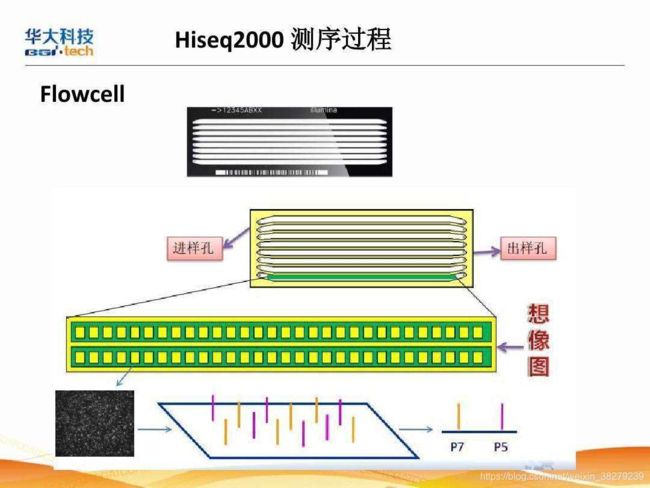
- 测序流动槽 flowcell
flowcell是用于吸附流动DNA片段的槽道,也是核心的测序反应容器——所有的测序过程就发生在这里。当文库建好后,这些文库中的DNA在通过flowcell的时候会随机附着在flowcell表面的槽道(称为lane)上。
如下图所示,每个flowcell有8个lane,每个lane的表面都附有很多接头,这些接头能和建库过程中加在DNA片段两端的接头相互配对,这就是为什么flowcell能吸附建库后的DNA的原因,并能支持DNA在其表面进行桥式PCR的扩增,理论上这些lane之间是不会相互影响的。

Flowcell(流动池)是有着2个或8个lane(泳道)的玻璃板,每个lane可以测一个样本或者多样本的混合物,且随机布满了能够与文库两端接头分别互补配对或一致的寡核苷酸(oligos,P7和P5接头)。一个lane包含两列,每一列有60个tile,每个tile会种下不同的cluster,每个tile在一次循环中会拍照4次(每个碱基一次)。
P5和P7是不同的,它们分别和flowcell上的接头互补和相同。为了方便阐述,将与P5互补的接头称为P5’,与P7互补的接头称为P7’。
index1和index2也是不同的,与P5相连的是index2,与P7相连的是index1
关于index,也叫barcodes,因为一个lane可以同时测多个样品,为了避免混淆样品的read products,每种样品的DNA由一种index修饰,这样测序得到的reads都是具有index标记的,在测序结果中,依据之前标签与样品的对应关系,就可以获得对应样品的数据。而这里的index1和index2是为了区分paired-end测序得到的双端reads。
-
PCR桥式扩增与变性
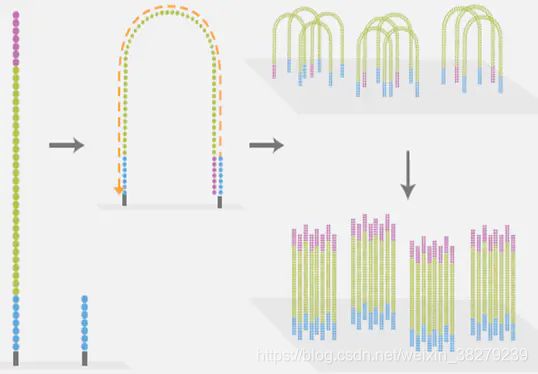
这是高通量测序技术NGS的一个核心特点。桥式PCR以flowcell表面所固定的序列为模板,进行桥形扩增,如下图所示。经过不断的扩增和变性循环,最终每个DNA片段都将在各自的位置上集中成束,每一个束都含有单个DNA模板的很多分拷贝,经桥式扩增把每个待测片段扩增到1000个拷贝左右。这一过程的目的在于实现将单一碱基的信号强度进行放大,以达到测序所需的信号要求。
-
测序
测序方法采用边合成边测序的方法。向反应体系中同时添加DNA聚合酶、接头引物和带有碱基特异荧光标记的4中dNTP(如同Sanger测序法)。这些dNTP的3’-OH被化学方法所保护,因而每次只能添加一个dNTP,这就确保了在测序过程中,一次只会被添加一个碱基。同时在dNTP被添加到合成链上后,所有未使用的游离dNTP和DNA聚合酶会被洗脱掉。接着,再加入激发荧光所需的缓冲液,用激光激发荧光信号,并有光学设备完成荧光信号的记录,最后利用计算机分析将光学信号转化为测序碱基。这样荧光信号记录完成后,再加入化学试剂淬灭荧光信号并去除dNTP 3’-OH保护基团,以便能进行下一轮的测序反应。
测序过程中,往每个泳道里加入DNA聚合酶和四种不同荧光标记的特殊核苷酸(A T C G)。在酶的催化作用下,从测序引物结合部位开始合成与测序模板互补的DNA链,每次加入一个核苷酸。一轮合成反应结束后,每一个簇被激发产生不同的荧光,由测序仪的光学系统拍照并记录,特定波长的荧光代表不同的核苷酸,从而实现了待测模板第一个碱基的测序。
即将dNTP的3’-OH以叠氮集团RTG(Reversible Terminating Group,可逆末端基团)进行修饰;将4种碱基分别与不同的荧光分子连接;DNA合成时,RTG能起到类似于ddNTP的作用终止反应;每次合成反应终止并读取信号之后,洗脱RTG和荧光分子,进行下一轮循环。
一个测序循环结束后,核苷酸3’端的屏蔽基团被酶切除,从而可以进行下一个循环的合成测序,合成的下一个核苷酸产生的荧光再次被记录。如此周而复始,经过100或150个循环后,实现了每个簇上待测模板的100bp/150bp的正向单向测序。
如果要进行双端测序,在单向测序完成后,往系统中加入缓冲液,洗掉测序过程中合成的DNA链,系统合成待测序列的互补链,然后以互补链为测序模板链,以与正向测序的方式进行反向测序,这样就得到了与正向序列相对的反向序列。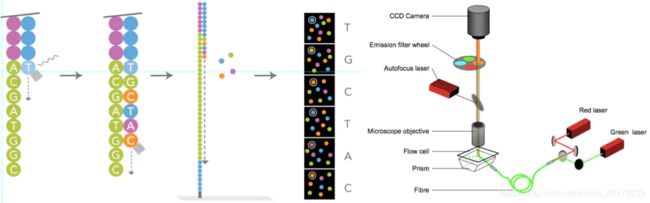
一个簇的图像数据就是一个DNA序列(read),每条泳道(lane)上簇的密度可达750~850K/mm2,这样在一张芯片上簇的数量可以达到数亿或数十亿个。然后再通过第二种引物检测index上的,继续测序数个碱基,这里的测序就不是为了DNA测序,而是为了确定每个接头上DNA片段的来源。
虽然每个接头能测序的量不多,但是由于每个Flowcell有上亿个接头,因此二代测序的通量是非常大的,准确率也是非常的高。
llumina的这种每次只添加一个dNTP的技术特点能够很好的地解决同聚物长度的准确测量问题,它的主要测序错误来源是碱基的替换,目前它的测序错误率在1%-1.5%左右。测序周期以人类基因组重测序为例,30x-50x测序深度对于Hisq系列需要3-5天时间,而对于2017年初最新推出的NovaSeq系列则只需要40个小时!
目前二代测序存在的测序错误主要原因有以下两点:
- Phasing,也就是由于酶的活性的原因,部分接头上的叠氮基没有掉落,导致分子掉队的现象,也就是没有聚合下一个碱基,随着聚合循环次数增多,会导致Phasing现象越来越严重,这也是为什么二代测序的长度一般比较短。
- Prephasing,这种情况产生的原因和Phasing的原因相反,是因为多结合了一个碱基,也就是单次聚合了两个或者以上的碱基,出现这种现象的原因就是叠氮基不稳定,容易掉落,一旦当前聚合的碱基中的叠氮基掉落,他就可以结合新的ddNTP,导致超前的聚合。
因此Illumina公司用一些PF(phase filter)率或者质量分数(quality score)来评判测序结果的可靠性。
不同系列测序仪的比较
测序量比较(双流动槽为例,如为单流动槽则测序量减少为下表的一半,时间不变)
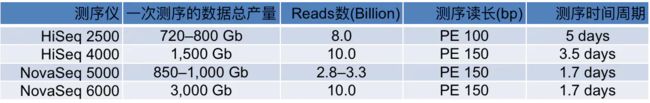
一次测序的数据总产量的单位Gb,不是计算机字节,而是测序碱基的数目(Giga base)
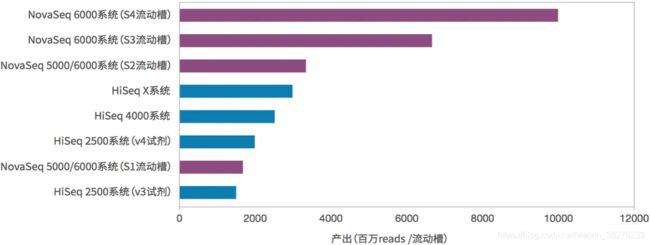
上面表图是NovaSeq和其他测序系列的比较,数据相当好。按照这个数据量估算,一台NovaSeq 6000(S4)在跑满的情况下,一年就可以测序6400多人!而且按照以往的经验,illumina的官方公布的数据都是偏于保守的,我们在实际的使用过程中发现 高质量(Q30)的read其实占到了总数据的90%以上,远高于官方公布的75%,数据的总产量也同样更高。
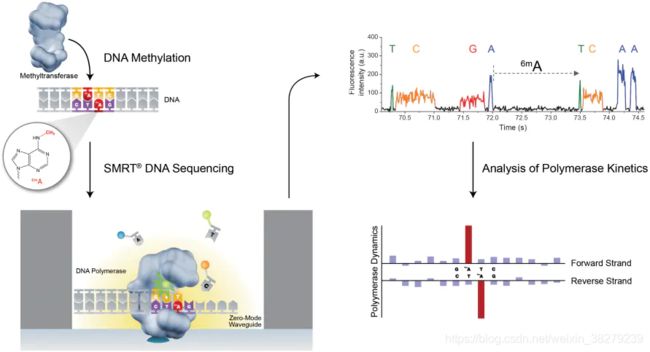
1.3 第三代测序技术——PacBio SMRT技术(纳米孔单分子技术)
目前第三代测序技术主要为以PacBio公司的SMRT为主,最大的特点就是单分子测序,对于DNA 样本不需要经过PCR 扩增,超长读长,测序读长平均达到10Kb-15Kb,是二代测序技术的100倍以上。
原理和特点
PacBio SMRT技术其实也应用了边合成边测序的思想,并以SMRT芯片为测序载体(如同flowcell)。
基本原理是: DNA聚合酶和模板结合,用4色荧光标记A,C,G,T这4种碱基(即是dNTP)。在碱基的配对阶段,不同的碱基加入,会发出不同的光,根据光的波长与峰值可判断进入的碱基类型。

这个DNA聚合酶是实现超长读长的关键之一,读长主要跟酶的活性保持有关,它主要受激光对其造成的损伤所影响。PacBio SMRT技术的一个关键点是在于如何将反应信号与周围游离碱基的强大荧光背景区别出来。
他们利用的是ZMW(零模波导孔)原理:在一个反应管(SMRTCell:单分子实时反应孔)中有许多这样的圆形纳米小孔,即 ZMW(零模波导孔),外径100多纳米,比检测激光波长小(数百纳米),激光从底部打上去后不会穿透小孔进入上方的溶液区,能量会被限制在一个小范围(体积20X 10-21 L)里(图A),正好足够覆盖需要检测的部分,使得信号仅仅只是来自于这个小反应区域,孔外过多的游离核苷酸单体依然留在黑暗中,从而实现将背景噪音降到最低的目的。
在纳米室底部有一个孔径70nm的小孔,由于远远小于激光的波长,所以激光从底部照射时,只会照亮一个小的区域,提高了信噪比。
PacBio SMRT技术除了能够检测普通的碱基之外,还可以通过检测相邻两个碱基之间的测序时间,来检测碱基的表观修饰情况,如甲基化。因为假设某个碱基存在表观修饰,则通过聚合酶时的速度会减慢,那么相邻两峰之间的距离会增大,我们可以通过这个时间上的差异来检测表观甲基化修饰等信息。
流程
- 聚合酶捕获文库DNA序列,锚定在零模波导孔底部
- 4种不同荧光标记的dNTP随机进入零模波导孔底部
- 荧光dNTP被激光照射,发出荧光,检测荧光
- 荧光dNTP与DNA模板的碱基匹配,在酶的作用下合成一个碱基
- 统计荧光信号,获得DNA序列
- 酶反应过程中,一方面使链延伸,另一方面使dNTP上的荧光基团脱落
- 聚合反应持续进行,测序同时持续进行
缺点
SMRT技术的测序速度很快,每秒约10个dNTP。但是,同时其测序错误率比较高(这几乎是目前单分子测序技术的通病),达到15%,但好在它的出错是随机的,并不会像第二代测序技术那样存在测序错误的偏向,因而可以通过多次测序来进行有效的纠错。此外,不能高效的将DNA聚合酶加入到阵列中,而且DNA聚合酶的降解也是丞待解决的问题 。
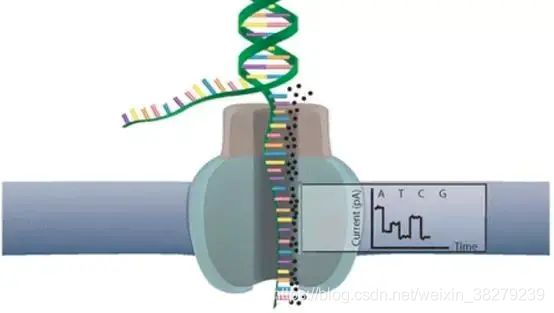
1.4 第三代测序技术——Oxford Nanopore
单分子实时测序的新一代技术,主要是通过ssDNA或RNA模板分子通过纳米孔而带来的“电信号”变化推测碱基组成进行实时测序。
原理和特点
当纳米孔充满导电液时,两端加上一定电压,分子模板通过纳米孔生成可测量电流。纳米孔的直径只能容纳一个核苷酸,单链模板就会在电场作用下依次通过纳米孔而引起电流强度变化,通过检测相应的电流峰判断碱基,实现实时测序。
四种核苷酸的空间构象不一样,因此当它们通过纳米孔时,所引起的电流变化不一样。由多个核苷酸组成的DNA或RNA链通过纳米孔时,检测通过纳米孔电流的强度变化,即可判断通过的核苷酸类型,从而进行实时测序。
这个技术的关键点在于他们所设计的一种特殊纳米孔,孔内共价结合分子接头。当DNA分子通过纳米孔时,它们使电荷发生变化,从而短暂地影响流过纳米孔的电流强度(每种碱基所影响的电流变化幅度是不同的),最后高灵敏度的电子设备检测到这些变化从而鉴定所通过的碱基。
纳米孔测序以及其他第三代测序技术,有可能会彻底地解决目前第二代测序平台的诸多不足。另外,MinION的主要特点是:读长很长,而且比PacBio的都长得多,基本都是在几十kb上百kb以上,最新的数据显示可以达到900 kb!错误率是5%-15%,也是随机错误,MinION最大的特点除了极小的体积之外,就是数据将是可实时读取的,并且起始DNA在测序过程中不被破坏!
此外,单分子纳米孔技术能够检测DNA甲基化,它能直接读出被甲基化的胞嘧啶,这对于在基因组水平直接研究表观遗传相关现象具有极大的帮助。
纳米孔技术读长很长,大约在几十kb,且通量高。然而原始的DNA可能被读错方向,对连续碱基的检测误差较大等问题仍然是一大痛点。

2. 四大测序技术的优缺点
- Sanger法测序读长长、准确度高,但是通量不高;
- Illumina测序读长短、通量高、准确度高,在进行基因组组装或者结构变异分析的时候没有优势,可用作三四代测序read的纠错;
- Pacbio测序读长长、通量高、准确度不高,但可通过测序深度弥补,GC偏差低,可进行甲基化的直接测序。
- Nanopore测序读长长、通量高、准确度低,不可通过测序深度弥补,但可通过Illumina read 纠错,可进行甲基化的直接测序。
2. FASTA&FASTQ
在WGS数据的分析过程中,我们会接触到许多生物信息学/基因组学领域所特有的数据文件和它们特殊的格式,在这一节中将要介绍的FASTA和FASTQ便是其中之一二。这是我们存储核苷酸序列信息(就是DNA序列)或者蛋白质序列信息最常使用的两种 文本文件,虽然看起来名字有些古怪,但它们完全是纯文本文件(如同.txt)!名字的发音分别是fast-A和fast-Q。
2.1 FASTA
FASTA这个文件的来源为一款名叫“FASTA”的比对软件,名字中最后一个字母A,其实就是Alignment的意思。
生物学家和遗传学家们决定把FASTA作为这种存储 有顺序的序列数据的文件后缀【注】,这包括我们常用的参考基因组序列、蛋白质序列、编码DNA序列(coding DNA sequence,简称CDS)、转录本序列等文件都是如此,文件后缀除了.fasta之外,也常用.fa或者.fa.gz(gz压缩)。
【注】这里的序列、序列数据,指的其实就是表示DNA或者蛋白质的一条字符串。
重点注意 是有顺序的。因此,我们可以通过数个数,就知道某个DNA碱基在某个基因组上的准确位置,这个位置会用所在序列的名字和所在位置来表达,比如基因数据比对的结果,方便后续数据分析。
FASTA的格式
FASTA文件主要由两个部分构成:序列头信息(有时包括一些其它的描述信息)和具体的序列数据。
头信息独占一行,以大于号(>)开头作为识别标记,其中除了记录该条序列的名字之外,有时候还会接上其它的信息。紧接的下一行是具体的序列内容,直到另一行碰到另一个大于号(>)开头的新序列或者文件末尾。
下面给出一个FASTA文件的例子,这是我们人类一个名为EGFR基因的部分序列。
>ENSMUSG00000020122|ENSMUST00000138518
CCCTCCTATCATGCTGTCAGTGTATCTCTAAATAGCACTCTCAACCCCCGTGAACTTGGT
TATTAAAAACATGCCCAAAGTCTGGGAGCCAGGGCTGCAGGGAAATACCACAGCCTCAGT
TCATCAAAACAGTTCATTGCCCAAAATGTTCTCAGCTGCAGCTTTCATGAGGTAACTCCA
GGGCCCACCTGTTCTCTGGT
>ENSMUSG00000020122|ENSMUST00000125984
GAGTCAGGTTGAAGCTGCCCTGAACACTACAGAGAAGAGAGGCCTTGGTGTCCTGTTGTC
TCCAGAACCCCAATATGTCTTGTGAAGGGCACACAACCCCTCAAAGGGGTGTCACTTCTT
CTGATCACTTTTGTTACTGTTTACTAACTGATCCTATGAATCACTGTGTCTTCTCAGAGG
CCGTGAACCACGTCTGCAAT
可以看到,FASTA其实很简单,但它往往都很大,比如人类基因组有30亿个碱基,就是30亿个字符存储在这样的一个文本文件中,就算是压缩也要占用约1GB的存储空间。
FASTA特点
第一,除了序列内容之外,FASTA的头信息并没有被严格地限制。这个特点有时会带来很多麻烦的事情,比如有时我们会看到相同的序列被不同的人处理之后、甚至是在不同的网站上或者数据库中它们的头信息都不尽相同,比如以下的几种情况都是可能存在的。
>ENSMUSG00000020122|ENSMUST00000125984
> ENSMUSG00000020122|ENSMUST00000125984
>ENSMUSG00000020122|ENSMUST00000125984|epidermal growth factor receptor
>ENSMUSG00000020122|ENSMUST00000125984|Egfr
>ENSMUSG00000020122|ENSMUST00000125984|11|ENSFM00410000138465
这对于程序处理来说,凌乱的格式显然是不合适的。因此后来在业内也慢慢地有一些不成文的规则被大家所使用,那就是,用一个空格把头信息分为两个部分:第一部分是序列名字,它和大于号(>)紧接在一起;第二部分是注释信息,这个可以没有,就看具体需要,比如下面这个序列例子,除了前面gene_00284728这个名字之外,注释信息(length=231;type=dna)给出这段序列的长度和它所属的序列类型。
>gene_00284728 length=231;type=dna
GAGAACTGATTCTGTTACCGCAGGGCATTCGGATGTGCTAAGGTAGTAATCCATTATAAGTAACATG
CGCGGAATATCCGGGAGGTCATAGTCGTAATGCATAATTATTCCCTCCCTCAGAAGGACTCCCTTGC
GAGACGCCAATACCAAAGACTTTCGTAAGCTGGAACGATTGGACGGCCCAACCGGGGGGAGTCGGCT
ATACGTCTGATTGCTACGCCTGGACTTCTCTT
虽然这样的格式还不算是真正的标准,但却非常有助于我们的数据分析和处理,很多生信软件(如:BWA,samtools,bcftools,bedtools等)都是将第一个空格前面的内容认定为序列名字来进行操作的
第二,FASTA由于是文本文件,它里面的内容是否有重复是无法自检的,在使用之前需要我们进行额外的检查。这个检查倒不用很复杂,只需检查序列名字是否有重复即可。但对于那些已经成为标准使用的参考序列来说,都有专门的团队进行维护,因此不会出现这种内容重复的情况,可以直接使用,但对于其它的一些序列来说,谨慎起见,最好进行检查。
2.2 FASTQ
这是目前存储测序数据最普遍、最公认的一个数据格式,另一个是uBam格式。
上面所讲的FASTA文件,它所存的都是已经排列好的序列(如参考序列),FASTQ存的则是产生自测序仪的原始测序数据,它由测序的图像数据转换过来,也是文本文件,文件大小依照不同的测序量(或测序深度)而有很大差异,小的可能只有几M,大的则常常有几十G上百G,文件后缀通常都是.fastq,.fq或者.fq.gz(gz压缩)。
FASTQ的格式
以下是它的一个例子:
@DJB775P1:248:D0MDGACXX:7:1202:12362:49613
TGCTTACTCTGCGTTGATACCACTGCTTAGATCGGAAGAGCACACGTCTGAA
+
JJJJJIIJJJJJJHIHHHGHFFFFFFCEEEEEDBD?DDDDDDBDDDABDDCA
@DJB775P1:248:D0MDGACXX:7:1202:12782:49716
CTCTGCGTTGATACCACTGCTTACTCTGCGTTGATACCACTGCTTAGATCGG
+
IIIIIIIIIIIIIIIHHHHHHFFFFFFEECCCCBCECCCCCCCCCCCCCCCC
它有着自己独特的格式:每四行成为一个独立的单元,称之为read。具体的格式描述如下:
-
****第一行:以‘@’开头,是这一条read的名字,这个字符串是根据测序时的状态信息转换过来的,中间不会有空格,它是每一条read的唯一标识符,同一份FASTQ文件中不会重复出现,甚至不同的FASTQ文件里也不会有重复;
-
第二行:测序read的序列,由A,C,G,T和N这五种字母构成,这也是我们真正关心的DNA序列,N代表的是测序时那些无法被识别出来的碱基;
-
第三行:以‘+’开头,在旧版的FASTQ文件中会直接重复第一行的信息,但现在一般什么也不加(节省存储空间);
-
第四行:测序read的质量值,这个和第二行的碱基信息一样重要,它描述的是每个测序碱基的可靠程度,用ASCII码表示。
2.3 碱基质量值
碱基质量值就是能够用来定量描述碱基 好坏程度的一个数值。如果测序测得越准确,这个碱基的质量就应该越高;反之,测得越不准确,质量值就应该越低。也就是说可以利用碱基被测错的概率来描述它的质量值,错误率越低,质量值就越高!
如下图,红线代表错误率,蓝线代表质量值,这便是我们希望达到的效果:
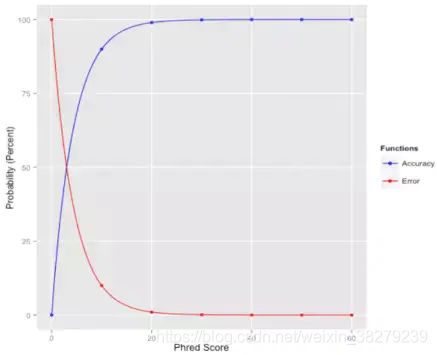
假定碱基的测序错误率为: P e r r o r {P}_{error} Perror质量值为Q,它们之间的关系如下:
Q = − 10 l o g 10 P e r r o r Q=-10log_{10}{P}_{error} Q=−10log10Perror
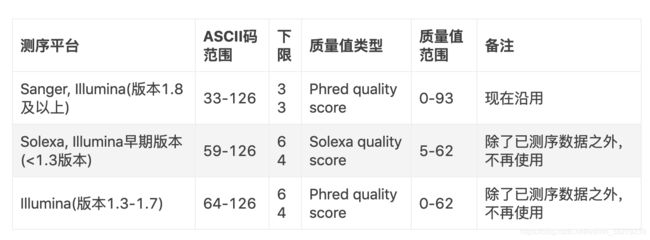
质量值是测序错误率的对数(10为底数)乘以-10(并取整)。这个公式也是目前测序质量值的计算公式,它非常简单,p_error的值和测序时的多个因素有关,体现为测序图像数据点的清晰程度,并由测序过程中的base calling 算法计算出来;公式右边的Q称之为Phred quality score,就是用它来描述测序碱基的靠谱程度。比如,如果该碱基的测序错误率是0.01,那么质量值就是20(俗称Q20),如果是0.001,那么质量值就是30(俗称Q30)。Q20和Q30的比例常常被我们用来评价某次测序结果的好坏,比例越高就越好。
下面我也详细给出一个表,更进一步地解释质量值高低的含义:
为什么要用ASCII码来代表,直接用数字不行吗?行!但很难看,而且数字不能直接连起来,还得在中间加一个分隔符,长度也对不齐,还占空间,又不符合美学设计。
因此,也是为了格式存储以及处理时的方便,这个数字被直接转换成了ASCII码,并与第二行的read序列构成一一对应的关系——每一个ASCII码都和它正上面的碱基对应,这就很完美。
2.4 质量体系
ASCII码虽然能够从小到大表示0-127的整数,但是并非所有的ASCII码都是 可见的字符,比如所有小于33的ASCII码值所表示的都是不可见字符,比如空格,换行符等,因此 为了能够让碱基的质量值表达出来,必须避开所有这些不可见字符。最简单的做法就是加上一个固定的整数!
但一开始对于要加哪一个整数,并没有什么指导标准,这就导致了在刚开始的时候,不同的测序平台加的整数也不同,总的来说有以下3种质量体系,演变到现在也基本只剩下第一种了,如下表
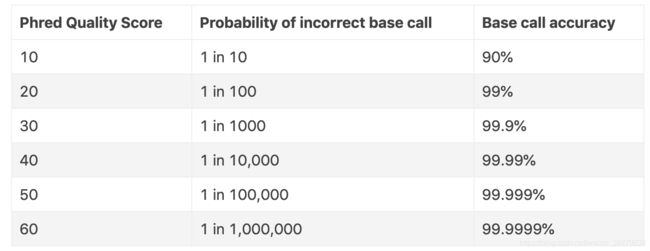
从表中可以看到下限有33和64两个值,我们把加33的的质量值体系称之为Phred33,加64的称之为Phred64(Solexa的除外,它叫Solexa64)。不过,现在一般都是使用Phred33这个体系,而且33也恰好是ASCII的第一个可见字符(’!’)
如果在实际做项目的过程不知道所用的质量体系(经验丰富者是可以直接看出来的),那么可以用下面这一段代码,简单地做个检查:
less $1 | head -n 1000 | awk '{if(NR%4==0) printf("%s",$0);}' \
| od -A n -t u1 -v \
| awk 'BEGIN{min=100;max=0;} \
{for(i=1;i<=NF;i++) {if($i>max) max=$i; if($i$i ;}}END \
{if(max<=126 && min<59) print "Phred33"; \
else if(max>73 && min>=64) print "Phred64"; \
else if(min>=59 && min<64 && max>73) print "Solexa64"; \
else print "Unknown score encoding"; \
print "( " min ", " max, ")";}'
将上面这段代码复制到任意一份shell文件中(比如:fq_qual_type.sh),就可以用它来进行质量值类型的检查了。代码的思路其实比较简单,就是截取FASTQ文件的前1000行数据,并抽取出质量值所在的行,分别计算出其中最小和最大的ASCII值,再比较一下就判断出来了。下面给出一个例子,这是我们在本文中用到的FASTQ文件,它是Phred33的:
$ sh fq_qual_type.sh untreated.fq
Phred33
( 34, 67 )
另外,在查看碱基质量值的过程中,如果存有ASCII码表当然可以直接“看”出各个碱基的质量值,但在实际的场景中都是通过程序直接进行转换处理。下面用Python的ord()函数举个转换的例子:
In [1]: qual='JJJJJIIJJJJJJHIHHHGHFFFFFFCEEEEEDBD'
In [2]: [ord(q)-33 for q in qual]
Out[2]:
[35, 20, 17, 18, 24, 34, 35, 35, 35, 34, 35, 34, 29, 29, 32, 32, 34, 34, 33,
29, 33, 33, 32, 35, 35, 35, 34, 34, 34, 34, 35, 35, 34, 35, 34, 35, 34, 35,
34, 34, 34, 35, 35, 35, 35, 34, 33, 33, 30, 33, 24, 27]
这里的ord()函数会将字符转换为ASCII对应的数字,减掉33后就得到了该碱基最后的质量值(即,Phred quality score)。
另外,根据上面phred quality score的计算公式,可以很方便地获得每个测序碱基的错误率,这个错误率在比对和变异检测中都十分重要,以下先给出一个转换的例子,还是以上述qual为例子:
In [1]: qual='JJJJJIIJJJJJJHIHHHGHFFFFFFCEEEEEDBD'
In [2]: phred_score = [ord(q)-33 for q in qual]
In [3]: [10**(-q/10.0) for q in phred_score]
Out[3]:
[3e-04, 1e-02, 2e-02, 2e-02, 4e-03, 4e-04, 3e-04, 3e-04, 3e-04,
4e-04, 3e-04, 4e-04, 1e-03, 1e-03, 6e-04, 6e-04, 4e-04, 4e-04,
5e-04, 1e-03, 5e-04, 5e-04, 6e-04, 3e-04, 3e-04, 3e-04, 4e-04,
4e-04, 4e-04, 4e-04, 3e-04, 3e-04, 4e-04, 3e-04, 4e-04, 3e-04,
4e-04, 3e-04, 4e-04, 4e-04, 4e-04, 3e-04, 3e-04, 3e-04, 3e-04,
4e-04, 5e-04, 5e-04, 1e-03, 5e-04, 4e-03, 2e-03]
这其实就是根据phred quality score的定义进行简单的指数运算。
3. WGS主流程
流程的具体形式其实是次要的,WGS本质上只是一个技术手段,重要的是,要明白自己所要解决的问题是什么,所希望获取的结果是什么,然后再选择合适的技术。
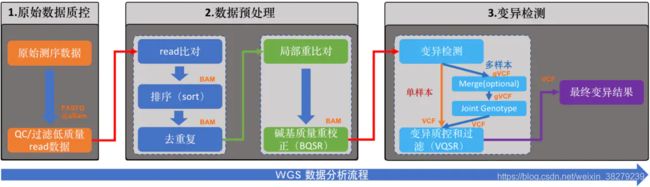
这是WGS数据分析的流程图。流程的目的是准确检测出每个样本(这里特指人)基因组中的变异集合,也就是人与人之间存在差异的那些DNA序列。
整个分析过程按照它们实际要完成的功能,将其分成了三个大的模块:
- 原始数据质控
- 数据预处理
- 变异检测
准备阶段
在开始之前,我们需要做一些准备工作,主要是部署好相关的软件和工具。我们在这个WGS数据分析过程中用到的所有软件都是开源的,它们的代码全部都能够在github上找到,具体如下:
- BWA(Burrow-Wheeler Aligner): 这是最权威,使用最广的NGS数据比对软件,目前已经更新到0.7.16版本;
- Samtools: 是一个专门用于处理比对数据的工具,由BWA的作者(lh3)所编写;
- Picard: 它是目前最著名的组学研究中心-Broad研究所开发的一款强大的NGS数据处理工具,功能方面和Samtools有些重叠,但更多的是互补,它是由java编写的,我们直接下载最新的.jar包就行了。
- GATK: 同样是Broad研究所开发的,是目前业内最权威、使用最广的基因数据变异检测工具。值得注意的是,目前GATK有3.x和4.x两个不同的版本,代码在github上也是分开的。4.x是今年新推出的,在核心算法层面并没太多的修改,但使用了新的设计模式,做了很多功能的整合,是更适合于大规模集群和云运算的版本,后续GATK团队也将主要维护4.x的版本,而且它的代码是100%开源的,这和3.x只有部分开源的情况不同。看得出GATK今年的这次升级是为了应对接下来越来越多的大规模人群测序数据而做出的改变,但现阶段4.x版本还不稳定,真正在使用的人和机构其实也还不多。短期来看,3.x版本还将在业内继续使用一段时间;其次,3.x对于绝大分部的分析需求来说是完全足够的。我们在这里也以GATK3.8(最新版本)作为流程的重要工具进行分析流程的构建。
事实上,对于构造WGS分析流程来说,以上这个四个工具就完全足够了。它们的安装都非常简单,除了BWA和Samtools由C编写的,安装时需要进行编译之外,另外两个只要保证系统中的java是1.8.x版本及以上的,那么直接下载jar包就可以使用了。操作系统方面推荐linux(集群)或者Mac OS。
3.1 原始数据质控
数据质控的意义
现在的NGS测序,以illumina为首基本都是运用边合成边测序的技术。碱基的合成依靠的是化学反应,这使得碱基链可以不断地从5’端一直往3’端合成并延伸下去。但在这个合成的过程中随着合成链的增长,DNA聚合酶的效率会不断下降,特异性也开始变差,这就会带来一个问题——越到后面碱基合成的错误率就会越高【注】,这也是为何当前NGS测序读长普遍偏短的一个原因。
【注】:有时候测序仪在刚开始进行合成反应的时候也会由于反应还不够稳定,同样会带来质量值的波动,不过这个波动一般都在高质量值区域(如下图)。
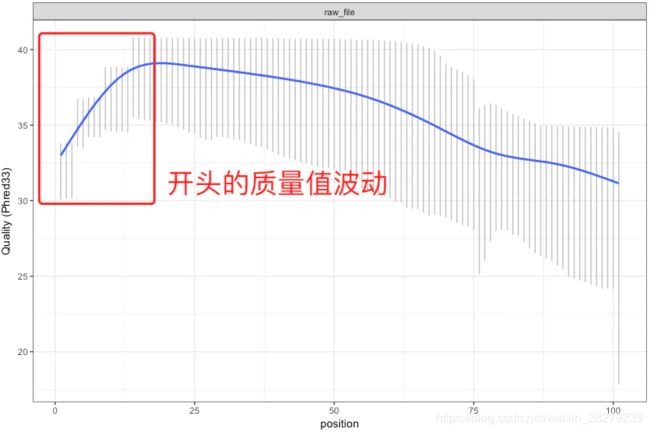
测序数据的质量好坏会影响我们的下游分析。但不同的测序平台其测序错误率的图谱都是有差别的。因此,非常建议在我们分析测序数据之前先搞清楚如下两个地方:
- 原始数据是通过哪种测序平台产生的,它们的错误率分布是怎么样的,是否有一定的偏向性和局限性,是否会显著受GC含量的影响等;
- 评估它们有可能影响哪些方面的分析;
第一点是我们认识数据质量的第一步,也是一定要知道的点。除了看官方的资料之外,最好的做法是自己分析。认识你的数据,不要过于相信你的工具! 这样也能够更好地避开很多不必要的坑。
第二点需视情况而定,例如你的测序深度是多少,检测变异的时候,变异的位点是否过于集中在read的末尾,比对的时候是否会出现了一定的正反链偏向性等诸如此类的问题;又或者我们在进行基因组序列组装的时候,由于对read的中出错的碱基更加敏感,因此往往需要进行更严格的切除,不然会由于这些错误的碱基消耗更大的计算资源和时间。
认识原始测序数据FASTQ
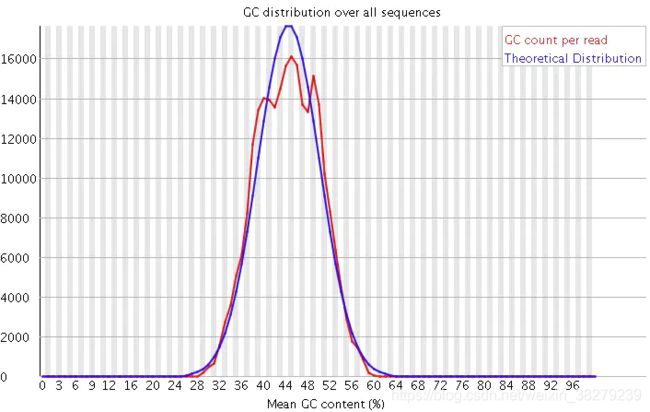
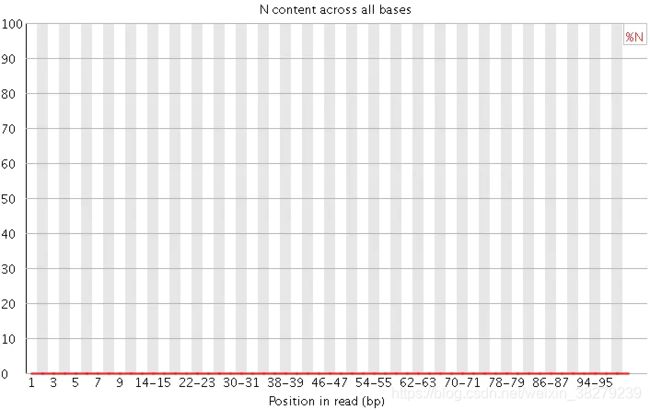
一般我们可以从如下几个方面来分析:
- read各个位置的碱基质量值分布
- 碱基的总体质量值分布
- read各个位置上碱基分布比例,目的是为了分析碱基的分离程度
- GC含量分布
- read各位置的N含量
- read是否还包含测序的接头序列
- read重复率,这个是实验的扩增过程所引入的
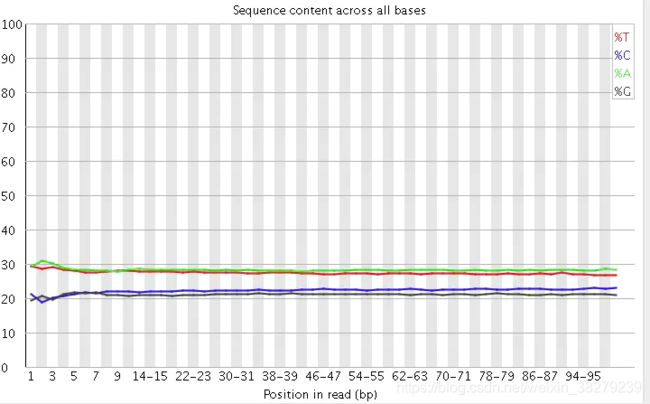
read碱基质量值分布
使用现成的工具将read的质量值和碱基的测序错误率画图更直观地表示出来。用得最广的FastQC,它是一个java程序,能够用于给出测序数据的QC报告,报告中会同时给出上述几个方面的数据图,并提示原来的数据可能还存在着哪些问题。
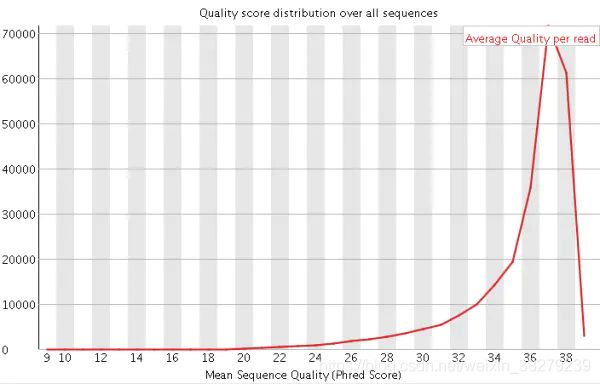
在做read质量值分析的时候,FastQC并不单独查看具体某一条read中碱基的质量值,而是将Fastq文件中所有的read数据都综合起来一起分析。下图是一个测序质量非常好的read各位置碱基质量分布图(如下图)。
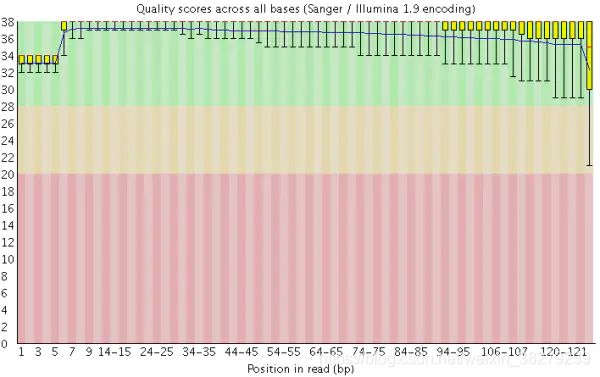
这个图的横轴是read上碱基的位置,纵轴是碱基质量值。在这个例子中,read的长度是126bp(来自HiSeq X10的测序结果),这应该算是比较长的二代测序序列了。我们可以看到read上的每一个位置都有一个黄色的箱型图表示在该位置上所有碱基的质量分布情况。除了最后一个碱基之外,其他的碱基质量值都基本都在大于30,而且波动很小,说明质量很稳定,这其实是一个非常高质量的结果。而且我们可以看到图中质量值的分布都在绿色背景(代表高质量)的区域。
质量很差的结果如下:
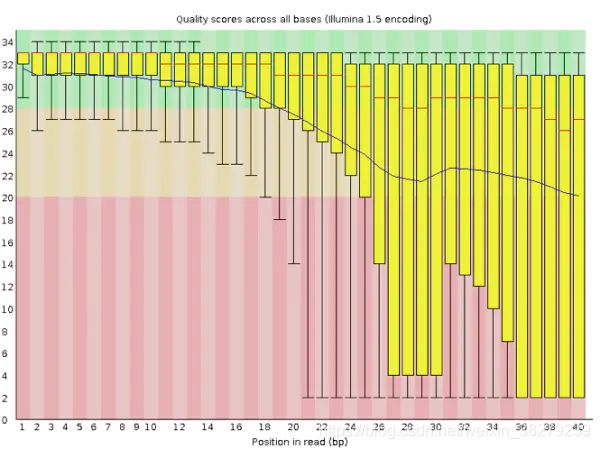
在这个图中可以明显看到,read各个位置上的碱基质量分布波动都比较大,特别从第18个碱基往后全部出现了大幅度的波动,而且很多read的碱基质量值都掉到非常低(红色)的区域中了,说明这个数据的测序结果真的非常差,有着大量不及格的read。最好的情况是重新测序,但如果不得不使用这个数据,就要把这些低质量的数据全都去除掉才行,同时还需留意是否还存在其他的问题,但不管如何都一定会丢掉很大一部分的数据。
FastQC其他统计结果
Q20和Q30的比例是衡量测序质量的一个重要指标。一般来说,对于二代测序,最好是达到Q20的碱基要在95%以上(最差不低于90%),Q30要求大于85%(最差也不要低于80%)。
这个是为了分析碱基的分离程度。何为碱基分离?已知AT配对,CG配对,假如测序过程是比较随机的话(随机意味着好),那么在每个位置上A和T比例应该差不多,C和G的比例也应该差不多,如上图所示,两者之间即使有偏差也不应该太大,最好平均在1%以内,如果过高,除非有合理的原因,比如某些特定的捕获测序所致,否则都需要注意是不是测序过程有什么偏差。
GC含量指的是G和C这两种碱基占总碱基的比例。二代测序平台或多或少都存在一定的测序偏向性,可以通过查看这个值来协助判断测序过程是否足够随机。
对于人类来说,基因组的GC含量一般在40%左右。因此,如果发现GC含量的图谱明显偏离这个值那么说明测序过程存在较高的序列偏向性,结果就是基因组中某些特定区域被反复测序的几率高于平均水平,除了覆盖度会有偏离之后,将会影响下游的变异检测和CNV分析。
N在测序数据中一般是不应该出现的,如果出现则意味着,测序的光学信号无法被清晰分辨,如果这种情况多的话,往往意味着测序系统或者测序试剂的错误。
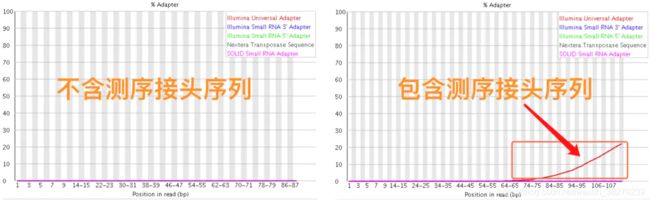
在测序之前需要构建测序文库,测序接头就是在这个时候加上的,其目的一方面是为了能够结合到flowcell上,另一方面是当有多个样本同时测序的时候能够利用接头信息进行区分。当测序read的长度大于被测序的DNA片段【注】时,就会在read的末尾测到这些接头序列(如下图)。
一般的WGS测序是不会测到这些接头序列的,因为构建WGS测序的文库序列(插入片段)都比较长,约几百bp,而read的测序长度都在100bp-150bp这个范围。不过在进行一些RNA测序的时候,由于它们的序列本来就比较短,很多只有几十bp长(特别是miRNA),那么就很容易会出现read测通的现象,这个时候就会在read的末尾测到这些接头序列。
【注】这些DNA片段也常被我们称之为“插入片段”
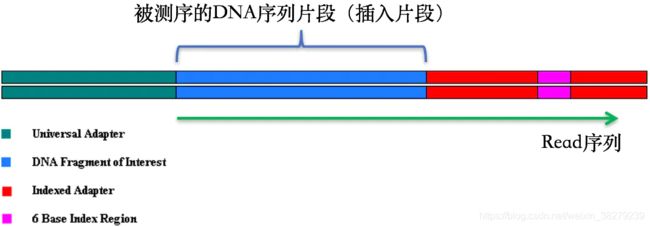
最后,这些被测到的接头序列和低质量碱基一样都是需要在正式分析之前进行切除的read片段。
FastQC的使用
FastQC的安装
$ wget https://www.bioinformatics.babraham.ac.uk/projects/fastqc/fastqc_v0.11.5.zip ./
解压之后,修改文件夹中fastqc的权限,就可以直接运行了
$ unzip fastqc_v0.11.5.zip
$ cd FastQC
$ chmod 755 fastqc
FastQC的运行
直接在终端通过命令行是最有效直接的
$ /path_to_fastqc/FastQC/fastqc untreated.fq -o fastqc_out_dir/
命令比较简单,这里唯一值得注意的地方就是 -o 参数用于指定FastQC报告的输出目录,这个目录需要事先创建好,如果不指定特定的目录,那么FastQC的结果会默认输出到文件untreated.fq的同一个目录下。它输出结果只有两个,一个html和一个.zip压缩包。
$ tree fastqc_out_dir/
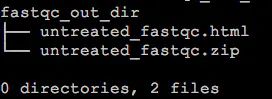
可以直接通过浏览器打开html,就可以看到FastQC给出的所有结果,zip压缩包解压后,从中我们也可以在对应的目录下找到所有的QC图表和Summary数据。
![]()
除了上述用法之外,FastQC支持同时输入多个fq文件(或者以通配符的形式输入fq),当我们的fq文件比较多时,这种用法会比较方便,如:
$ /path_to_fastqc/FastQC/fastqc /path_to_fq/*.fq -o fastqc_out_dir/
切除测序接头序列和read的低质量序列
理解了fq数据之后,做这些过滤就不会很难,完全可以自己编写工具来进行个性化的过滤。目前也已有很多工具用来切除接头序列和低质量碱基,比如SOAPnuke、cutadapt、untrimmed等不下十个,但这其中比较方便好用的是Trimmomatic(也是一个java程序)、sickle和seqtk。
Trimmomatic的好处在于,它不但可以用来切除illumina测序平台的接头序列,还可以去除由我们自己指定的特定接头序列,而且同时也能够过滤read末尾的低质量序列,sickle和seqtk只能去除低质量碱基。具体的原理就是通过滑动一定长度的窗口,计算窗口内的碱基平均质量,如果过低,就直接往后全部切除【注】并不是去掉read中的这部分低质量序列,而是像切菜一样,直接从低质量区域开始把这条read后面的所有其它碱基全部去掉。否则就是在人为改变实际的基因组序列情况。
除了trimmomatic之外,也可以直接使用sickle(同时支持PE和SE数据)或者seqtk(仅支持SE),这两个处理起来会更快,消耗的计算资源也更少。
如何使用Trimmomatic构造序列过滤流程
安装Trimmomatic
我们可以到它的官网上获取最新的版本,下载打包好的binary即可,如果打算看它具体的代码,可以在github上找到

下载后,直接解压,目录下的trimmomatic-*.jar(我下载的是0.36版本)就是执行程序,可以直接使用java来运行。
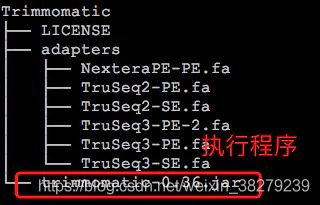
$ java -jar trimmomatic-0.36.jar
同个目录下还有一个名为adapters的文件夹,这个文件夹中的内容对于我们去除接头序列来说非常重要。其中默认存放的是illumina测序平台的接头序列(fasta格式),在实际的使用过程中,如果需要去除接头,需要明确指定对应的序列作为输入参数。
那么这些接头序列具体该如何选择呢?一般来说,目前的HiSeq系列和MiSeq系列用的都是TruSeq3,TruSeq2是以前GA2系列的测序仪所用的,已经很少见了。这些信息都可以在illumina的官网上找到,至于具体该用PE(Pair End)还是SE(Single End)就按照具体的测序类型进行选择即可。如果用的不是illumina测序平台,那么也可以按照adapters文件夹下的这些文件的格式做一个新的接头序列,然后再作为参数传入。不过在自定义接头序列的时候,命名时有一些小的细节需要注意,可以参考Trimmomatic的主页文档(The Adapter Fasta)。
Trimmomatic有两种运行模式:PE和SE。顾名思义,PE就是对应Pair End测序的,SE则是对应Single End测序的。
$ java -jar trimmomatic-0.36.jar
Usage:
PE [-version] [-threads <threads>] [-phred33|-phred64] [-trimlog <trimLogFile>] [-quiet] [-validatePairs] [-basein <inputBase> | <inputFile1> <inputFile2>] [-baseout <outputBase> | <outputFile1P> <outputFile1U> <outputFile2P> <outputFile2U>] <trimmer1>...
or:
SE [-version] [-threads <threads>] [-phred33|-phred64] [-trimlog <trimLogFile>] [-quiet] <inputFile> <outputFile> <trimmer1>...
or:
-version
PE模式,HiSeq PE测序
$ java -jar /path/Trimmomatic/trimmomatic-0.36.jar PE -phred33 -trimlog logfile reads_1.fq.gz reads_2.fq.gz out.read_1.fq.gz out.trim.read_1.fq.gz out.read_2.fq.gz out.trim.read_2.fq.gz ILLUMINACLIP:/path/Trimmomatic/adapters/TruSeq3-PE.fa:2:30:10 SLIDINGWINDOW:5:20 LEADING:5 TRAILING:5 MINLEN:50
SE模式,HiSeq SE测序
$ java -jar /path/Trimmomatic/trimmomatic-0.36.jar SE -phred33 -trimlog se.logfile raw_data/untreated.fq out.untreated.fq.gz ILLUMINACLIP:/path/Trimmomatic/adapters/TruSeq3-SE.fa:2:30:10 SLIDINGWINDOW:5:20 LEADING:5 TRAILING:5 MINLEN:50
同时需要明确指明质量值体系是Phred33还是Phred64,默认是Phred64,这需要特别注意,因为现在的测序数据基本都是Phred33的了,所以一定要指定这个参数。
剩下的就是输入的fq和输出的fq,可以用-basein和-baseout指定,也可以不用(如上例子),以及被过滤掉的fq要输出到文件。
这里PE和SE有一个区别:
在SE模式中,是不需要指定文件来存放被过滤掉的read信息的,后面直接就接Trimmer信息!这是需要注意到的一个地方。
后面的Trimmer信息,规定了很多切除接头序列和低质量序列的细节。如下:
ILLUMINACLIP,接头序列切除参数。LLUMINACLIP:TruSeq3-PE.fa:2:30:10(省掉了路径)意思分别是:TruSeq3-PE.fa是接头序列,2是比对时接头序列时所允许的最大错配数;30指的是要求PE的两条read同时和PE的adapter序列比对,匹配度加起来超30%,那么就认为这对PE的read含有adapter,并在对应的位置需要进行切除【注】。10和前面的30不同,它指的是,我就什么也不管,反正只要这条read的某部分和adpater序列有超过10%的匹配率,那么就代表含有adapter了,需要进行去除;
【注】测序的时候一般只会测到一部分的adapter,因此read和adaper对比的时候肯定是不需要要求百分百匹配率的,上述30%和10%其实是比较推荐的值。
SLIDINGWINDOW,滑动窗口长度的参数,SLIDINGWINDOW:5:20代表窗口长度为5,窗口中的平均质量值至少为20,否则会开始切除;
LEADING,规定read开头的碱基是否要被切除的质量阈值;
TRAILING,规定read末尾的碱基是否要被切除的质量阈值;
MINLEN,规定read被切除后至少需要保留的长度,如果低于该长度,会被丢掉。
另一个值得注意的地方是,Trimmomatic的报错给出的提示信息都比较难以定位错误问题(如下图),但这往往都只是参数用没设置正确所致。
Exception in thread "main" java.lang.RuntimeException: Unknown trimmer: u.trim.txt
at org.usadellab.trimmomatic.trim.TrimmerFactory.makeTrimmer(TrimmerFactory.java:70)
at org.usadellab.trimmomatic.Trimmomatic.createTrimmers(Trimmomatic.java:59)
at org.usadellab.trimmomatic.TrimmomaticSE.run(TrimmomaticSE.java:303)
at org.usadellab.trimmomatic.Trimmomatic.main(Trimmomatic.java:85)
3.2 数据预处理
1. 序列比对
NGS测序下来的短序列(read)存储于FASTQ文件里面。虽然它们原本都来自于有序的基因组,但在经过DNA建库和测序之后,文件中不同read之间的前后顺序关系就已经全部丢失了。因此,FASTQ文件中紧挨着的两条read之间没有任何位置关系,它们都是随机来自于原本基因组中某个位置的短序列而已。
因此,需要先把这一大堆的短序列捋顺,一个个去跟该物种的参考基因组【注】比较,找到每一条read在参考基因组上的位置,然后按顺序排列好,这个过程就称为测序数据的比对。这 也是核心流程真正意义上的第一步,只有完成了这个序列比对我们才有下一步的数据分析。
【注】参考基因组:指该物种的基因组序列,是已经组装成的完整基因组序列,常作为该物种的标准参照物,比如人类基因组参考序列,fasta格式。
序列比对本质上是一个寻找最大公共子字符串的过程。大家如果有学过生物信息学的话,应该或多或少知道BLAST,它使用的是动态规划的算法来寻找这样的子串,但在面对巨量的短序列数据时,类似BLAST这样的软件实在太慢了!因此,需要更加有效的数据结构和相应的算法来完成这个搜索定位的任务。
这里将用于流程构建的BWA就是其中最优秀的一个,它将BW(Burrows-Wheeler)压缩算法和后缀树相结合,能够让我们以较小的时间和空间代价,获得准确的序列比对结果。
序列比对流程搭建
- 为参考基因组构建索引
首先,我们需要为参考基因组的构建索引——这其实是在为参考序列进行Burrows Wheeler变换(wiki: 块排序压缩),以便能够在序列比对的时候进行快速的搜索和定位。
$ bwa index human.fasta
以我们人类的参考基因组(3Gb长度)为例,这个构造过程需要消耗几个小时的时间(一般3个小时左右)。完成之后,你会看到类似如下几个以human.fasta为前缀的文件:
.
├── human.fasta.amb
├── human.fasta.ann
├── human.fasta.bwt
├── human.fasta.pac
└── human.fasta.sa
这些就是在比对时真正需要被用到的文件。这一步完成之后,我们就可以将read比对至参考基因组。
- read比对参考基因组
$ bwa mem -t 4 -R '@RG\tID:foo_lane\tPL:illumina\tLB:library\tSM:sample_name' /path/to/human.fasta read_1.fq.gz read_2.fq.gz > sample_name.sam
这里调用的是bwa的mem比对模块,在解释这样做之前,不妨先看一下bwa mem的官方用法说明,它就一句话:
Usage: bwa mem [options] <idxbase> <in1.fq> [in2.fq]
其中[options]是一系列可选的参数,暂时不多说。这里的 < idxbase>要输入的是参考基因组的BW索引文件,我们上面通过bwa index构建好的那几个以human.fasta为前缀的文件便是;其中< in1.fq>和 [in2.fq]输入的是质控后的fastq文件。但这里输入的时候为什么会需要两个fq(in1.fq和in2.fq)呢?我们上面的例子也是有两个:read_1.fq.gz和read_2.fq.gz。这是因为这是双末端测序(也称Pair-End)的情况。
双末端测序
那什么是“双末端测序”呢?这两个fq之间的关系又是什么?这个我需要简单解析一下。
已经知道NGS是短读长的测序技术,一次测出来的read的长度都不会太长,那为了尽可能把一个DNA序列片段尽可能多地测出来,既然测一边不够,那就测两边,于是就有了一种从被测DNA序列两端各测序一次的模式,这就被称为双末端测序(Pair-End Sequencing,简称PE测序)。
如下图是Pair-End测序的示意图,中间灰色的是被测序的DNA序列片段,左边黄色带箭头和右边蓝色带箭头的分别是测序出来的read1和read2序列,这里假定它们的长度都是100bp。虽然很多时候Pair-End测序还是无法将整个被测的DNA片段完全测通,但是它依然提供了极其有用的信息,比如,我们知道每一对的read1和read2都来自于同一个DNA片段,read1和read2之间的距离是这个DNA片段的长度,而且read1和read2的方向刚好是相反的(这里排除mate-pair的情形)等,这些信息对于后面的变异检测等分析来说都是非常有用的。
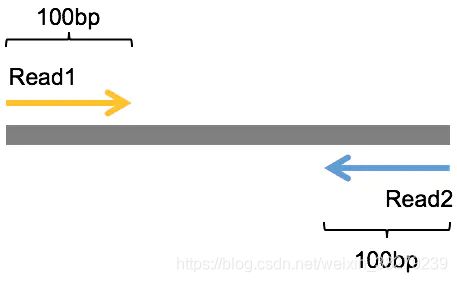
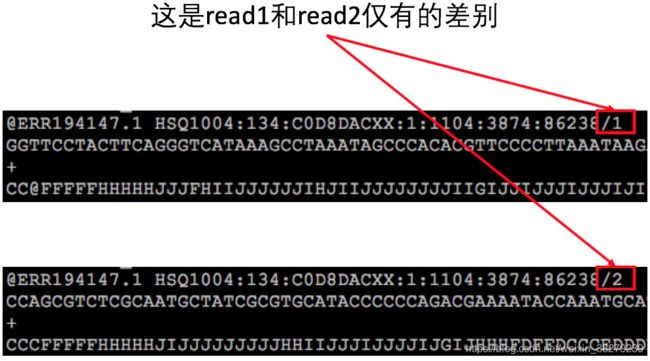
另外,在read1在fq1文件中位置和read2在fq2文件中的文件中的位置是相同的,而且read ID之间只在末尾有一个’/1’或者’/2’的差别。
单末端测序
既然有双末端测序,那么与之对应的就有单末端测序(Single End Sequecing,简称SE测序),即只测序其中一端。因此,我们在使用bwa比对的时候,实际上,in2.fq是非强制性的(所以用方括号括起来),只有是双末端测序的数据时才需要添加。
回到上面我们的例子,大伙可以看到我这里除了用法中提到的参数之外,还多了2个额外的参数,分别是:-t,线程数,我们在这里使用4个线程;-R 接的是 Read Group的字符串信息,这是一个非常重要的信息,以@RG开头,它是用来将比对的read进行分组的。不同的组之间测序过程被认为是相互独立的,这个信息对于我们后续对比对数据进行错误率分析和Mark duplicate时非常重要。在Read Group中,有如下几个信息非常重要:
1.ID,这是Read Group的分组ID,一般设置为测序的lane ID(不同lane之间的测序过程认为是独立的),下机数据中我们都能看到这个信息的,一般都是包含在fastq的文件名中;
- PL,指的是所用的测序平台,这个信息不要随便写!特别是当我们需要使用GATK进行后续分析的时候,更是如此!这是一个很多新手都容易忽视的一个地方,在GATK中,PL只允许被设置为:ILLUMINA,SLX,SOLEXA,SOLID,454,LS454,COMPLETE,PACBIO,IONTORRENT,CAPILLARY,HELICOS或UNKNOWN这几个信息。基本上就是目前市场上存在着的测序平台,当然,如果实在不知道,那么必须设置为UNKNOWN,名字方面不区分大小写。如果你在分析的时候这里没设置正确,那么在后续使用GATK过程中可能会碰到类似如下的错误:
ERROR MESSAGE: The platform (xx) associated with read group GATKSAMReadGroupRecord @RG:xx is not a recognized platform.
- 这个时候你需要对比对文件的header信息进行重写,就会稍微比较麻烦。
我们上面的例子用的是PL:illumina。如果你的数据是CG测序的那么记得不要写成CG!而要写COMPLETE。
- SM,样本ID,同样非常重要,有时候我们测序的数据比较多的时候,那么可能会分成多个不同的lane分布测出来,这个时候SM名字就是可以用于区分这些样本。
- LB,测序文库的名字,这个重要性稍微低一些,主要也是为了协助区分不同的group而存在。文库名字一般可以在下机的fq文件名中找到,如果上面的lane ID足够用于区分的话,也可以不用设置LB;
除了以上这四个之外,还可以自定义添加其他的信息,不过如无特殊的需要,对于序列比对而言,这4个就足够了。这些信息设置好之后,在RG字符串中要用制表符(\t)将它们分开。
最后在我们的例子中,我们将比对的输出结果直接重定向到一份sample_name.sam文件中,这类文件是BWA比对的标准输出文件,它的具体格式我会在下一篇文章中进行详细说明。但SAM文件是文本文件,一般整个文件都非常巨大,因此,为了有效节省磁盘空间,一般都会用samtools将它转化为BAM文件(SAM的特殊二进制格式),而且BAM会更加方便于后续的分析。所以我们上面比对的命令可以和samtools结合并改进为:
$ bwa mem -t 4 -R '@RG\tID:foo_lane\tPL:illumina\tLB:library\tSM:sample_name' /path/to/human.fasta read_1.fq.gz read_2.fq.gz | samtools view -S -b - > sample_name.bam
通过管道(“|”)把比对的输出如同引导水流一样导流给samtools去处理,上面samtools view的-b参数指的就是输出为BAM文件,这里需要注意的地方是-b后面的’-’,它代表就是上面管道引流过来的数据,经过samtools转换之后我们再重定向为sample_name.bam。
[Tips] BWA MEM比对模块是有一定适用范围的:它是专门为长read比对设计的,目的是为了解决,第三代测序技术这种能够产生长达几十kb甚至几Mbp的read情况。一般只有当read长度≥70bp的时候,才推荐使用,如果比这个要小,建议使用BWA ALN模块。
2. 排序
排序这一步我们也是通过使用samtools来完成的,命令很简单:
Usage: samtools sort [options...] [in.bam]
执行之前,我们有必要先搞明白为什么需要排序,为什么BWA比对后输出的BAM文件是没顺序的!原因就是FASTQ文件里面这些被测序下来的read是随机分布于基因组上面的,第一步的比对是按照FASTQ文件的顺序把read逐一定位到参考基因组上之后,随即就输出了,它不会也不可能在这一步里面能够自动识别比对位置的先后位置重排比对结果。因此,比对后得到的结果文件中,每一条记录之间位置的先后顺序是乱的,我们后续去重复等步骤都需要在比对记录按照顺序从小到大排序下来才能进行,所以这才是需要进行排序的原因。对于我们的例子来说,这个排序的命令如下:
$ time samtools sort -@ 4 -m 4G -O bam -o sample_name.sorted.bam sample_name.bam
其中,-@,用于设定排序时的线程数,我们设为4;-m,限制排序时最大的内存消耗,这里设为4GB;-O 指定输出为bam格式;-o 是输出文件的名字,这里叫sample_name.sorted.bam。比较建议在做类似分析的时候在文件名字将所做的关键操作包含进去,因为这样即使过了很长时间,当你再去看这个文件的时候也能够立刻知道当时对它做了什么;最后就是输入文件——sample_name.bam。
【注意】排序后如果发现新的BAM文件比原来的BAM文件稍微小一些,不用觉得惊讶,这是压缩算法导致的结果,文件内容是没有损失的
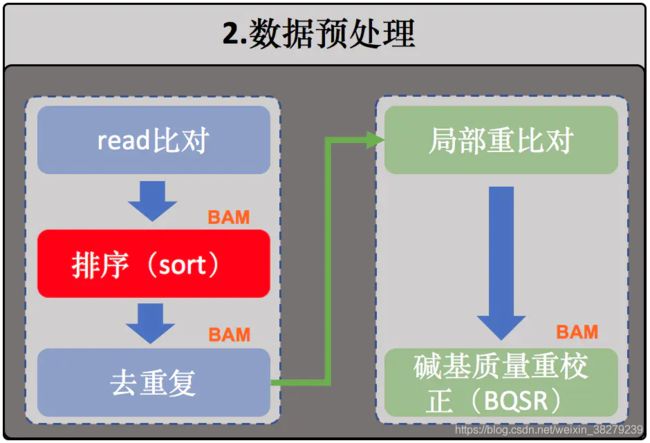
3. 去除重复序列(或者标记重复序列)
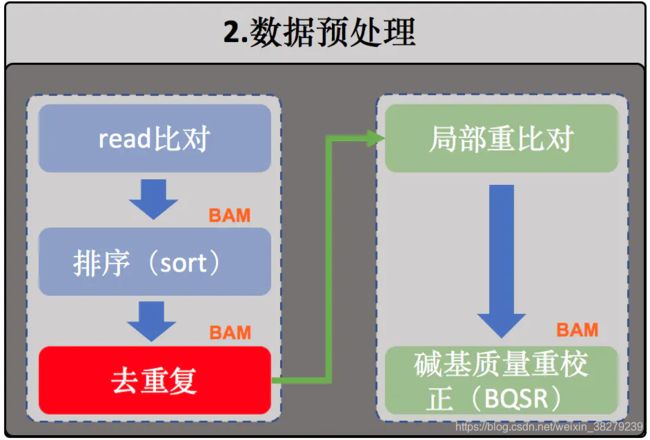
在排序完成之后我们就可以开始执行去除重复(准确来说是 去除PCR重复序列)的步骤了。
在NGS测序之前都需要先构建测序文库:通过物理(超声)打断或者化学试剂(酶切)切断原始的DNA序列,然后选择特定长度范围的序列去进行PCR扩增并上机测序。
因此,这里重复序列的来源实际上就是由PCR过程中所引入的。因为所谓的PCR扩增就是把原来的一段DNA序列复制多次。可是为什么需要PCR扩增呢?如果没有扩增不就可以省略这一步了吗?
情况确实如此,但是很多时候我们构建测序文库时能用的细胞量并不会非常充足,而且在打断的步骤中也会引起部分DNA的降解,这两点会使整体或者局部的DNA浓度过低,这时如果直接从这个溶液中取样去测序就很可能漏掉原本基因组上的一些DNA片段,导致测序不全。
而PCR扩增的作用就是为了把这些微弱的DNA多复制几倍乃至几十倍,以便增大它们在溶液中分布的密度,使得能够在取样时被获取到。
所以这里大家需要记住一个重点,PCR扩增原本的目的是为了增大微弱DNA序列片段的密度,但由于整个反应都在一个试管中进行,因此其他一些密度并不低的DNA片段也会被同步放大,那么这时在取样去上机测序的时候,这些DNA片段就很可能会被重复取到相同的几条去进行测序(下图为PCR扩增示意图)。
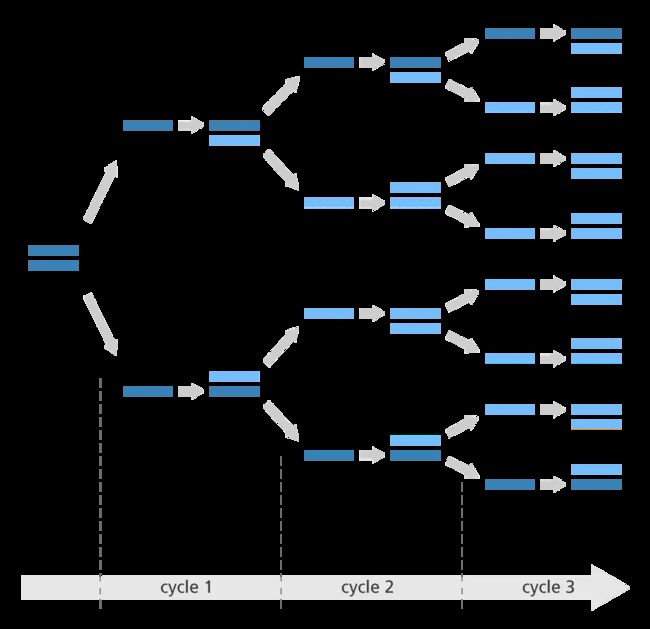
PCR扩增是一个指数扩增的过程,图中原本只有一段双链DNA序列,在经过3轮PCR后就被扩增成了8段。PCR扩增会对结果有影响,而且有时影响会很大!最直接的后果就是同时增大了变异检测结果的假阴和假阳率。主要有几个原因:
- DNA在打断的那一步会发生一些损失,主要表现是会引发一些碱基发生颠换变换(嘌呤-变嘧啶或者嘧啶变嘌呤),带来假的变异。PCR过程会扩大这个信号,导致最后的检测结果中混入了假的结果;
- PCR反应过程中也会带来新的碱基错误。发生在前几轮的PCR扩增发生的错误会在后续的PCR过程中扩大,同样带来假的变异;
- 对于真实的变异,PCR反应可能会对包含某一个碱基的DNA模版扩增更加剧烈(这个现象称为PCR Bias)。如果反应体系是对含有reference allele的模板扩增偏向强烈,那么变异碱基的信息会变小,从而会导致假阴。
PCR对真实的变异检测和个体的基因型判断都有不好的影响。GATK、Samtools、Platpus等这种利用贝叶斯原理的变异检测算法都是认为所用的序列数据都不是重复序列(即将它们和其他序列一视同仁地进行变异的判断,所以带来误导),因此必须要进行标记(去除)或者使用PCR-Free的测序方案(这个方案目前正变得越来越流行,特别是对于RNA-Seq来说尤为重要,现在著名的基因组学研究所——Broad Institute,基本都是使用PCR-Free的测序方案)。
那么具体是如何做到去除这些PCR重复序列的呢?我们可以抛开任何工具,仔细想想,既然PCR扩增是把同一段DNA序列复制出很多份,那么这些序列在经过比对之后它们一定会定位到基因组上相同的位置,比对的信息看起来也将是一样的!于是,我们就可以根据这个特点找到这些重复序列了!
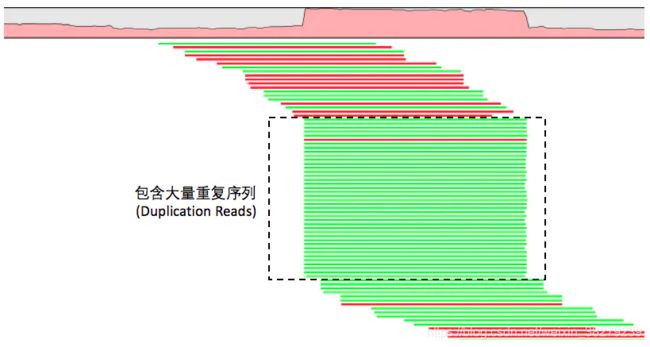
事实上,现有的工具包括Samtools和Picard中去除重复序列的算法也的确是这么做的。不同的地方在于,samtools的rmdup是直接将这些重复序列从比对BAM文件中删除掉,而Picard的MarkDuplicates默认情况则只是在BAM的FLAG信息中标记出来,而不是删除,因此这些重复序列依然会被留在文件中,只是我们可以在变异检测的时候识别到它们,并进行忽略。
考虑到尽可能和现在主流的做法一致(但并不是说主流的做法就一定是对的,要分情况看待,只是主流的做法容易被做成生产流程而已),这里也用Picard来完成这个事情:
java -jar picard.jar MarkDuplicates \
I=sample_name.sorted.bam \
O=sample_name.sorted.markdup.bam \
M=sample_name.markdup_metrics.txt
这里只把重复序列在输出的新结果中标记出来,但不删除。如果非要把这些序列完全删除的话可以这样做:
java -jar picard.jar MarkDuplicates \
REMOVE_DUPLICATES=true \
I=sample_name.sorted.bam \
O=sample_name.sorted.markdup.bam \
M=sample_name.markdup_metrics.txt
把参数REMOVE_DUPLICATES设置为ture,那么重复序列就被删除掉,不会在结果文件中留存。我比较建议使用第一种做法,只是标记出来,并留存这些序列,以便在你需要的时候还可以对其做分析。
这一步完成之后,需要为sample_name.sorted.markdup.bam创建索引文件,它的作用能够让我们可以随机访问这个文件中的任意位置,而且后面的“局部重比对”步骤也要求这个BAM文件一定要有索引,命令如下:
$ samtools index sample_name.sorted.markdup.bam
完成之后,会生成一份sample_name.sorted.markdup.bam.bai文件,这就是上面这份BAM的index。
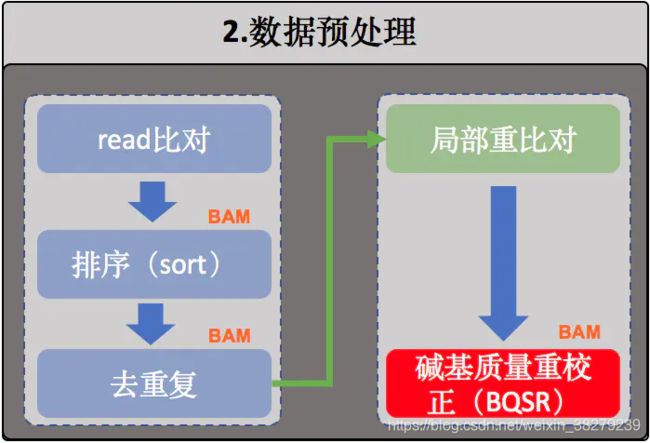
4. 局部重比对
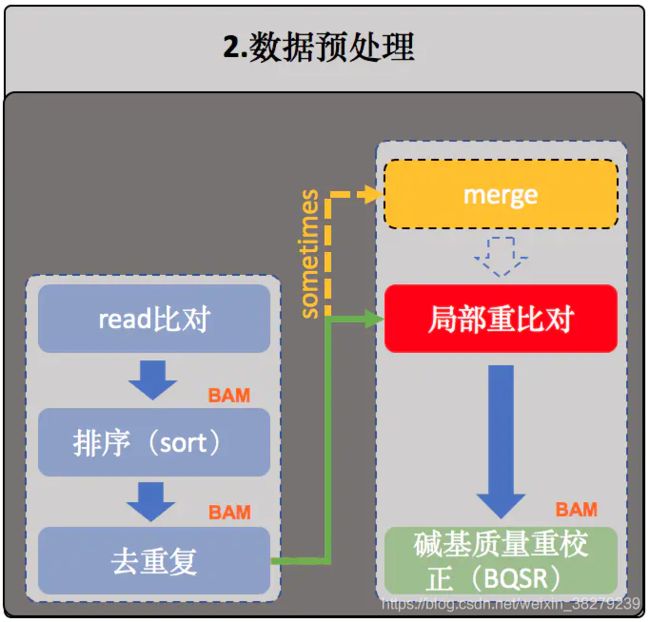
接下来是局部区域重比对,通常也叫Indel局部区域重比对。有时在进行这一步骤之前还有一个merge的操作,将同个样本的所有比对结果合并成唯一一个大的BAM文件【注】,merge的例子如下:
$ samtools merge <out.bam> <in1.bam> [<in2.bam> ... <inN.bam>]
【注意】之所以会有这种情况,是因为有些样本测得非常深,其测序结果需要经过多次测序(或者分布在多个不同的测序lane中)才全部获得,这个时候我们一般会先分别进行比对并去除重复序列后再使用samtools进行合并。
局部重比对的目的是将BWA比对过程中所发现有 潜在序列插入或者序列删除(insertion和deletion,简称Indel)的区域进行重新校正。这个过程往往还会把一些已知的Indel区域一并作为重比对的区域,但为什么需要进行这个校正呢?
其根本原因来自于参考基因组的序列特点和BWA这类比对算法本身,注意这里不是针对BWA,而是针对所有的这类比对算法,包括bowtie等。这类在全局搜索最优匹配的算法在存在Indel的区域及其附近的比对情况往往不是很准确,特别是当一些存在长Indel、重复性序列的区域或者存在长串单一碱基(比如,一长串的TTTT或者AAAAA等)的区域中更是如此。
另一个重要的原因是在这些比对算法中,对碱基错配和开gap的容忍度是不同的。具体体现在罚分矩阵的偏向上,例如,在read比对时,如果发现碱基错配和开gap都可以的话,它们会更偏向于错配。但是这种偏向错配的方式,有时候却还会反过来引起错误的开gap!这就会导致基因组上原本应该是一个长度比较大的Indel的地方,被错误地切割成多个错配和短indel的混合集,这必然会让我们检测到很多错误的变异。而且,这种情况还会随着所比对的read长度的增长(比如三代测序的Read,通常都有几十kbp)而变得越加严重。
因此,我们需要有一种算法来对这些区域进行局部的序列重比对。这个算法通常就是大名鼎鼎的Smith-Waterman算法,它非常适合于这类场景,可以极其有效地实现对全局比对结果的校正和调整,最大程度低地降低由全局比对算法的不足而带来的错误。而且GATK的局部重比对模块,除了应用这个算法之外,还会对这个区域中的read进行一次局部组装,把它们连接成为长度更大的序列,这样能够更进一步提高局部重比对的准确性。
下图给大家展示一个序列重比对之前和之后的结果,其中灰色的横条指的是read,空白黑线指的是deletion,有颜色的碱基指的是错配碱基。

可以明显地看到在序列重比对之前,在这个区域的比对数据是多么的糟糕,如果就这样进行变异检测,那么一定会得到很多假的结果。而在经过局部重比对之后,这个区域就变得非常清晰而分明,它原本发生的就只是一个比较长的序列删除(deletion)事件,但在原始的比对结果中却被错误地用碱基错配和短的Indel所代替。
具体该怎么做呢?我们的WGS分析流程从这个步骤开始就需要用到GATK (GenomeAnalysisTK.jar)了,我们的执行命令如下:
java -jar /path/to/GenomeAnalysisTK.jar \
-T RealignerTargetCreator \
-R /path/to/human.fasta \
-I sample_name.sorted.markdup.bam \
-known /path/to/gatk/bundle/1000G_phase1.indels.b37.vcf \
-known /path/to/gatk/bundle/Mills_and_1000G_gold_standard.indels.b37.vcf \
-o sample_name.IndelRealigner.intervals
java -jar /path/to/GenomeAnalysisTK.jar \
-T IndelRealigner \
-R /path/to/human.fasta \
-I sample_name.sorted.markdup.bam \
-known /path/to/gatk/bundle/1000G_phase1.indels.b37.vcf \
-known /path/to/gatk/bundle/Mills_and_1000G_gold_standard.indels.b37.vcf \
-o sample_name.sorted.markdup.realign.bam \
--targetIntervals sample_name.IndelRealigner.intervals
这里包含了两个步骤:
- 第一步,RealignerTargetCreator ,目的是定位出所有需要进行序列重比对的目标区域(如下图);
- 第二步,IndelRealigner,对所有在第一步中找到的目标区域运用算法进行序列重比对,最后得到捋顺了的新结果。
![]()
以上这两个步骤是缺一不可的,顺序也是固定的。而且,需要指出的是,这里的-R参数输入的human.fasta不是BWA比对中的索引文件前缀,而是参考基因组序列(FASTA格式)文件,下同。
另外,在重比对步骤中,我们还看到了两个陌生的VCF文件,分别是:1000G_phase1.indels.b37.vcf和Mills_and_1000G_gold_standard.indels.b37.vcf。这两个文件来自于千人基因组和Mills项目,里面记录了那些项目中检测到的人群Indel区域。我上面其实也提到了,候选的重比对区除了要在样本自身的比对结果中寻找之外,还应该把人群中已知的Indel区域也包含进来,而这两个是我们在重比对过程中最常用到的。这些文件你可以很方便地在GATK bundle ftp中下载,注意一定要选择和你的参考基因组对应的版本,我们这里用的是b37版本。
那么既然Indel局部重比对这么好,这么重要,似乎看起来在任何情况下都应该是必须的。然鹅,我的回答是否定的!
但否定是有前提的!那就是我们后面的变异检测必须是使用GATK,而且必须使用GATK的HaplotypeCaller模块,仅当这个时候才可以减少这个Indel局部重比对的步骤。原因是GATK的HaplotypeCaller中,会对潜在的变异区域进行相同的局部重比对!但是其它的变异检测工具或者GATK的其它模块就没有这么干了!所以切记!
5. 重新校正碱基质量值(BQSR)
在WGS分析中,变异检测是一个极度依赖测序碱基质量值的步骤。因为这个质量值是衡量我们测序出来的这个碱基到底有多正确的重要(甚至是唯一)指标。它来自于测序图像数据的base calling。因此,基本上是由测序仪和测序系统来决定的。但不幸的是,影响这个值准确性的系统性因素有很多,包括物理和化学等对测序反应的影响,甚至连仪器本身和周围环境都是其重要的影响因素。当把所有这些东西综合在一起之后,往往会发现计算出来的碱基质量值要么高于真实结果,要么低于真实结果。那么,我们到底该如何才能获得符合真实情况的碱基质量值?
BQSR(Base Quality Score Recalibration)这个步骤就是为此而存在的,这一步同样非常重要。它主要是通过机器学习的方法构建测序碱基的错误率模型,然后对这些碱基的质量值进行相应的调整。
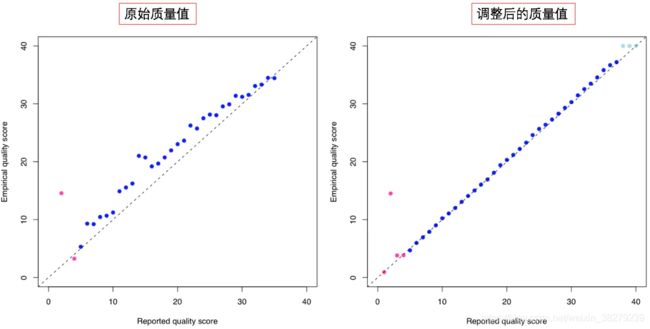
图中,横轴(Reported quality score)是测序结果在Base calling之后报告出来的质量值,也就是我们在FASTQ文件中看到的那些;纵轴(Empirical quality score)代表的是“真实情况的质量值”。
但是且慢,这个“真实情况的质量值”是怎么来的?因为实际上我们并没有办法直接测得它们啊!没错,确实没办法直接测量到,但是我们可以通过统计学的技巧获得极其接近的分布结果(因此我加了引号)。试想一下,如果我们在看到某一个碱基报告的质量值是20时,那么它的预期错误率是1%,反过来想,就等于是说如果有100个质量值都是20的碱基,那么从统计上讲它们中将只有1个是错的!做了这个等效变换之后,我们的问题就可以转变成为寻找错误碱基的数量了。
这时问题就简单多了。我们知道人与人之间的差异其实是很小的,那么在一个群体中发现的已知变异,在某个人身上也很可能是同样存在的。因此,这个时候我们可以对比对结果进行直接分析,首先排除掉所有的已知变异位点,然后计算每个(报告出来的)质量值下面有多少个碱基在比对之后与参考基因组上的碱基是不同的,这些不同碱基就被我们认为是错误的碱基,它们的数目比例反映的就是真实的碱基错误率,换算成Phred score(Phred score的定义可以参考第2节的相关内容)之后,就是纵轴的Empirical quality score了。
上面‘BQSR质量校正对比’的图中左边是原始质量值与真实质量值的比较,在这个图的例子中我们可以发现,base calling给出的质量值并没有正确地反映真实的错误率情况,测序报告出来的碱基质量值大部分被高估了,换句话说,就是错误率被低估了。
在我们的流程中,BQSR的具体执行命令如下:
java -jar /path/to/GenomeAnalysisTK.jar \
-T BaseRecalibrator \
-R /path/to/human.fasta \
-I sample_name.sorted.markdup.realign.bam \
--knownSites /path/to/gatk/bundle/1000G_phase1.indels.b37.vcf \
--knownSites /path/to/gatk/bundle/Mills_and_1000G_gold_standard.indels.b37.vcf \
--knownSites /path/to/gatk/bundle/dbsnp_138.b37.vcf \
-o sample_name.recal_data.table
java -jar /path/to/GenomeAnalysisTK.jar \
-T PrintReads \
-R /path/to/human.fasta \
-I sample_name.sorted.markdup.realign.bam \
--BQSR sample_name.recal_data.table \
-o sample_name.sorted.markdup.realign.BQSR.bam
这里同样包含了两个步骤:
- 第一步,BaseRecalibrator,这里计算出了所有需要进行重校正的read和特征值,然后把这些信息输出为一份校准表文件(sample_name.recal_data.table)
- 第二步,PrintReads,这一步利用第一步得到的校准表文件(sample_name.recal_data.table)重新调整原来BAM文件中的碱基质量值,并使用这个新的质量值重新输出一份新的BAM文件。
注意,因为BQSR实际上是为了(尽可能)校正测序过程中的系统性错误,因此,在执行的时候是按照不同的测序lane或者测序文库来进行的,这个时候@RG信息(BWA比对时所设置的)就显得很重要了,算法就是通过@RG中的ID来识别各个独立的测序过程,这也是我开始强调其重要性的原因。
3.3 变异检测
1. 变异检测
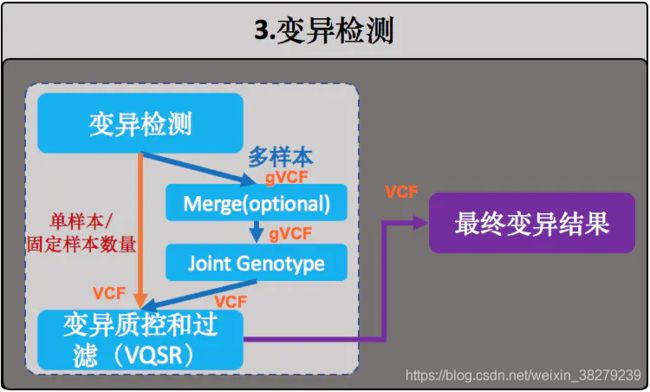
事实上,这是目前所有WGS数据分析流程的一个目标——获得样本准确的变异集合。这里变异检测的内容一般会包括:SNP、Indel,CNV和SV等,这个流程中我们只做其中最主要的两个:SNP和Indel。我们这里使用GATK HaplotypeCaller模块对样本中的变异进行检测,它也是目前最适合用于对二倍体基因组进行变异(SNP+Indel)检测的算法。
HaplotypeCaller和那些直接应用贝叶斯推断的算法有所不同,它会先推断群体的单倍体组合情况,计算各个组合的几率,然后根据这些信息再反推每个样本的基因型组合。因此它不但特别适合应用到群体的变异检测中,而且还能够依据群体的信息更好地计算每个个体的变异数据和它们的基因型组合。
一般来说,在实际的WGS流程中对HaplotypeCaller的应用有两种做法,差别只在于要不要在中间生成一个gVCF:
-
直接进行HaplotypeCaller,这适合于单样本,或者那种固定样本数量的情况,也就是执行一次HaplotypeCaller之后就老死不相往来了。否则你会碰到仅仅只是增加一个样本就得重新运行这个HaplotypeCaller的坑爹情况(即,N+1难题),而这个时候算法需要重新去读取所有人的BAM文件,这将会是一个很费时间的痛苦过程;
-
每个样本先各自生成gVCF,然后再进行群体joint-genotype。这其实就是GATK团队为了解决(1)中的N+1难题而设计出来的模式。gVCF全称是genome VCF,是每个样本用于变异检测的中间文件,格式类似于VCF,它把joint-genotype过程中所需的所有信息都记录在这里面,文件无论是大小还是数据量都远远小于原来的BAM文件。这样一旦新增加样本也不需要再重新去读取所有人的BAM文件了,只需为新样本生成一份gVCF,然后重新执行这个joint-genotype就行了。
我们先以第一种(直接HaplotypeCaller)做法为例子:
java -jar /path/to/GenomeAnalysisTK.jar \
-T HaplotypeCaller \
-R /path/to/human.fasta \
-I sample_name.sorted.markdup.realign.BQSR.bam \
-D /path/to/gatk/bundle/dbsnp_138.b37.vcf \
-stand_call_conf 50 \
-A QualByDepth \
-A RMSMappingQuality \
-A MappingQualityRankSumTest \
-A ReadPosRankSumTest \
-A FisherStrand \
-A StrandOddsRatio \
-A Coverage \
-o sample_name.HC.vcf
这里我特别提一下-D参数输入的dbSNP同样可以再GATK bundle目录中找到,这份文件汇集的是目前几乎所有的公开人群变异数据集。另外,由于我们的例子只有一个样本因此只输入一个BAM文件就可以了,如果有多个样本那么可以继续用-I参数输入:
java -jar GenomeAnalysisTK.jar \
-T HaplotypeCaller \
-R reference.fasta \
-I sample1.bam [-I sample2.bam ...] \
...
以上的命令是直接对全基因组做变异检测,这个过程会消耗很长的时间,通常需要几十个小时甚至几天。
然而,基因组上各个不同的染色体之间其实是可以理解为相互独立的(结构性变异除外),也就是说,为了提高效率我们可以按照染色体一条条来独立执行这个步骤,最后再把结果合并起来就好了,这样的话就能够节省很多的时间。下面我给出一个按照染色体区分的例子:
java -jar /path/to/GenomeAnalysisTK.jar \
-T HaplotypeCaller \
-R /path/to/human.fasta \
-I sample_name.sorted.markdup.realign.BQSR.bam \
-D /path/to/gatk/bundle/dbsnp_138.b37.vcf \
-L 1 \
-stand_call_conf 50 \
-A QualByDepth \
-A RMSMappingQuality \
-A MappingQualityRankSumTest \
-A ReadPosRankSumTest \
-A FisherStrand \
-A StrandOddsRatio \
-A Coverage \
-o sample_name.HC.1.vcf
这里我特别提一下-D参数输入的dbSNP同样可以再GATK bundle目录中找到,这份文件汇集的是目前几乎所有的公开人群变异数据集。另外,由于我们的例子只有一个样本因此只输入一个BAM文件就可以了,如果有多个样本那么可以继续用-I参数输入:
java -jar GenomeAnalysisTK.jar \
-T HaplotypeCaller \
-R reference.fasta \
-I sample1.bam [-I sample2.bam ...] \
...
以上的命令是直接对全基因组做变异检测,这个过程会消耗很长的时间,通常需要几十个小时甚至几天。
然而,基因组上各个不同的染色体之间其实是可以理解为相互独立的(结构性变异除外),也就是说,为了提高效率我们可以按照染色体一条条来独立执行这个步骤,最后再把结果合并起来就好了,这样的话就能够节省很多的时间。下面我给出一个按照染色体区分的例子:
java -jar /path/to/GenomeAnalysisTK.jar \
-T HaplotypeCaller \
-R /path/to/human.fasta \
-I sample_name.sorted.markdup.realign.BQSR.bam \
-D /path/to/gatk/bundle/dbsnp_138.b37.vcf \
-L 1 \
-stand_call_conf 50 \
-A QualByDepth \
-A RMSMappingQuality \
-A MappingQualityRankSumTest \
-A ReadPosRankSumTest \
-A FisherStrand \
-A StrandOddsRatio \
-A Coverage \
-o sample_name.HC.1.vcf
其它参数都没任何改变,就只增加了一个 -L 参数,通过这个参数我们可以指定特定的染色体(或者基因组区域)!我们这里指定的是 1 号染色体,有些地方会写成chr1,具体看human.fasta中如何命名,与其保持一致即可。其他染色体的做法也是如此,就不再举例了。最后合并:
java -jar /path/to/GenomeAnalysisTK.jar \
-T CombineVariants \
-R /path/to/human.fasta \
--genotypemergeoption UNSORTED \
--variant sample_name.HC.1.vcf \
--variant sample_name.HC.2.vcf \
...
--variant sample_name.HC.MT.vcf \
-o sample_name.HC.vcf
第二种,先产生gVCF,最后再joint-genotype的做法:
java -jar /path/to/GenomeAnalysisTK.jar \
-T HaplotypeCaller \
-R /path/to/human.fasta \
-I sample_name.sorted.markdup.realign.BQSR.bam \
--emitRefConfidence GVCF \
-o sample_name.g.vcf
#调用GenotypeGVCFs完成变异calling
java -jar /path/to/GenomeAnalysisTK.jar \
-T GenotypeGVCFs \
-R /path/to/human.fasta \
--variant sample_name.g.vcf \
-o sample_name.HC.vcf
其实,就是加了–emitRefConfidence GVCF的参数。而且,假如嫌慢,同样可以按照染色体或者区域去产生一个样本的gVCF,然后在GenotypeGVCFs中把它们全部作为输入文件完成变异calling。也许你会担心同个样本被分成多份gVCF之后,是否会被当作不同的多个样本?回答是不会!因为生成gVCF文件的过程中,GATK会根据@RG信息中的SM(也就是sample name)来判断这些gVCF是否来自同一个样本,如果名字相同,那么就会被认为是同一个样本,不会产生多样本问题。
2. 变异检测质控和过滤(VQSR)
在获得了原始的变异检测结果之后,我们还需要做的就是质控和过滤。这一步或多或少都有着一些个性化的要求,我暂时就不做太多解释吧(一旦解释恐怕同样是一篇万字长文)。只用一句话来概括,VQSR是通过构建GMM模型对好和坏的变异进行区分,从而实现对变异的质控。
下面就直接给出例子吧:
## SNP Recalibrator
java -jar /path/to/GenomeAnalysisTK.jar \
-T VariantRecalibrator \
-R reference.fasta \
-input sample_name.HC.vcf \
-resource:hapmap,known=false,training=true,truth=true,prior=15.0 /path/to/gatk/bundle/hapmap_3.3.b37.vcf \
-resource:omini,known=false,training=true,truth=false,prior=12.0 /path/to/gatk/bundle/1000G_omni2.5.b37.vcf \
-resource:1000G,known=false,training=true,truth=false,prior=10.0 /path/to/gatk/bundle/1000G_phase1.snps.high_confidence.b37.vcf \
-resource:dbsnp,known=true,training=false,truth=false,prior=6.0 /path/to/gatk/bundle/dbsnp_138.b37.vcf \
-an QD -an MQ -an MQRankSum -an ReadPosRankSum -an FS -an SOR -an DP \
-mode SNP \
-recalFile sample_name.HC.snps.recal \
-tranchesFile sample_name.HC.snps.tranches \
-rscriptFile sample_name.HC.snps.plots.R
java -jar /path/to/GenomeAnalysisTK.jar -T ApplyRecalibration \
-R human_g1k_v37.fasta \
-input sample_name.HC.vcf \
--ts_filter_level 99.5 \
-tranchesFile sample_name.HC.snps.tranches \
-recalFile sample_name.HC.snps.recal \
-mode SNP \
-o sample_name.HC.snps.VQSR.vcf
## Indel Recalibrator
java -jar /path/to/GenomeAnalysisTK.jar -T VariantRecalibrator \
-R human_g1k_v37.fasta \
-input sample_name.HC.snps.VQSR.vcf \
-resource:mills,known=true,training=true,truth=true,prior=12.0 /path/to/gatk/bundle/Mills_and_1000G_gold_standard.indels.b37.vcf \
-an QD -an DP -an FS -an SOR -an ReadPosRankSum -an MQRankSum \
-mode INDEL \
-recalFile sample_name.HC.snps.indels.recal \
-tranchesFile sample_name.HC.snps.indels.tranches \
-rscriptFile sample_name.HC.snps.indels.plots.R
java -jar /path/to/GenomeAnalysisTK.jar -T ApplyRecalibration \
-R human_g1k_v37.fasta\
-input sample_name.HC.snps.VQSR.vcf \
--ts_filter_level 99.0 \
-tranchesFile sample_name.HC.snps.indels.tranches \
-recalFile sample_name.HC.snps.indels.recal \
-mode INDEL \
-o sample_name.HC.snps.indels.VQSR.vcf
最后,sample_name.HC.snps.indels.VQSR.vcf便是我们最终的变异检测结果。对于人类而言,一般来说,每个人最后检测到的变异数据大概在400万左右(包括SNP和Indel)。
八、转录组数据分析处理
高通量分析转录组的核心就是选择一种更准确更快的方法对基因表达量进行定量从而找出差异基因。
流程概括
拿到测序数据后,
- 质控;
- 把质控后的序列回帖到参考基因组,
这步就需要考虑回帖的时候用什么软件什么策略更准确; - 回帖到参考基因组后怎样衡量基因表达量是高还是低;
- 找可变剪切找差异表达基因。
1. 转录组的建库与质量控制
1.1 人类基因组与转录组的组成
常用的基因有27000左右个(两万七千个常用的基因)
其中编码蛋白质的有两万个左右;能转录成有功能的RNA的基因有七千个左右
20000个左右蛋白基因+7000个左右RNA gene
RNA有很多种类,rRNA也就是核糖体RNA占80%-90%,剩下的10%-20%绝大多数是tRNA,真正的我们所专注的 mRNA 只占5%左右。
转录组建库的核心
-
转录组建库的核心是把目标RNA(最常见的是mRNA)的比例提高,然后进行扩增,使其浓度达到可以建库测序的要求。
-
单细胞转录组测序的核心,就是通过特殊的扩增方法,只扩增mRNA而不扩增其它种类的RNA,然后进行测序
1.2 转录组的2种建库策略
1.正选择——polyA positive
成熟的mRNA都有polyA尾巴,我们先用Oligo dT(带有一段T序列的磁珠)标签去识别polyA尾巴从而将mRNA富集出来,然后直接打断,再用random primer将mRNA进行反转录得到RNA、DNA杂合体,再用 random 的 primer 对第二条链进行扩增,这时得到DNA和DNA的结构。再用末端修复酶把它补平为平末端然后在它 3’ 末端加一个A,加完A后相当于把平末端变成一个黏性末端,之后加上adapter,就可以上机进行测序了。
2. 负选择——rRNA minus
第一步还是提出 total 的 RNA,我们有18S、28S、5.8S等这些不同种类的 rRNA 的标签,它特异性的结合 rRNA 的序列,这些标签上都有抗体方便我们特异性的识别并把它们取出,此时我们就去除了细胞中含量最多的RNA种类 — rRNA,一般为了保险起见,需要进行两次rRNA minus的去除,然后对剩下的RNA进行打断和建库。
1.3 两种建库方法比较
第一种方法 — polyA positive 只拿成熟的mRNA进行建库,第二种方法 — rRNA minus 除了有不成熟的 mRNA 还有一些 tRNA、microRNA 等等
如果你只想研究成熟的mRNA那肯定选第一种建库策略,如果想研究一些其他种类的RNA或者RNA降解的一些问题,那必须选择第二种建库策略。rRNA minus建库方法可以获得比较多的没有被剪切掉的Introne(内含子)的信息,用它来非常好的衡量 alternative splicing (可变剪切)。
从两种建库策略我们可以看出,第二种建库策略涵盖的信息多,那为什么我们不都选择第二种呢?因为:第一,第二种策略需要更多的测序量;第二,第二种建库策略更贵,贵大概两倍左右。
九、python并发编程
1. 基础知识
线程
线程是一个基本的 CPU 执行单元。它必须依托于进程存活。一个线程是一个execution context(执行上下文),即一个 CPU 执行时所需要的一串指令。
进程
进程是指一个程序在给定数据集合上的一次执行过程,是系统进行资源分配和运行调用的独立单位。可以简单地理解为操作系统中正在执行的程序。也就说,每个应用程序都有一个自己的进程。
每一个进程启动时都会最先产生一个线程,即主线程。然后主线程会再创建其他的子线程。
进程vs程序
程序仅仅只是一堆代码而已,而进程指的是程序的运行过程。
需要强调的是:同一个程序执行两次,那也是两个进程。
进程vs线程
- 同一个进程中的线程共享同一内存空间,但是进程之间是独立的。
- 同一个进程中的所有线程的数据是共享的(进程通讯),进程之间的数据是独立的。
- 对主线程的修改可能会影响其他线程的行为,但是父进程的修改(除了删除以外)不会影响其他子进程。
- 线程是一个上下文的执行指令,而进程则是与运算相关的一簇资源。
- 同一个进程的线程之间可以直接通信,但是进程之间的交流需要借助中间代理来实现。
- 创建新的线程很容易,但是创建新的进程需要对父进程做一次复制。
- 一个线程可以操作同一进程的其他线程,但是进程只能操作其子进程。
- 线程启动速度快,进程启动速度慢(但是两者运行速度没有可比性)。
更形象化的解释进程和线程
- 计算机的核心是CPU,它承担了所有的计算任务。它就像一座工厂,时刻在运行。
- 假定工厂的电力有限,一次只能供给一个车间使用。也就是说,一个车间开工的时候,其他车间都必须停工。背后的含义就是,单个CPU一次只能运行一个任务。编者注:多核的CPU就像有了多个发电厂,使多工厂(多进程)实现可能。
- 进程就好比工厂的车间,它代表CPU所能处理的单个任务。任一时刻,CPU总是运行一个进程,其他进程处于非运行状态。
- 一个车间里,可以有很多工人。他们协同完成一个任务。 线程就好比车间里的工人。一个进程可以包括多个线程。
- 车间的空间是工人们共享的,比如许多房间是每个工人都可以进出的。这象征一个进程的内存空间是共享的,每个线程都可以使用这些共享内存。
- 可是,每间房间的大小不同,有些房间最多只能容纳一个人,比如厕所。里面有人的时候,其他人就不能进去了。这代表一个线程使用某些共享内存时,其他线程必须等它结束,才能使用这一块内存。
- 一个防止他人进入的简单方法,就是门口加一把锁。先到的人锁上门,后到的人看到上锁,就在门口排队,等锁打开再进去。这就叫"互斥锁"(Mutual exclusion,缩写 Mutex),防止多个线程同时读写某一块内存区域。
- 还有些房间,可以同时容纳n个人,比如厨房。也就是说,如果人数大于n,多出来的人只能在外面等着。这好比某些内存区域,只能供给固定数目的线程使用。
- 这时的解决方法,就是在门口挂n把钥匙。进去的人就取一把钥匙,出来时再把钥匙挂回原处。后到的人发现钥匙架空了,就知道必须在门口排队等着了。这种做法叫做"信号量"(Semaphore),用来保证多个线程不会互相冲突。
- 不难看出,mutex是semaphore的一种特殊情况(n=1时)。也就是说,完全可以用后者替代前者。但是,因为mutex较为简单,且效率高,所以在必须保证资源独占的情况下,还是采用这种设计。
操作系统的设计,因此可以归结为三点:
- 以多进程形式,允许多个任务同时运行;
- 以多线程形式,允许单个任务分成不同的部分运行;
- 提供协调机制,一方面防止进程之间和线程之间产生冲突,另一方面允许进程之间和线程之间共享资源。
操作系统的2个核心作用
- 为用户屏蔽了复杂繁琐的硬件接口,为应用程序提供了,清晰易用的系统接口
- 操作系统将应用程序对硬件资源的竞争变成有序的使用
多道技术
- 产生背景:针对单核,实现并发
多道技术中的多道指的是多个程序,多道技术的实现是为了解决多个程序竞争或者说共享同一个资源(比如cpu)的有序调度问题,解决方式即多路复用,多路复用分为时间上的复用和空间上的复用
ps:现在的主机一般是多核,那么每个核都会利用多道技术。有4个cpu,运行于cpu1的某个程序遇到io阻塞,会等到io结束再重新调度,会被调度到4个 cpu中的任意一个,具体由操作系统调度算法决定。
- 空间上的复用:
将内存分为几部分,每个部分放入一个程序,这样,同一时间内存中就有了多道程序。
空间上的复用最大的问题是:
程序之间的内存必须分割,这种分割需要在硬件层面实现,由操作系统控制。如果内存彼此不分割,则一个程序可以访问另外一个程序的内存,
首先丧失的是安全性,比如你的qq程序可以访问操作系统的内存,这意味着你的qq可以拿到操作系统的所有权限。
其次丧失的是稳定性,某个程序崩溃时有可能把别的程序的内存也给回收了,比方说把操作系统的内存给回收了,则操作系统崩溃。
- 时间上的复用:
当一个程序在等待I/O时,另一个程序可以使用cpu,如果内存中可以同时存放足够多的作业,则cpu的利用率可以接近100%,类似于我们小学数学所学的统筹方法。(操作系统采用了多道技术后,可以控制进程的切换,或者说进程之间去争抢cpu的执行权限。这种切换不仅会在一个进程遇到io时进行,一个进程占用cpu时间过长也会切换,或者说被操作系统夺走cpu的执行权限)
并发vs并行
无论是并行还是并发,在用户看来都是“同时”运行的,不管是进程还是线程,都只是一个任务而已,真是干活的是cpu,cpu来做这些任务,而一个cpu同一时刻只能执行一个任务。
并发:
是伪并行,即看起来是同时运行。单个cpu+多道技术就可以实现并发。
单核下,可以利用多道技术,多个核,每个核也都可以利用多道技术(多道技术是针对单核而言的)有四个核,六个任务,这样同一时间有四个任务被执行,假设分别被分配给了cpu1,cpu2,cpu3,cpu4。
一旦任务1遇到I/O就被迫中断执行,此时任务5就拿到cpu1的时间片去执行,这就是单核下的多道技术。而一旦任务1的I/O结束了,操作系统会重新调用它(需知进程的调度、分配给哪个cpu运行,由操作系统说了算)可能被分 配给四个cpu中的任意一个去执行。
并行:
同时运行,只有具备多个cpu才能实现并行。
多线程
线程和进程就是上下级的关系,相互依赖,许许多多的线程共同组成了进程,而一个进程至少包含一个线程,在这里将会谈到在节省开支的条件下,达到使用资源的最大化。进程只是将资源集合到一起,而线程才是CPU上的执行单位。
多线程,即多个控制线程,需要注意的是,多个线程是共享进程的地址空间的。
其他语言,CPU 是多核时是支持多个线程同时执行。但在 Python 中,无论是单核还是多核,同时只能由一个线程在执行。其根源是 GIL 的存在。
GIL 的全称是 Global Interpreter Lock(全局解释器锁),来源是 Python 设计之初的考虑,为了数据安全所做的决定。某个线程想要执行,必须先拿到 GIL,我们可以把 GIL 看作是“通行证”,并且在一个 Python 进程中,GIL 只有一个。拿不到通行证的线程,就不允许进入 CPU 执行。
目前python的解释器有多种:Cpython、PyPy、Jython、IronPython。GIL 只在 CPython 中才有,而在 PyPy 和 Jython 中是没有 GIL 的。
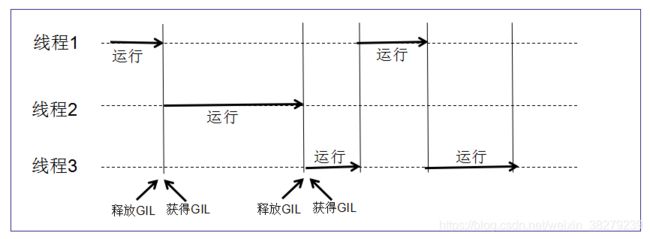
每次释放 GIL锁,线程进行锁竞争、切换线程,会消耗资源。这就导致打印线程执行时长,会发现耗时更长的原因。并且由于 GIL 锁存在,Python 里一个进程永远只能同时执行一个线程(拿到 GIL 的线程才能执行),这就是为什么在多核CPU上,Python 的多线程效率并不高的根本原因。
那么是不是意味着python中就只能使用多进程去提高效率,多线程就要被淘汰了呢?那也不是的。
这里分两种情况来讨论,CPU密集型操作和IO密集型操作。针对前者,大多数时间花在CPU运算上,所以希望CPU利用的越充分越好,这时候使用多进程是合适的,同时运行的进程数和CPU的核数相同;针对后者,大多数时间花在IO交互的等待上,此时一个CPU和多个CPU是没有太大差别的,反而是线程切换比进程切换要轻量得多,这时候使用多线程是合适的。
CPU密集型操作
程序比较偏重于计算,需要经常使用 CPU 来运算。
使用多进程比较合适,例如海量运算,机器学习等
IO密集型操作
程序需要频繁进行输入输出操作。
使用多线程比较合适,例如爬虫,文件处理,批量ssh操作服务器等
线程池
每一个线程的从生成到消亡也是需要时间和资源的,太多的线程会占用过多的系统资源(内存开销,cpu开销),而且生成太多的线程时间也是可观的,很可能会得不偿失。
最佳线程数的获取
- 通过用户慢慢递增来进行性能压测,观察QPS(即每秒的响应请求数,也即是最大吞吐能力。),响应时间
- 根据公式计算:服务器端最佳线程数量=((线程等待时间+线程cpu时间)/线程cpu时间) * cpu数量
- 单用户压测,查看CPU的消耗,然后直接乘以百分比,再进行压测,一般这个值的附近应该就是最佳线程数量。
使用线程池的意义
对于任务数量不断增加的程序,每有一个任务就生成一个线程,最终会导致线程数量的失控,例如,整站爬虫,假设初始只有一个链接a,那么,这个时候只启动一个线程,运行之后,得到这个链接对应页面上的b,c,d,,,等等新的链接,作为新任务,这个时候,就要为这些新的链接生成新的线程,线程数量暴涨。在之后的运行中,线程数量还会不停的增加,完全无法控制。所以,对于任务数量不端增加的程序,固定线程数量的线程池是必要的。
ref
- 转录组分析进阶
- [python线程池的多种实现方法]