今天跟大家分享的是八月份发表在frontiers in Oncology杂志(IF:4.848)上的一篇文章,文章主要讲的是利用CGGA的RNA: m5C甲基转移酶的RNA-sequence和样本的临床病理学数据,分析了RNA:m5C甲基转移酶基因在不同临床病理特征胶质瘤中的表达,并通过一致性聚类分析确定了不同亚型。利用GO和GSEA对这些基因的功能进行注释。使用单变量Cox回归和LASSO Cox回归算法确定了风险特征。此外,还开发了Cox比例回归模型分析来评估风险评分和临床因素的联系。
在神经胶质瘤中识别RNA:5-甲基胞嘧啶甲基转移酶相关的预后特征
一、摘要
胶质瘤是颅内最常见的原发性肿瘤,占颅内恶性肿瘤的绝大多数。RNA:5-甲基胞嘧啶(m5C)甲基转移酶的表达异常是近年来肿瘤发生发展研究的热点,但在胶质瘤中RNA:m5C甲基转移酶的预后价值尚不清楚。本工作研究了RNA: m5C甲基转移酶的表达,并确定了其在胶质瘤中的临床病理特征和预后价值。本工作利用Chinese Glioma Genome Atlas (CGGA)的RNA: m5C甲基转移酶的RNA-sequence和样本的临床病理学数据,分析了RNA:m5C甲基转移酶基因在不同临床病理特征胶质瘤中的表达,并通过一致性聚类分析确定了不同亚型。利用Gene Ontology (GO)和基因集富集分析(GSEA)对这些基因的功能进行注释。使用单变量Cox回归和LASSO Cox回归算法确定了风险特征。此外,还开发了Cox比例回归模型分析来评估风险评分和临床因素的联系。本工作揭示了RNA:m5C甲基转移酶基因在具有不同临床病理特征的胶质瘤中的差异表达。对RNA:m5C甲基转移酶的一致性聚类分析识别到三组具有不同预后和临床病理特征的胶质瘤。同时,功能注释显示,RNA:m5C甲基转移酶与胶质瘤恶性进展显著相关。对5个RNA:m5C甲基转移酶基因进行了筛选,构建了一个风险特征,不仅可以用于预测胶质瘤的总体生存,还可以用于预测临床病理特征。多因素Cox回归分析显示风险评分是影响胶质瘤预后的独立因素。本工作证明了RNA:m5C甲基转移酶在胶质瘤的起始和进展中的预后作用。并且扩展了对相关分子机制的理解,并提供了一种独特的方法来预测生物标志物和神经胶质瘤的靶向治疗。
二、数据及方法
1. 样本及数据
(1)从CGGA data portal(http://www.cgga.org.cn/)下载CGGA RNA测序数据和样本的临床信息作为训练集。从TCGA data portal(https://www.cancergenome.nih.gov/)下载TCGA RNA测序数据和样本的临床信息作为验证集。此外还下载TCGA 的体细胞突变数据。
去除缺少生存期和WHO分级数据的样本后,获得了306例(CGGA数据集)和616例(TCGA数据集)胶质瘤患者。
(2)根据已发表的文献筛选了7个RNA:m5C甲基转移酶。
(3)研究中使用的所有胶质瘤组织样本都来自于2015年至2019间南昌大学第二附属医院的手术患者。将胶质瘤标本分为4组:Grade I、Grade、II、Grade III和Grade IV。
(4)采用来自顽固性癫痫患者手术的25例脑样本作为对照,包括9例高级别胶质瘤(WHO III、IV级)、10例低级别胶质瘤(WHO II级)和6例非肿瘤性脑组织(NBTs)。
2.分析方法
(1)聚类分析和主成分分析
根据CGGA数据集的306个样本的RNA:m5C甲基转移酶表达,使用R包ConsensusClusterPlus进行一致性聚类分析,将样本分成三类。然后,通过R包pca3d和rgl使用主成分分析(PCA)来检测三种亚型之间的差异基因表达。
(2)生物学功能分析
利用String网站(https://string-db.org/)和R包corrplot预测了7个RNA:m5C甲基转移酶的功能和相互作用。使用Metascape (http:// metascape.org/)进行GO分析,以注释不同亚型差异表达基因的功能。然后使用DAVID进行KEGG通路富集,用imageGP website进行可视化。使用JAVA 进行GSEA。
(3)构建和评估风险评分
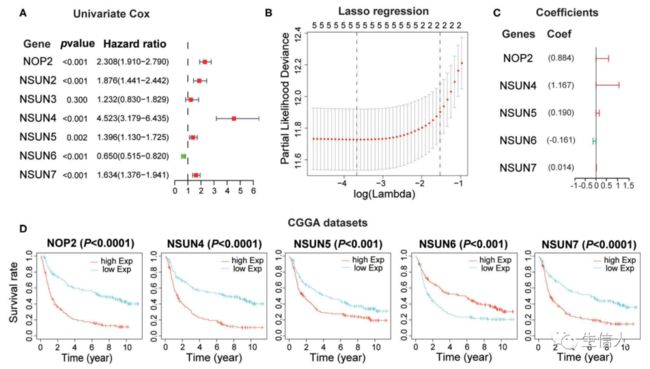
使用单因素Cox回归分析确定与生存显著相关的基因(P < 0.05)。使用R包glmnet进行LASSO多元Cox回归分析筛选出6个RNA:m5C甲基转移酶(NOP2、NSUN2、NSUN4、NSUN5、NSUN6和NUSN7)。5个RNA:m5C甲基转移酶 (NOP2、NSUN4、NSUN5、NSUN6和NUSN7)的风险评分系统是在CGGA数据集中构建,用TCGA的数据集进行评估。使用中值风险评分将患者分为高危组和低危组。此外,使用R包Maftools分析这些胶质瘤患者的基因组改变。
(4)人类蛋白质图谱
从The Human Protein Atlas (https://www.proteinatlas.org/)下载RNA:m5C甲基转移酶的免疫组化病理标本。可在线查询染色强度、染色数量及患者信息。
(5)统计分析
采用单因素方差分析和R包limma来比较不同WHO分级和IDH状态胶质瘤中RNA:m5C甲基转移酶的表达水平。通过t-tests对比神经胶质瘤的临床病理特征。采用卡方检验比较按风险分值分层的不同组间临床和分子病理特征的分布。使用R软件包glmnet进行单变量、多变量和LASSO Cox回归分析,R包survival进行生存分析。R包survivalROC进行ROC曲线分析来预测胶质瘤患者的OS。P<0.05认为具有统计学显著性。
三、结果解析
1. RNA:m5C甲基转移酶的异常表达与胶质瘤临床病理特征的关联
热图展示RNA:m5C甲基转移酶的异常表达与世卫组织(WHO)分级之间的关系(图1A,B)。几乎所有RNA:m5C甲基转移酶的表达与WHO分级显著相关,显著差异表达的RNA:m5C甲基转移酶包括NOP2、NSUN2、NSUN4、NSUN5、NSUN6和NSUN7。然后,定量分析了这些差异表达基因在CGGA数据集中的表达情况(图1C),并且在TCGA数据集中进行验证(图1D)。
IDH基因的突变已经在胶质瘤中有报道,特别是在低级别胶质瘤(LGGs)中,该突变的预后价值已经被许多作者在文献中证实。接下来研究了RNA:m5C甲基转移酶的异常表达与LGGs中IDH突变状态的关系。结果显示在IDH突变和野生型中,NSUN3、NSUN4、NSUN5、NSUN6和NSUN7差异表达,在CGGA (图1E)和TCGA(图1F)数据集中都是如此。还研究了RNA:m5C甲基转移酶在胶质母细胞瘤(GBM)中的表达,按IDH突变状态分层,结果显示NSUN5、NSUN6、NSUN7仍存在差异表达。
此外,还预测了7个RNA:m5C甲基转移酶的基因在cBioPortal中突变频率,并使用TCGA数据集验证,这些RNA:m5C甲基转移酶的突变是罕见的。即使是排名最高的RNA:m5C甲基转移酶如NOP2,其突变频率也只有3%。说明的异常表达可能不是由基因突变产生的。
图1.CGGA和TCGA数据集胶质瘤中RNA:m5C甲基转移酶的异常表达与临床病理特征的关系
二、RNA:m5C甲基转移酶的相互作用和无监督的一致性分析
为了研究RNA:m5C甲基转移酶与胶质瘤临床病理特征的密切联系,系统的研究了RNA:m5C甲基转移酶的功能、相互作用和相关性。发现所有的RNA:m5C甲基转移酶基因都参与了各种类型的甲基化,其中NOP2、NSUN3、NSUN4和NSUN5主要参与rRNA甲基化,NSUN2、NSUN6和NSUN3参与tRNA甲基化。文本挖掘和共表达分析的结果显示它们的功能和互作(图2A)。通过Pearson相关分析研究了7个RNA:m5C甲基转移酶在CGGA(图2 B)和TCGA中的表达谱。NOP2、NSUN2、NSUN4、NSUN5、NSUN7在胶质瘤中的表达呈正相关,而NSUN6的表达与NSUN4, NSUN5和NSUN7显著负相关。这些结果符合对胶质瘤RNA:m5C甲基转移酶表达定量分析的结果,表明NOP2、NSUN2、NSUN4、NSUN5、NSUN7的表达水平与胶质瘤的恶性进展呈正相关,而NSUN6的表达水平与胶质瘤呈负相关。
基于CGGA数据集中306例胶质瘤患者的RNA:m5C甲基转移酶表达谱,使用无监督一致性聚类分析确定了3个亚型,即MC1、MC2和MC3(图2C)。使用PCA分析来比较这三个亚组的转录谱。结果表明,它们可以被充分地划分为三个不同的簇(图2E)。接下来研究了这三种亚型的RNA:m5C甲基转移酶表达谱与胶质瘤临床病理特征之间的关系(图2D)。在这三个亚型中,MC2亚型相比于MC3亚型,MC3亚型相比于MC1亚型与胶质瘤高级别、IDH-wildtype状态、1p/19q-noncodel 状态、更高的平均诊断年龄和接受额外化疗具有更显著的相关性。此外,发现三组患者的总生存率存在显著差异。MC2亚型患者的生存期明显短于其他两种亚型(图2F)。对TCGA数据集中这些RNA:m5C甲基转移酶的无监督一致性分析与CGGA数据集的结果一致。以上结果表明,RNA:m5C甲基转移酶的一致性聚类可以识别胶质瘤中具有不同临床病理特征和预后的亚型。
图2.RNA:m5C甲基转移酶的相互作用和无监督一致性聚类分析
三、亚型的功能注释
为了调查不同的临床病理特征和三组胶质瘤的总生存率,对与MC2型胶质瘤相关的特定基因进行生物学过程注释。相对于MC1和MC3亚型,MC2中664个基因表达上调,645个基因表达下调。对上调基因的GO分析显示,“细胞外基质组织”、“脉管系统发育”、“上皮细胞增殖”、“细胞底物粘附”和“细胞对肿瘤坏死因子的反应”的生物过程和通路富集,可能与胶质瘤的恶性进展高度相关。显著富集前20个生物过程如图所示(图3A)。KEGG通路分析进一步揭示这些基因也与肿瘤相关信号通路显著相关,例如:ECM-受体相互作用、Jak-STAT信号通路和P53信号通路等(图3B)。
GSEA结果显示,P53通路、P13K/AKT/mTOR信号通路、DNA修复和MTORC1信号(图3C,D)在MC2亚型中富集。结合以上分析,通过RNA:m5C甲基转移酶识别的亚型与胶质瘤的恶性进展显著相关。
图3.MC2亚型特定基因的功能注释
四、RNA:m5C甲基转移酶的预后价值和5个RNA:m5C甲基转移酶基因的风险评分特征的构建
基于RNA:m5C甲基转移酶和神经胶质瘤的恶性进展的关系,接下来进一步探索RNA:m5C甲基转移酶在神经胶质瘤的预后作用,通过对作为训练数据集的CGGA数据集使用Cox比例风险模型来进行单因素生存分析。获得了6个与预后相关的基因,其中NOP2、NSUN2、NSUN4、NSUN5、NSUN7为风险因素(HR> 1),NSUN6在胶质瘤中起保护作用(HR < 1)(图4A)。为了提高6个RNA:m5C甲基转移酶的鲁棒性,使用这些基因在CGGA数据集中通过LASSO Cox回归算法进行额外分析(图4 B)。筛选出5个RNA:m5C甲基转移酶基因和系数(图4C)构建风险评分特征。在风险评分特征中,四个基因(NOP2、NSUN4、NSUN5和NSUN7)具有促癌作用,NSUN6是一个抑癌基因。为了更好地了解这5个预后基因在胶质瘤中的作用,在CGGA数据集和TCGA数据集中都进行了生存分析,在这些数据集中,根据中位基因表达水平将样本分为高表达或低表达。所有5个RNA:m5C甲基转移酶基因均与OS显著相关(图4D)。
图4.使用5个RNA:m5C甲基转移酶基因构建风险评分特征
五、胶质瘤风险评分标记的预后价值
为了获得风险评分特征对胶质瘤患者临床结果的预测效果,以所有患者评分的中位数作为标准,在CGGA和TCGA数据集中将样本分为高风险组和低风险组。分析表明,随着风险评分的增加,死亡患者的数量显著增加(图5A,D)。此外,高风险组和低风险组在总生存率上有显著差异(图5B,E)。然后,对1、3和5年的预后风险评分进行ROC曲线分析,以检验风险特征的预测效率。结果表明,风险评分在识别胶质瘤OS上具有较高的准确性(图5C,F)。此外,根据WHO分级和IDH突变状态对这一特征在胶质瘤患者中的预后价值进行了进一步研究。结果显示,在CGGA数据集中,在具有不同的WHO分级亚型(LGG和GBM)和IDH突变状态亚型情况下,风险评分特征可用于将胶质瘤患者分为两种不同的预后组(图5G–J)。使用TCGA数据集也得到了一致结果。综合以上分析,认为风险评分的预后效果准确、稳定。
图5.CGGA和TCGA数据集中的风险评分特征的预后价值
六、胶质瘤患者风险评分与临床病理特征的关系
CGGA数据集中筛选的5个RNA:m5C甲基转移酶在高、低风险患者中的表达由热图表示(图6A)。在CGGA和TCGA数据集中,基于WHO分级、组织学、IDH状态、1p/19q状态、年龄和接受额外化疗上,发现低风险组和高风险组之间存在显著的统计学差异。然后,对胶质瘤的风险评分进行定量分析,来分析风险评分与临床病理特征之间的关系。根据以上WHO分级、组织学等对CGGA数据集进行划分,这些组的风险评分显著不同(图6B),TCGA数据集也得到验证。结果表明,5种RNA:m5C甲基转移酶识别的风险评分与胶质瘤恶性程度显著相关。此外,考虑到已报道的胶质瘤相关驱动基因改变在胶质瘤启动和进展中的重要性,包括ATRX、P53通路(TP53、MDM2和MDM4)、RB通路(CDK4、CDK6、CCND2、CDKNA/B和RB1)和P13K/RTK通路(PIK3CA、PIK3R1、PTEN、EGFR、PDGFRA和NF1),从TCGA数据库中获取体细胞突变数据。高、低风险胶质瘤患者之间的肿瘤驱动基因改变的突变景观呈现为瀑布图(图6C)。高风险患者肿瘤驱动基因的改变较低风险患者更为频繁。这意味着高风险患者的癌症进展更为严重。综合以上结果,风险评分不仅可以预测总生存率,还可以预测临床病理特征。
接下来,根据8个临床病理特征来研究这个风险评分是否是一个独立的预后因素。使用CGGA数据集进行单变量和多变量Cox回归分析。通过单因素分析,观察到风险评分、年龄、WHO级别、IDH状态、1p/19q codel状态、化疗状态、放疗状态与预后显著相关(图7A)。基于上述因素进行多因素分析,风险评分仍然与OS密切相关(图7B)。在验证数据集(TCGA)中也得到了相似的结果,在多因素分析中包含了相同的因素;风险评分也与OS密切相关(图7C,D)。一致的结果表明,通过RNA:m5C甲基转移酶构建的风险评分是胶质瘤的一个强有力的独立的预后因素。
图6.胶质瘤的风险评分和临床病理特征的相互关系,以及肿瘤驱动基因改变的突变景观
图7.八种临床病理特征及生物学功能的单因素和多因素Cox回归分析
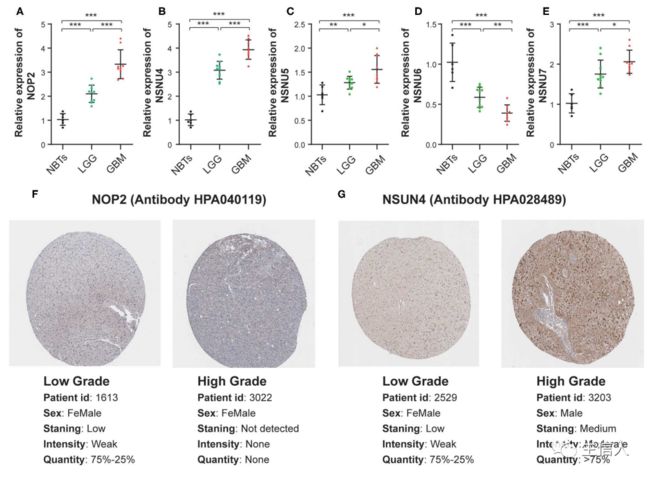
七、胶质瘤中5种预后RNA:m5C甲基转移酶的mRNA和蛋白表达模式
为了评估对构建风险评分特征有重要意义的基因,进行了qRT-PCR检测,并从Human Protein Atlas网站获得免疫组化病理标本数据。5个RNA:m5C甲基转移酶基因NOP2、NSUN4、NSUN5、NSUN6和NSUN7在正常脑组织、低级别和高级别胶质瘤组织中表达差异(图8A–E)。此外,发现高级别胶质瘤组织中NOP2和NSUN4的蛋白表达远高于低级别胶质瘤组织(图8F,G)。然而,NSUN5、NSUN6、NSUN7的蛋白表达模式由于缺乏免疫组化图像,本文未见报道。
图8.胶质瘤中5种预后RNA:m5C甲基转移酶的mRNA和蛋白表达模式
总结:有分析意向(http://gaptechsxr.mikecrm.com/1vdMmqy)生信人WX公众号
本工作利用CGGA的RNA: m5C甲基转移酶的RNA-sequence和样本的临床病理学数据,分析了RNA:m5C甲基转移酶基因在不同临床病理特征胶质瘤中的表达,发现几乎所有RNA:m5C甲基转移酶基因的表达与胶质瘤分级显著相关,几个与IDH突变状态相关。NOP2、NSUN2、NSUN4、NSUN5、NSUN7的表达水平与胶质瘤的恶性进展呈正相关,而NSUN6的表达水平与胶质瘤呈负相关。通过一致性聚类分析确定了3种不同亚型,并三组患者的总生存率存在显著差异,且MC2亚型患者的生存期明显短于其他两种亚型。然后利用Gene Ontology (GO)和基因集富集分析(GSEA)对这些基因的功能进行注释。使用单变量Cox回归和LASSO Cox回归算法确定了风险特征。此外,还开发了Cox比例回归模型分析来评估风险评分和临床因素的联系,证明风险评分是影响胶质瘤预后的独立因素。