Pre-processing
本文将要介绍的是在R中进行RNA-seq 数据预处理的实战代码
原文地址:https://bioinformatics-core-shared-training.github.io/RNAseq-R/rna-seq-preprocessing.nb.html
主要包括以下方面:
载入mapping, counting后的数据
过滤低表达基因
对表达数据进行质量控制
标准化处理
本次流程需要的包
library(edgeR)
library(limma)
library(Glimma)
library(gplots)
library(org.Mm.eg.db)
library(RColorBrewer)
- 温馨提示:
org.Mm.eg.db有60多M,个人推荐没有安装过的朋友先切换到国内的镜像,再下载这个包。
options(repos = 'https://mirrors.tuna.tsinghua.edu.cn/CRAN/')
getOption('repos')
BiocManager::install('org.Mm.eg.db')
另外,本次所需要的数据均可以在该网站下载:https://figshare.com/s/1d788fd384d33e913a2a
下载的数据有:
- Sampleinfo.txt
- GSE60450_Lactation-GenewiseCounts.txt
- mouse_c2_v5.rdata
- mouse_H_v5.rdata
载入数据
首先,我们读入样本信息"SampleInfo.txt"
> sampleinfo <- read.delim("data/SampleInfo.txt")
> sampleinfo
FileName SampleName CellType Status
1 MCL1.DG_BC2CTUACXX_ACTTGA_L002_R1 MCL1.DG luminal virgin
2 MCL1.DH_BC2CTUACXX_CAGATC_L002_R1 MCL1.DH basal virgin
3 MCL1.DI_BC2CTUACXX_ACAGTG_L002_R1 MCL1.DI basal pregnant
4 MCL1.DJ_BC2CTUACXX_CGATGT_L002_R1 MCL1.DJ basal pregnant
5 MCL1.DK_BC2CTUACXX_TTAGGC_L002_R1 MCL1.DK basal lactate
6 MCL1.DL_BC2CTUACXX_ATCACG_L002_R1 MCL1.DL basal lactate
7 MCL1.LA_BC2CTUACXX_GATCAG_L001_R1 MCL1.LA basal virgin
8 MCL1.LB_BC2CTUACXX_TGACCA_L001_R1 MCL1.LB luminal virgin
9 MCL1.LC_BC2CTUACXX_GCCAAT_L001_R1 MCL1.LC luminal pregnant
10 MCL1.LD_BC2CTUACXX_GGCTAC_L001_R1 MCL1.LD luminal pregnant
11 MCL1.LE_BC2CTUACXX_TAGCTT_L001_R1 MCL1.LE luminal lactate
12 MCL1.LF_BC2CTUACXX_CTTGTA_L001_R1 MCL1.LF luminal lactate
再读入基因计数后的数据"GSE60450_Lactation-GenewiseCounts.txt"
> seqdata <- read.delim("data/GSE60450_Lactation-GenewiseCounts.txt",
+ stringsAsFactors = FALSE)
> head(seqdata)
EntrezGeneID Length MCL1.DG_BC2CTUACXX_ACTTGA_L002_R1 MCL1.DH_BC2CTUACXX_CAGATC_L002_R1
1 497097 3634 438 300
2 100503874 3259 1 0
3 100038431 1634 0 0
4 19888 9747 1 1
5 20671 3130 106 182
6 27395 4203 309 234
MCL1.DI_BC2CTUACXX_ACAGTG_L002_R1 MCL1.DJ_BC2CTUACXX_CGATGT_L002_R1
1 65 237
2 1 1
3 0 0
4 0 0
5 82 105
6 337 300
MCL1.DK_BC2CTUACXX_TTAGGC_L002_R1 MCL1.DL_BC2CTUACXX_ATCACG_L002_R1
1 354 287
2 0 4
3 0 0
4 0 0
5 43 82
6 290 270
MCL1.LA_BC2CTUACXX_GATCAG_L001_R1 MCL1.LB_BC2CTUACXX_TGACCA_L001_R1
1 0 0
2 0 0
3 0 0
4 10 3
5 16 25
6 560 464
MCL1.LC_BC2CTUACXX_GCCAAT_L001_R1 MCL1.LD_BC2CTUACXX_GGCTAC_L001_R1
1 0 0
2 0 0
3 0 0
4 10 2
5 18 8
6 489 328
MCL1.LE_BC2CTUACXX_TAGCTT_L001_R1 MCL1.LF_BC2CTUACXX_CTTGTA_L001_R1
1 0 0
2 0 0
3 0 0
4 0 0
5 3 10
6 307 342
> dim(seqdata) # 数据包含了27179行的基因信息,1列ID、1列基因长度和12列的样本数据
[1] 27179 14
由于我们关注的是基因计数的信息,因此为了方便下游分析处理,我们移除seqdata中前两列,并将第一列的ID命名为行名
> countdata <- seqdata[,-(1:2)]
> rownames(countdata) <- seqdata[,1]
> head(countdata)
MCL1.DG_BC2CTUACXX_ACTTGA_L002_R1 MCL1.DH_BC2CTUACXX_CAGATC_L002_R1
497097 438 300
100503874 1 0
100038431 0 0
19888 1 1
20671 106 182
27395 309 234
MCL1.DI_BC2CTUACXX_ACAGTG_L002_R1 MCL1.DJ_BC2CTUACXX_CGATGT_L002_R1
497097 65 237
100503874 1 1
100038431 0 0
19888 0 0
20671 82 105
27395 337 300
MCL1.DK_BC2CTUACXX_TTAGGC_L002_R1 MCL1.DL_BC2CTUACXX_ATCACG_L002_R1
497097 354 287
100503874 0 4
100038431 0 0
19888 0 0
20671 43 82
27395 290 270
MCL1.LA_BC2CTUACXX_GATCAG_L001_R1 MCL1.LB_BC2CTUACXX_TGACCA_L001_R1
497097 0 0
100503874 0 0
100038431 0 0
19888 10 3
20671 16 25
27395 560 464
MCL1.LC_BC2CTUACXX_GCCAAT_L001_R1 MCL1.LD_BC2CTUACXX_GGCTAC_L001_R1
497097 0 0
100503874 0 0
100038431 0 0
19888 10 2
20671 18 8
27395 489 328
MCL1.LE_BC2CTUACXX_TAGCTT_L001_R1 MCL1.LF_BC2CTUACXX_CTTGTA_L001_R1
497097 0 0
100503874 0 0
100038431 0 0
19888 0 0
20671 3 10
27395 307 342
另外,我们注意到countdata中的列名过于冗长,而样本信息中的名字也只是前7位字母而已,因此我们使用substr()函数截取列名
> sampleinfo$SampleName
[1] MCL1.DG MCL1.DH MCL1.DI MCL1.DJ MCL1.DK MCL1.DL MCL1.LA MCL1.LB MCL1.LC MCL1.LD MCL1.LE MCL1.LF
12 Levels: MCL1.DG MCL1.DH MCL1.DI MCL1.DJ MCL1.DK MCL1.DL MCL1.LA MCL1.LB MCL1.LC ... MCL1.LF
> colnames(countdata)
[1] "MCL1.DG_BC2CTUACXX_ACTTGA_L002_R1" "MCL1.DH_BC2CTUACXX_CAGATC_L002_R1"
[3] "MCL1.DI_BC2CTUACXX_ACAGTG_L002_R1" "MCL1.DJ_BC2CTUACXX_CGATGT_L002_R1"
[5] "MCL1.DK_BC2CTUACXX_TTAGGC_L002_R1" "MCL1.DL_BC2CTUACXX_ATCACG_L002_R1"
[7] "MCL1.LA_BC2CTUACXX_GATCAG_L001_R1" "MCL1.LB_BC2CTUACXX_TGACCA_L001_R1"
[9] "MCL1.LC_BC2CTUACXX_GCCAAT_L001_R1" "MCL1.LD_BC2CTUACXX_GGCTAC_L001_R1"
[11] "MCL1.LE_BC2CTUACXX_TAGCTT_L001_R1" "MCL1.LF_BC2CTUACXX_CTTGTA_L001_R1"
> colnames(countdata) <- substr(colnames(countdata), 1, 7)
> colnames(countdata)
[1] "MCL1.DG" "MCL1.DH" "MCL1.DI" "MCL1.DJ" "MCL1.DK" "MCL1.DL" "MCL1.LA" "MCL1.LB" "MCL1.LC"
[10] "MCL1.LD" "MCL1.LE" "MCL1.LF"
> table(colnames(countdata)==sampleinfo$SampleName) #使用table函数检验修改后的名字与原名字是否相同
TRUE
12
数据过滤
对于表达量过低的基因而言,其数据在差异分析中的可信度是较差的。因此,在本流程中我们先使用edgeR的cpm()函数,将counting后的数据转换为CPM(counts-per-million),并取CPM在两个重复样本中均大于0.5的基因进入后续分析。
> myCPM <- cpm(countdata)
> head(myCPM)
MCL1.DG MCL1.DH MCL1.DI MCL1.DJ MCL1.DK MCL1.DL MCL1.LA
497097 18.85684388 13.77543859 2.69700983 10.45648006 16.442685 14.3389690 0.0000000
100503874 0.04305215 0.00000000 0.04149246 0.04412017 0.000000 0.1998463 0.0000000
100038431 0.00000000 0.00000000 0.00000000 0.00000000 0.000000 0.0000000 0.0000000
19888 0.04305215 0.04591813 0.00000000 0.00000000 0.000000 0.0000000 0.4903857
20671 4.56352843 8.35709941 3.40238163 4.63261775 1.997275 4.0968483 0.7846171
27395 13.30311589 10.74484210 13.98295863 13.23605071 13.469996 13.4896224 27.4615975
MCL1.LB MCL1.LC MCL1.LD MCL1.LE MCL1.LF
497097 0.0000000 0.0000000 0.00000000 0.0000000 0.0000000
100503874 0.0000000 0.0000000 0.00000000 0.0000000 0.0000000
100038431 0.0000000 0.0000000 0.00000000 0.0000000 0.0000000
19888 0.1381969 0.4496078 0.09095771 0.0000000 0.0000000
20671 1.1516411 0.8092940 0.36383085 0.1213404 0.4055595
27395 21.3744588 21.9858214 14.91706476 12.4171715 13.8701357
> thresh <- myCPM > 0.5 # This produces a logical matrix with TRUEs and FALSEs
> head(thresh)
MCL1.DG MCL1.DH MCL1.DI MCL1.DJ MCL1.DK MCL1.DL MCL1.LA MCL1.LB MCL1.LC MCL1.LD MCL1.LE
497097 TRUE TRUE TRUE TRUE TRUE TRUE FALSE FALSE FALSE FALSE FALSE
100503874 FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
100038431 FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
19888 FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE
20671 TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE FALSE FALSE
27395 TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
MCL1.LF
497097 FALSE
100503874 FALSE
100038431 FALSE
19888 FALSE
20671 FALSE
27395 TRUE
用table()函数每个基因(每行)在几个样本中是符合条件的
> table(rowSums(thresh))
0 1 2 3 4 5 6 7 8 9 10 11 12
10857 518 544 307 346 307 652 323 547 343 579 423 11433
#可以发现有11433个基因在12个样本中CPM均大于0.5
随后,保留至少在两个样本符合条件的基因。
> keep <- rowSums(thresh) >= 2
# Subset the rows of countdata to keep the more highly expressed genes
> counts.keep <- countdata[keep,]
> summary(keep)
Mode FALSE TRUE
logical 11375 15804
> dim(counts.keep)
[1] 15804 12
我们保留了15804个相对表达可信的基因。至于,counts threshold的选择可以参考原文:
As a general rule, a good threshold can be chosen by identifying the CPM that corresponds to a count of 10, which in this case is about 0.5. You should filter with CPMs rather than filtering on the counts directly, as the latter does not account for differences in library sizes between samples.
创建表达矩阵
接着我们使用edgeR的DGEList函数来创建表达矩阵
# 将counts 转换为 DGEList 对象
> dgeObj <- DGEList(counts.keep)
> dgeObj
An object of class "DGEList"
$counts
MCL1.DG MCL1.DH MCL1.DI MCL1.DJ MCL1.DK MCL1.DL MCL1.LA MCL1.LB MCL1.LC MCL1.LD MCL1.LE
497097 438 300 65 237 354 287 0 0 0 0 0
20671 106 182 82 105 43 82 16 25 18 8 3
27395 309 234 337 300 290 270 560 464 489 328 307
18777 652 515 948 935 928 791 826 862 668 646 544
21399 1604 1495 1721 1317 1159 1066 1334 1258 1068 926 508
MCL1.LF
497097 0
20671 10
27395 342
18777 581
21399 500
15799 more rows ...
$samples
group lib.size norm.factors
MCL1.DG 1 23218026 1
MCL1.DH 1 21768136 1
MCL1.DI 1 24091588 1
MCL1.DJ 1 22656713 1
MCL1.DK 1 21522033 1
7 more rows ...
# 看看 dgeObj 中保存了什么信息
> names(dgeObj)
[1] "counts" "samples"
根据CellType和小鼠的Status,我们可以创建分组信息。
group <- paste(sampleinfo$CellType,sampleinfo$Status,sep=".")
group <- factor(group)
质量控制
由于我们的数据并不是正态分布的,为了下游分析的进行,使用cpm函数对我们的数据取log2处理
> logcounts <- cpm(dgeObj,log=TRUE)
> # 使用箱线图检查数据的分布
> boxplot(logcounts, xlab="", ylab="Log2 counts per million",las=2)
> # 使用蓝色的横线标出logCPM的中值
> abline(h=median(logcounts),col="blue")
> title("Boxplots of logCPMs (unnormalised)")
从箱线图中可以看出样本数据分布虽然存在差异,但这种差异程度是我们还能接受的程度。
主成分分析
主成分分析在RNA-seq分析中是一个较为重要的步骤,其指出了样本数据中造成主要差异的因子是什么。一般而言,我们当然希望数据中最大的差异是由于处理与否引起的
> plotMDS(dgeObj)
可以看到两个生物学重复样本都能较好的聚在一起
另外,我们也可以运用各种参数使主成分分析的可视化更加符合我们的要求,例如根据细胞类型或小鼠状态标注颜色:
总而言之,利用主成分分析,可以观察出引起样本变化的主要因子。
标准化处理
此处我们采用了The trimmed mean of M-values normalization method (TMM) 去校正文库之间的组成偏好。
> dgeObj <- calcNormFactors(dgeObj)
> dgeObj$samples
group lib.size norm.factors
MCL1.DG 1 23218026 1.2368993
MCL1.DH 1 21768136 1.2139485
MCL1.DI 1 24091588 1.1255640
MCL1.DJ 1 22656713 1.0698261
MCL1.DK 1 21522033 1.0359212
MCL1.DL 1 20008326 1.0872153
MCL1.LA 1 20384562 1.3684449
MCL1.LB 1 21698793 1.3653200
MCL1.LC 1 22235847 1.0047431
MCL1.LD 1 21982745 0.9232822
MCL1.LE 1 24719697 0.5291015
MCL1.LF 1 24652963 0.5354877
# 此步在‘samples’信息中直接添加了校正因子。
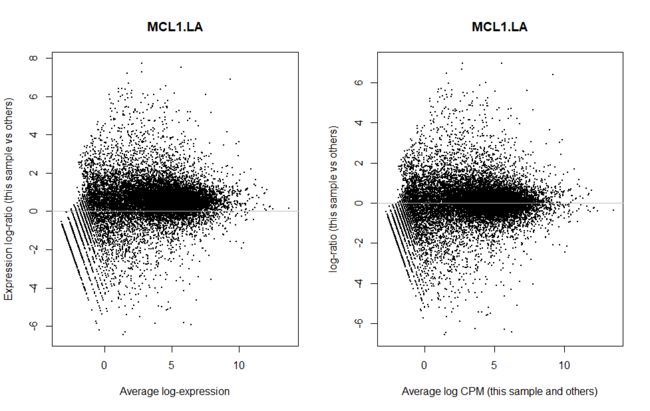
我们可以单独抽一个样本出来看看校正前后的数据分布情况
可以看出校正后,样本表达更加集中于0。
至此,数据的预处理也已经完结,文中的可视化部分仅是为了辅助我们查看数据与方便我们的学习,在实战中,部分数据预处理时的可视化也可省略。
#保存预处理分析结果
save(group,dgeObj,sampleinfo,file="~preprocessing.Rdata")
暂完。