张伟伟-层次1 Mean_shift聚类算法和其他的聚类算法
pycharm使用的简单教程
层次一 Mean_shift聚类算法和其他的聚类算法
- 作者介绍
- 1. 安装包的准备:
- 2. Mean-shift原理及实现
-
- 2.1 Mean-shift原理
- 2.2 主要参数
- 2.3 实现
- 2.4 结果
- 2.5 实例扩展:利用Meanshift进行目标追踪
- 3. Spectral Clustering原理及实现
-
- 3.1 Spectral Clustering原理
- 3.2 主要参数
- 3.3 实现
- 3.4 结果
- 4.Hierarchical Clustering原理及实现
-
- 4.1 Hierarchical Clustering原理
- 4.2 主要参数
- 4.3 实现
- 4.3 结果
- 5.DBSCAN原理及实现
-
- 5.1 DBSCAN原理
- 5.2 主要参数
- 5.3 实现
- 5.4 结果
- 6.Birch原理及实现
-
- 6.1 Birch原理
- 6.2 主要参数
- 6.3 实现
- 6.4 结果
- 7 GaussianMixtureModel原理及实现
-
- 7.1 GaussianMixtureModel原理
- 7.2 主要参数
- 7.3 实现
- 7.4 结果
作者介绍
张伟伟,男,西安工程大学电子信息学院,2019级硕士研究生,张宏伟人工智能课题组。
研究方向:机器视觉与人工智能。
电子邮件:[email protected]
1. 安装包的准备:
pip install sklearn
pip install matplotlib
pip install opencv-python
代码的百度网盘链接:https://pan.baidu.com/s/1neOlGt-3240B9tjhJVOy0A
链接提取码:2021
2. Mean-shift原理及实现
2.1 Mean-shift原理
Mean-shift(均值迁移)的基本思想:在数据集中选定一个点,然后以这个点为圆心,r为半径,画一个圆(二维下是圆)先算出当前点的偏移均值,移动该点到其偏移均值,然后以此为新的起始点,继续移动,然后迭代此过程,直到满足一点的条件结束。Mean-shift 算法的核心思想就是不断的寻找新的圆心坐标,直到密度最大的区域。

2.2 主要参数
- bandwidth :半径(或带宽),float型。如果没有给出,则使用- - sklearn.cluster.estimate_bandwidth计算出半径(带宽).(可选)
- seeds :圆心(或种子),数组类型,即初始化的圆心。(可选)
- bin_seeding :布尔值。如果为真,初始内核位置不是所有点的位置,而是点的离散版本的位置,其中点被分类到其粗糙度对应于带宽的网格上。将此选项设置为True将加速算法,因为较少的种子将被初始化。默认值:False.如果种子参数(seeds)不为None则忽略。
2.3 实现
# -*- coding:utf-8 -*-
from sklearn.datasets import make_blobs
from sklearn.cluster import MeanShift, estimate_bandwidth
import numpy as np
import matplotlib.pyplot as plt
from itertools import cycle ##python自带的迭代器模块
##产生随机数据的中心
centers = [[1, 1], [-1, -1], [1, -1]]
##产生的数据个数
n_samples=10000
##生产数据
X, _ = make_blobs(n_samples=n_samples, centers= centers, cluster_std=0.6,
random_state =0)
##带宽,也就是以某个点为核心时的搜索半径
bandwidth = estimate_bandwidth(X, quantile=0.2, n_samples=500)
##设置均值偏移函数
ms = MeanShift(bandwidth=bandwidth, bin_seeding=True)
##训练数据
ms.fit(X)
##每个点的标签
labels = ms.labels_
print(labels)
##簇中心的点的集合
cluster_centers = ms.cluster_centers_
print('cluster_centers:',cluster_centers)
##总共的标签分类
labels_unique = np.unique(labels)
##聚簇的个数,即分类的个数
n_clusters_ = len(labels_unique)
print("number of estimated clusters : %d" % n_clusters_)
##绘图
plt.figure(1)
plt.clf()
colors = cycle('bgrcmykbgrcmykbgrcmykbgrcmyk')
for k, col in zip(range(n_clusters_), colors):
##根据lables中的值是否等于k,重新组成一个True、False的数组
my_members = labels == k
cluster_center = cluster_centers[k]
##X[my_members, 0] 取出my_members对应位置为True的值的横坐标
plt.plot(X[my_members, 0], X[my_members, 1], col + '.')
plt.plot(cluster_center[0], cluster_center[1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=14)
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
2.4 结果
2.5 实例扩展:利用Meanshift进行目标追踪
# -*- coding:utf-8 -*-
__author__ = 'Microcosm'
import cv2
import numpy as np
cap=cv2.VideoCapture(0)
# 获取第一帧
ret, frame = cap.read()
# 设置初始跟踪对象的窗口大小
# r,h,c,w = 120,100,253,100
# r, h, c, w = 180, 80, 140, 90
r, h, c, w = 0, 80, 0, 80
track_window = (c, r, w, h)
cv2.rectangle(frame, (c, r), (c + w, r + h), 255, 2)
cv2.imshow("frame", frame)
cv2.waitKey(0)
# 设置感兴趣的区域
roi = frame[r:r + h, c:c + w]
# cv2.imshow("roi",roi)
# cv2.waitKey(0)
hsv_roi = cv2.cvtColor(frame, cv2.COLOR_BGR2HSV)
mask = cv2.inRange(hsv_roi, np.array((0., 0., 32.)), np.array((180., 255., 255.)))
roi_hist = cv2.calcHist([hsv_roi], [0], None, [180], [0, 180])
cv2.normalize(roi_hist, roi_hist, 0, 255, cv2.NORM_MINMAX)
term_crit = (cv2.TERM_CRITERIA_EPS | cv2.TERM_CRITERIA_COUNT, 10, 1)
while (True):
ret, frame = cap.read()
if ret is True:
hsv = cv2.cvtColor(frame, cv2.COLOR_BGR2HSV)
dst = cv2.calcBackProject([hsv], [0], roi_hist, [0, 180], 1)
# 调用meanshift获取新的位置
ret, track_window = cv2.meanShift(dst, track_window, term_crit)
# 画出它的位置
x, y, w, h = track_window
cv2.rectangle(frame, (x, y), (x + w, y + h), 255, 2)
cv2.imshow("frame", frame)
k = cv2.waitKey(60) & 0xff
if k == 27:
break
# else:
# cv2.imwrite(chr(k)+".jpg",frame)
else:
break
cv2.destroyAllWindows()
cap.release()
3. Spectral Clustering原理及实现
3.1 Spectral Clustering原理
-
Spectral Clustering(SC,即谱聚类),是一种基于图论的聚类方法,它能够识别任意形状的样本空间且收敛于全局最有解,其基本思想是利用样本数据的相似矩阵进行特征分解后得到的特征向量进行聚类.它与样本特征无关而只与样本个数有关。
-
基本思路:将样本看作顶点,样本间的相似度看作带权的边,从而将聚类问题转为图分割问题:找到一种图分割的方法使得连接不同组的边的权重尽可能低(这意味着组间相似度要尽可能低),组内的边的权重尽可能高(这意味着组内相似度要尽可能高).
3.2 主要参数
因为是基于图论的算法,所以输入必须是对称矩阵。
-
主要参数(参数较多,详细参数)
-
n_clusters:聚类的个数。(官方的解释:投影子空间的维度)
-
affinity:核函数,默认是’rbf’,可选:“nearest_neighbors”,“precomputed”,"rbf"或sklearn.metrics.pairwise_kernels支持的其中一个内核之一。
-
gamma :affinity指定的核函数的内核系数,默认1.0
3.3 实现
from sklearn.datasets import make_blobs
from sklearn.cluster import spectral_clustering
import numpy as np
import matplotlib.pyplot as plt
from sklearn import metrics
from itertools import cycle ##python自带的迭代器模块
##产生随机数据的中心
centers = [[1, 1], [-1, -1], [1, -1]]
##产生的数据个数
n_samples=3000
##生产数据
X, lables_true = make_blobs(n_samples=n_samples, centers= centers, cluster_std=0.6,
random_state =0)
##变换成矩阵,输入必须是对称矩阵
metrics_metrix = (-1 * metrics.pairwise.pairwise_distances(X)).astype(np.int32)
metrics_metrix += -1 * metrics_metrix.min()
##设置谱聚类函数
n_clusters_= 4
lables = spectral_clustering(metrics_metrix,n_clusters=n_clusters_)
##绘图
plt.figure(1)
plt.clf()
colors = cycle('bgrcmykbgrcmykbgrcmykbgrcmyk')
for k, col in zip(range(n_clusters_), colors):
##根据lables中的值是否等于k,重新组成一个True、False的数组
my_members = lables == k
##X[my_members, 0] 取出my_members对应位置为True的值的横坐标
plt.plot(X[my_members, 0], X[my_members, 1], col + '.')
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
3.4 结果
4.Hierarchical Clustering原理及实现
4.1 Hierarchical Clustering原理
简介:Hierarchical Clustering(层次聚类):就是按照某种方法进行层次分类,直到满足某种条件为止。
主要分成两类:
a)凝聚:从下到上。首先将每个对象作为一个簇,然后合并这些原子簇为越来越大的簇,直到所有的对象都在一个簇中,或者某个终结条件被满足。
b)分裂:从上到下。首先将所有对象置于同一个簇中,然后逐渐细分为越来越小的簇,直到每个对象自成一簇,或者达到了某个终止条件。(较少用)
步骤:
a)将每个对象归为一类, 共得到N类, 每类仅包含一个对象. 类与类之间的距离就是它们所包含的对象之间的距离.
b)找到最接近的两个类并合并成一类, 于是总的类数少了一个.
c)重新计算新的类与所有旧类之间的距离.
d)重复第2步和第3步, 直到最后合并成一个类为止(此类包含了N个对象).
4.2 主要参数
sklearn.cluster.AgglomerativeClustering
-
n_clusters:聚类的个数
-
linkage:指定层次聚类判断相似度的方法,有以下三种:
-
ward:组间距离等于两类对象之间的最小距离。(即single-linkage聚类)
-
average:组间距离等于两组对象之间的平均距离。(average-linkage聚类)
-
complete:组间距离等于两组对象之间的最大距离。(complete-linkage聚类)
4.3 实现
from sklearn.datasets import make_blobs
from sklearn.cluster import AgglomerativeClustering
import numpy as np
import matplotlib.pyplot as plt
from itertools import cycle ##python自带的迭代器模块
##产生随机数据的中心
centers = [[1, 1], [-1, -1], [1, -1]]
##产生的数据个数
n_samples=3000
##生产数据
X, lables_true = make_blobs(n_samples=n_samples, centers= centers, cluster_std=0.6,
random_state =0)
##设置分层聚类函数
linkages = ['ward', 'average', 'complete']
n_clusters_ = 3
ac = AgglomerativeClustering(linkage=linkages[2],n_clusters = n_clusters_)
##训练数据
ac.fit(X)
##每个数据的分类
lables = ac.labels_
##绘图
plt.figure(1)
plt.clf()
colors = cycle('bgrcmykbgrcmykbgrcmykbgrcmyk')
for k, col in zip(range(n_clusters_), colors):
##根据lables中的值是否等于k,重新组成一个True、False的数组
my_members = lables == k
##X[my_members, 0] 取出my_members对应位置为True的值的横坐标
plt.plot(X[my_members, 0], X[my_members, 1], col + '.')
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
4.3 结果
5.DBSCAN原理及实现
5.1 DBSCAN原理
简介:
DBSCAN(Density-Based Spatial Clustering of Applications with Noise,具有噪声的基于密度的聚类方法)是一种基于密度的空间聚类算法。该算法将具有足够密度的区域划分为簇(即要求聚类空间中的一定区域内所包含对象的数目不小于某一给定阈值),并在具有噪声的空间数据库中发现任意形状的簇,它将簇定义为密度相连的点的最大集合。
步骤:
a)遍历所有点,寻找核心点
b)连通核心点,并且在此过程中扩展某个分类集合中点的个数

在上图中,第一步就是寻找红色的核心点,第二步就是用绿色箭头联通红色点。图中点以绿色线条为中心被分成了两类。没在黑色圆中的点是噪声点。
5.2 主要参数
DBSCAN需要二个参数:扫描半径 (eps)和最小包含点数(min_samples)
5.3 实现
from sklearn.datasets import make_blobs
from sklearn.cluster import DBSCAN
import numpy as np
import matplotlib.pyplot as plt
from itertools import cycle ##python自带的迭代器模块
from sklearn.preprocessing import StandardScaler
##产生随机数据的中心
centers = [[1, 1], [-1, -1], [1, -1]]
##产生的数据个数
n_samples = 750
##生产数据:此实验结果受cluster_std的影响,或者说受eps 和cluster_std差值影响
X, lables_true = make_blobs(n_samples=n_samples, centers=centers, cluster_std=0.4,
random_state=0)
##设置分层聚类函数
db = DBSCAN(eps=0.3, min_samples=10)
##训练数据
db.fit(X)
##初始化一个全是False的bool类型的数组
core_samples_mask = np.zeros_like(db.labels_, dtype=bool)
'''
这里是关键点(针对这行代码:xy = X[class_member_mask & ~core_samples_mask]):
db.core_sample_indices_ 表示的是某个点在寻找核心点集合的过程中暂时被标为噪声点的点(即周围点
小于min_samples),并不是最终的噪声点。在对核心点进行联通的过程中,这部分点会被进行重新归类(即标签
并不会是表示噪声点的-1),也可也这样理解,这些点不适合做核心点,但是会被包含在某个核心点的范围之内
'''
core_samples_mask[db.core_sample_indices_] = True
##每个数据的分类
lables = db.labels_
##分类个数:lables中包含-1,表示噪声点
n_clusters_ = len(np.unique(lables)) - (1 if -1 in lables else 0)
##绘图
unique_labels = set(lables)
'''
1)np.linspace 返回[0,1]之间的len(unique_labels) 个数
2)plt.cm 一个颜色映射模块
3)生成的每个colors包含4个值,分别是rgba
4)其实这行代码的意思就是生成4个可以和光谱对应的颜色值
'''
colors = plt.cm.Spectral(np.linspace(0, 1, len(unique_labels)))
plt.figure(1)
plt.clf()
for k, col in zip(unique_labels, colors):
##-1表示噪声点,这里的k表示黑色
if k == -1:
col = 'k'
##生成一个True、False数组,lables == k 的设置成True
class_member_mask = (lables == k)
##两个数组做&运算,找出即是核心点又等于分类k的值 markeredgecolor='k',
xy = X[class_member_mask & core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', c=col, markersize=14)
'''
1)~优先级最高,按位对core_samples_mask 求反,求出的是噪音点的位置
2)& 于运算之后,求出虽然刚开始是噪音点的位置,但是重新归类却属于k的点
3)对核心分类之后进行的扩展
'''
xy = X[class_member_mask & ~core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', c=col, markersize=6)
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
5.4 结果
6.Birch原理及实现
6.1 Birch原理
Birch(利用层次方法的平衡迭代规约和聚类):就是通过聚类特征(CF)形成一个聚类特征树,root层的CF个数就是聚类个数。
聚类特征(CF):每一个CF是一个三元组,可以用(N,LS,SS)表示.其中N代表了这个CF中拥有的样本点的数量;LS代表了这个CF中拥有的样本点各特征维度的和向量,SS代表了这个CF中拥有的样本点各特征维度的平方和。
6.2 主要参数
-
n_clusters :聚类的目标个数。(可选)
-
threshold :扫描半径(个人理解,官方说法比较绕口),设置小了分类就多。
-
branches_factor:每个节点中CF子集群的最大数量,默认为50。
6.3 实现
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
from sklearn.cluster import Birch
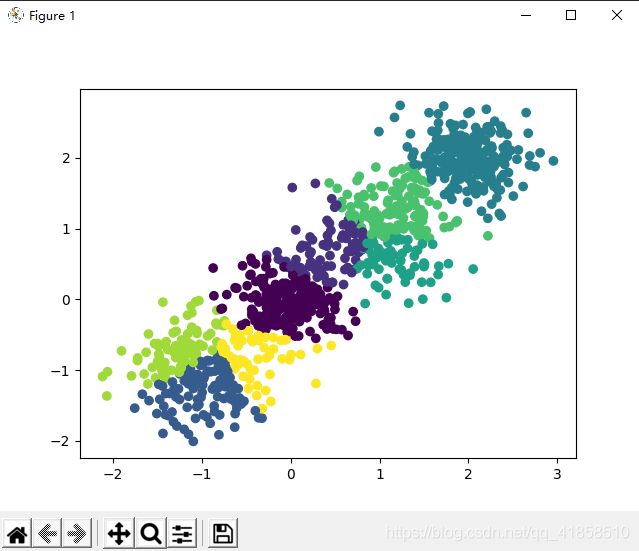
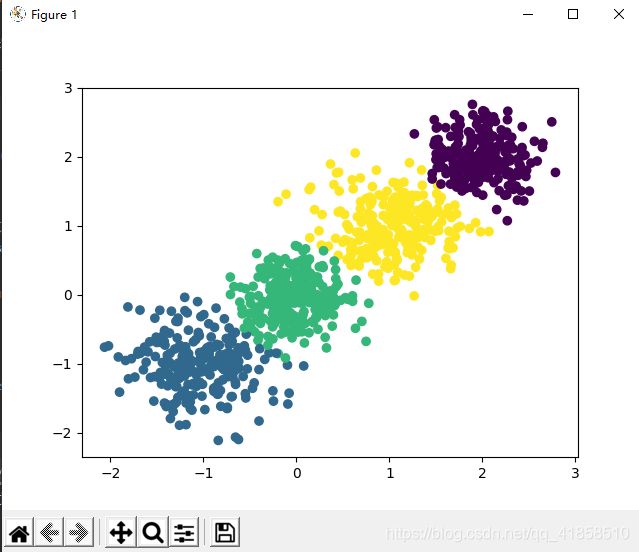
# X为样本特征,Y为样本簇类别, 共1000个样本,每个样本2个特征,共4个簇,簇中心在[-1,-1], [0,0],[1,1], [2,2]
X, y = make_blobs(n_samples=1000, n_features=2, centers=[[-1,-1], [0,0], [1,1], [2,2]], cluster_std=[0.4, 0.3, 0.4, 0.3],
random_state =9)
##设置birch函数
birch = Birch(n_clusters = None)
##训练数据
y_pred = birch.fit_predict(X)
##绘图
plt.scatter(X[:, 0], X[:, 1], c=y_pred)
plt.show()
6.4 结果
7 GaussianMixtureModel原理及实现
7.1 GaussianMixtureModel原理
正太分布也叫高斯分布,正太分布的概率密度曲线也叫高斯分布概率曲线。
GaussianMixtureModel(混合高斯模型,GMM)。
聚类算法大多数通过相似度来判断,而相似度又大多采用欧式距离长短作为衡量依据。而GMM采用了新的判断依据:概率,即通过属于某一类的概率大小来判断最终的归属类别。
GMM的基本思想就是:任意形状的概率分布都可以用多个高斯分布函数去近似,也就是说GMM就是有多个单高斯密度分布(Gaussian)组成的,每个Gaussian叫一个"Component",这些"Component"线性加成在一起就组成了 GMM 的概率密度函数,也就是下面的函数
7.2 主要参数
-
n_components :高斯模型的个数,即聚类的目标个数
-
covariance_type : 通过EM算法估算参数时使用的协方差类型,默认是"full"
-
full:每个模型使用自己的一般协方差矩阵
-
tied:所用模型共享一个一般协方差矩阵
-
diag:每个模型使用自己的对角线协方差矩阵
-
spherical:每个模型使用自己的单一方差
7.3 实现
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
from sklearn.mixture import GaussianMixture
# X为样本特征,Y为样本簇类别, 共1000个样本,每个样本2个特征,共4个簇,簇中心在[-1,-1], [0,0],[1,1], [2,2]
X, y = make_blobs(n_samples=1000, n_features=2, centers=[[-1,-1], [0,0], [1,1], [2,2]], cluster_std=[0.4, 0.3, 0.4, 0.3],
random_state = 0)
##设置gmm函数
gmm = GaussianMixture(n_components=4, covariance_type='full').fit(X)
##训练数据
y_pred = gmm.predict(X)
##绘图
plt.scatter(X[:, 0], X[:, 1], c=y_pred)
plt.show()