今天跟大家分享的是三月11日发表在clinical cancer research(IF:8.9)杂志上的一篇关于头颈癌免疫浸润亚型文章。文章整合了多组学、多平台数据进行分析,很值得学习哦!
ntegrative analysis of multi-omics data identified EGFR and PTGS2 as key nodes in a gene regulatory network related to immune phenotypes in head and neck cancer
多组学数据综合分析识别头颈癌免疫表型相关的调控网络中关键节点:EGFR、PTGS2
目的:肿瘤的恶性进展表现出免疫效应和耐受的紧密协调的平衡。然而驱动肿瘤免疫表型的形成和维持的分子原理仍有待阐明。
实验设计:基于PD-L1 and IFN-g表达相关的免疫细胞子集训练了新的分子分类器,该分类器可区分高(cluster A)或低细胞免疫毒性(sub-cluster B3)子集。该研究基于多组学数据综合性分析以确定遗传和表观遗传景观差异,以及对不同免疫表型之间差异表达基因的影响。使用 LASSO-Cox回归模型构建了免疫检查位点抑制(ICI)的预后基因标志。
结论:研究使用的数据突显了肿瘤免疫表型建立过程中遗传和表观遗传事件之间的复杂相互作用,并提供了ICI治疗失败风险较高的HNSCC患者可能是由于EGFR的抑制实验证据。
材料和方法
一、表达和临床队列
1、TCGA:33个独立TCGA癌症队列的RNA表达及临床数据:https://portal.gdc.cancer.gov/;TCGA-HNSC蛋白表达数据:https://www.tcpaportal.org/tcpa
2、GEO:HNSCC转录组验证集:GSE39368 , GSE40774 , GSE65858,HIPO-HNC (GSE117973未公开)
3、TCGA-HNSC 功能性miRNA、lncRNA数据:https://portal.gdc.cancer.gov/
4、GEO细胞系数据:GSE45891
5、PD-1阻断治疗GEO转录组数据:GSE93157
二、CIBERSORT免疫细胞得分
CIBERSORT基于基因表达队列计算绝对免疫细胞评分(22细胞),https://cibersort.stanford.edu/
三、Lasso回归分析
用Lasso回归分析对于预后相关的差异基因进行优先级排序,构建了包含10个基因预后模型并GSE93157队列进行了模型的测试。
四、聚类分析
样本绝对免疫细胞评分值经过 ln(x + 1)转换,在经过Euclidean distance和Ward linkage进行聚类。ClustVis在线工具可视化热图。
五、体细胞突变分析
TCGA-HNSC体细胞突变数据获取:cBioPortal(http://www.cbioportal.org/),经过MutSig分析得到q<0.05且体细胞突变频率> 5%的候选基因。比较它们在不同免疫表型之间的相对分布。体细胞突变模式由R包“ ComplexHeatmap”分析。
六、拷贝数变异分析
TCGA-HNSC拷贝数数据获取:http://www.firebrowse.org/。Segment_Mean值>0.2为扩增,<0.2为缺失。IGV_2.4.19工具生成CNV summary图,R 包Rciorcos生成CNV圈图。
七、DNA甲基化分析
TCGA-HNSC DNA甲基化数据获取:https://gdc.xenahubs.net/download/TCGA-HNSC.methylation450.tsv.gz/。使用R包wateRmelon对甲基化数据归一化,R包minfi筛选差异甲基化探针。
八、免疫组化染色(IHC)
使用兔的单克隆anti-EGFR抗体对HIPI-HNSC队列FFPE肿瘤切片进行IHC染色。Anti-EGFR抗体的特异性通过HNSC样本的连续FFPE组织切片的兔IgG同型对照抗体或无第一抗体的IHC染色证实。EGFR免疫分数是染色强度与阳性染色肿瘤细胞总数百分比的乘积,范围为0至100%。
九、统计
由SPSS Statistics 25软件和R软件(版本3.5.1以上)进行统计分析。P值<0.05被认为具有统计学意义。
十、研究批准
HIPO-HNC队列患者于2012年至2016年在德国海德堡大学医院接受治疗,根据海德堡大学伦理委员会批准的S-206 / 2011和S220 / 2016协议获取并分析患者样本,并书面所有参与者的知情同意。
结果
一、HNSCC样本中与PD-L1、IFN-g表达相关的分子免疫亚型
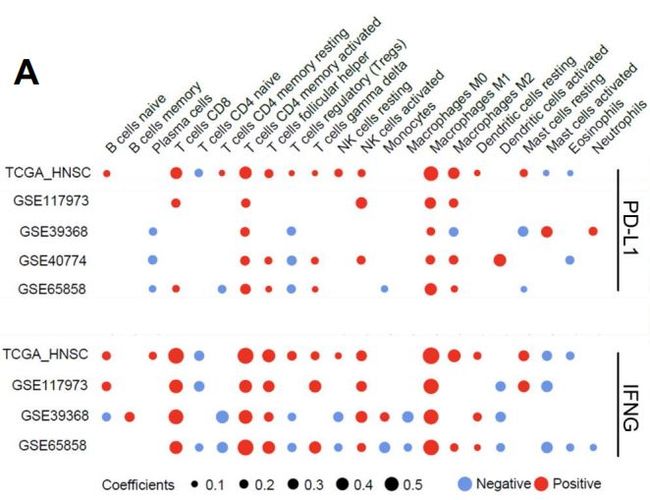
基于5个包含1106个样本的独立基因表达队列(训练集:TCGA-HNSC (n =500)、验证集:GSE117973、GSE39368、GSE40774 、GSE65858)使用CIBERSORT评估单个样本中不同免疫细胞比例。计算PD-L1(CD274)、IFN-g(IFNG)表达和免疫细胞比例之间的Spearman相关性(图1A)。Lasso回归分析筛选了上一步中与PD-L1、IFNG表达最相关的8种免疫细胞,最终得到5种用于后续分析。该研究还使用Meta分析确认了Lasso选择的免疫细胞类型的比例升高与各HNSCC队列中PD-L1、IFNG高表达显著相关。
图1A
二、通过选择免疫细胞子集来将亚群分层
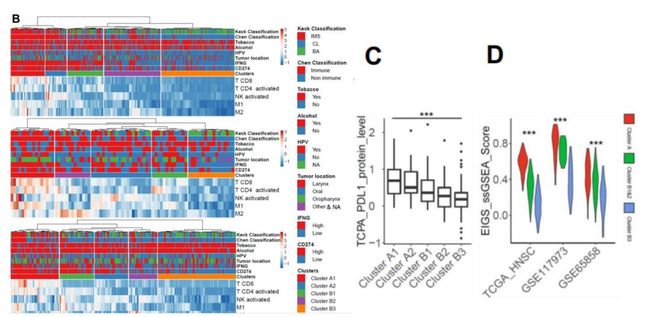
基于5个免疫细胞类型对TCGA-HNSC队列进行无监督聚类,主要划分成A、B两类(见图1B:从上到下依次为TCGA-HNSC、GSE117973 、GSE 65858队列)。亚型A显示出较高的免疫细胞浸润比例,且PD-L1、IFNG表达水平及PD-L1蛋白水平较高(图1C)。由于激活的NK细胞的绝对得分存在显着差异,因此将簇A分为两个子簇A1和A2。B簇表现出较低免疫细胞浸润比例和较高的异质性得分。这对于B3尤为明显,子群B1则展现出活化CD4-T细胞和MI巨噬细胞中等浸润及更高的PD-L1、IFNG表达水平(图1B)。
因此,研究将亚型A定义为高细胞毒性免疫表型,亚型B3为低细胞毒性免疫表型。ssGSEA得分A中显著高于B3(图1D)。在不同的HNSC队列中都有这样的现象,那是否对于其他亚型也同样如此呢。该研究对25个TCGA队列(n>100)做了同样的研究的研究,排除了CD4-T细胞在>50%的样本都缺失的癌型后,无监督聚类分析发现了同样的聚类模式。
图1B-D
三、MHC class I基因在不同免疫表型中的表达差异
通过MHC class I复合物呈递肿瘤相关抗原是免疫监视的前提条件,并且对于针对免疫检查点的免疫疗法的临床反应至关重要。因此,研究评估了TCGA -HNSC和独立验证队列免疫表型中编码MHC class I蛋白的B2M和人类白细胞抗原(HLA)基因的转录水平。结果可见A1到B3的表达逐渐下降(图S8A),表明肿瘤细胞上抗原呈递受损,是亚群B3免疫监视的逃逸机制。在其他TCGA队列得到了同样的结果(图S8B-C)。
图S8
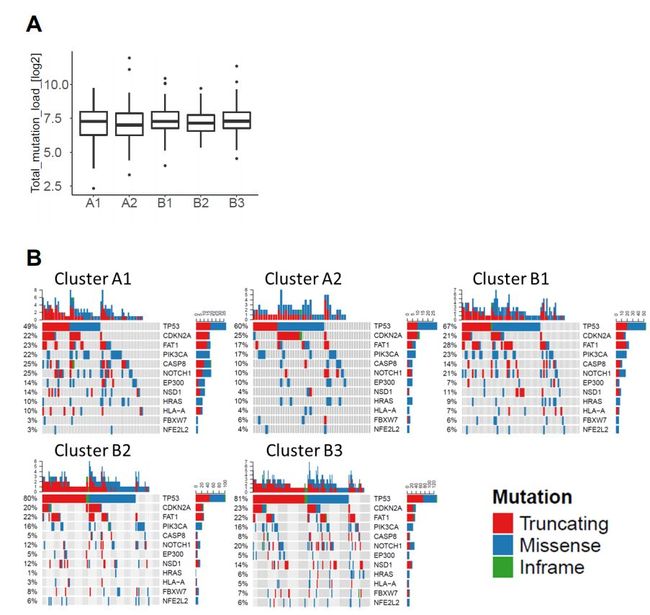
四、不同免疫表型之间的体细胞突变差异
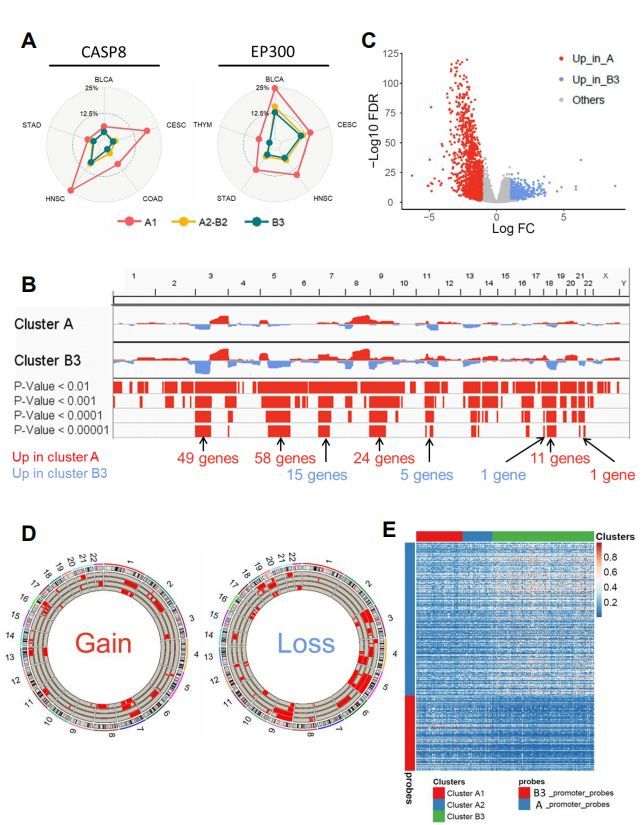
基于TCGA-HNSC队列分析了各免疫表型的体细胞突变。尽管总的突变负荷在整体上没有显著的差异(图S10A)。但与B3比亚型A CASP8突变频率更高(A1尤为显著),亚型A1-B3 EP300突变频率逐渐降低,TP53逐渐升高(图S10B)。与HNSCC相似的是4个TCGA队列(BLCA, CESC, STAD, COAD) CASP8突变频率相对较高,4个队列 (BLCA, CESC, STAD, THYM) EP300突变频率由A1-B3逐渐降低(图2A)。
图S10
五、不同免疫表型之间的基因组拷贝数差异
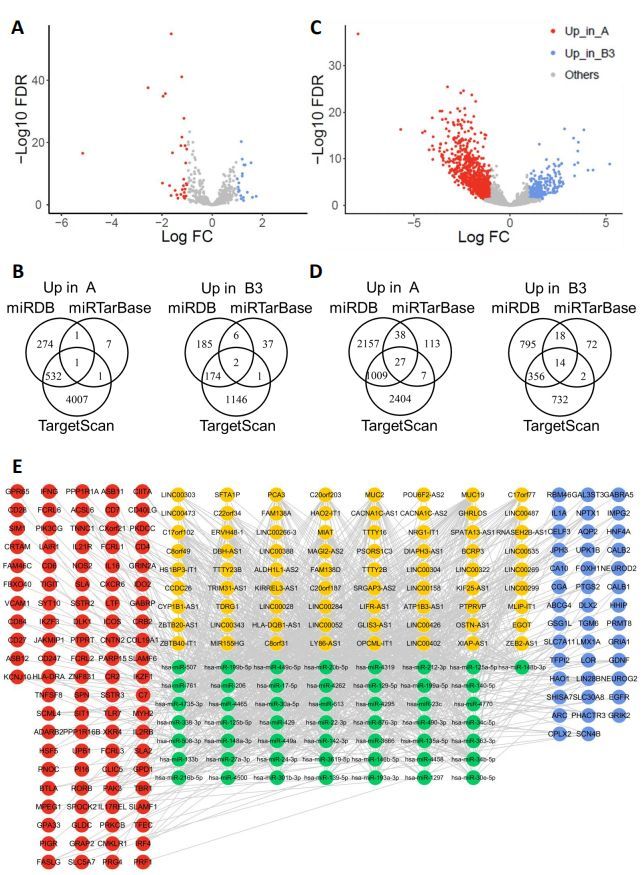
TCGA-HNSC队列基因组改变分析表明,在几个热点区域与亚型A相比亚型B3在(染色体7p,11q,13q和18p)显著扩增或在(染色体3p,5q,9p和18q)显著缺失(图2B)。为了评估拷贝数变异是都影响基因表达,筛选了亚型A和亚型B3的差异基因,发现1609个基因在亚型A中上调,707个在B3中上调(图2C)。GO富集分析发现A上调的基因参与免疫细胞增殖和活化。而B3上调基因则参与了cornification、发育、分化相关功能。亚型A中143个上调基因由亚型B3中缺失频率显著更高的基因组区域编码。B3中21个上调基因被该类型中显著拷贝数扩增的基因组区域编码(图2B)。与HNSCC相似,与亚型A相比,几个TCGA队列(LUSC,LUAD和STAD)共享了3p,5q或9p染色体的显着富集缺失,以及亚型B3中7p染色体的拷贝数增加(图2D)。小结:这些数据表明通过获得特定的体细胞突变,免疫亚型A肿瘤中的癌细胞适应了肿瘤浸润淋巴细胞的存在。相反,亚群B3肿瘤中的癌细胞通过影响淋巴细胞激活和增殖调节因子的基因组改变而逃脱了免疫监视。
图2
六、不同免疫表型之间的DNA甲基化差异
为了探索DNA甲基化对肿瘤免疫表型形成和维持的影响,研究分析了TCGA-HNSC队列的总体甲基化数据。筛选了亚型A和亚型B3间2796种差异甲基化探针(-0.35> log2 FC> 0.35,FDR <0.001),其中267个在B3中具有显着更高的beta值的探针,他们位于亚型A中152个差异上调基因的近端启动子(图2E)。另外130个在亚型A中具有显着更高的beta值的探针,他们位于亚型B3中69个差异上调基因的近端启动子(图2E)。
七、不同免疫表型之间的miRNA表达差异
基于TCGA-HNSC队列,筛选免疫亚型A和B3之间的差异表达功能miRNA(DEFM、-1>log2FC>1, FDR < 0.05) (图3A)。B3亚型高表达的miRNA 关联的535个DEFM-DEG关系对中(三个在线靶基因数据库至少两个证实的靶向关系)347个DEGs在亚型A中上调。A亚型高表达的miRNA 关联的183个DEFM-DEG关系对中133个DEGs在亚型B3中上调(图3B)。
图3
八、不同免疫表型之间的lncRNA表达差异
基于TCGA-HNSC队列,筛选免疫亚型A和B3之间的差异表达lncRNA(-1>log2
FC>1, FDR < 0.05) (图3C)。根据数据库预测到了3276个lncRNA-miR关系对,包括74个lncRNA和208个miRNA(高度保守的miR家族的一部分)。其中在亚型A中1081个关系对中关联的423个DEGs上调,亚型B3中390个关系对中关联的168个DEGs上调(图3D)。基于差异表达lncRNA(黄色点) 、靶向MiR(绿色点)和DEGs(红色点:在亚型A中上调、蓝色点:在亚型B3上调)构建了不同免疫表型潜在的分子特征复杂网络(图3D)。
九、DEGs受多种遗传和表观遗传调控模式影响
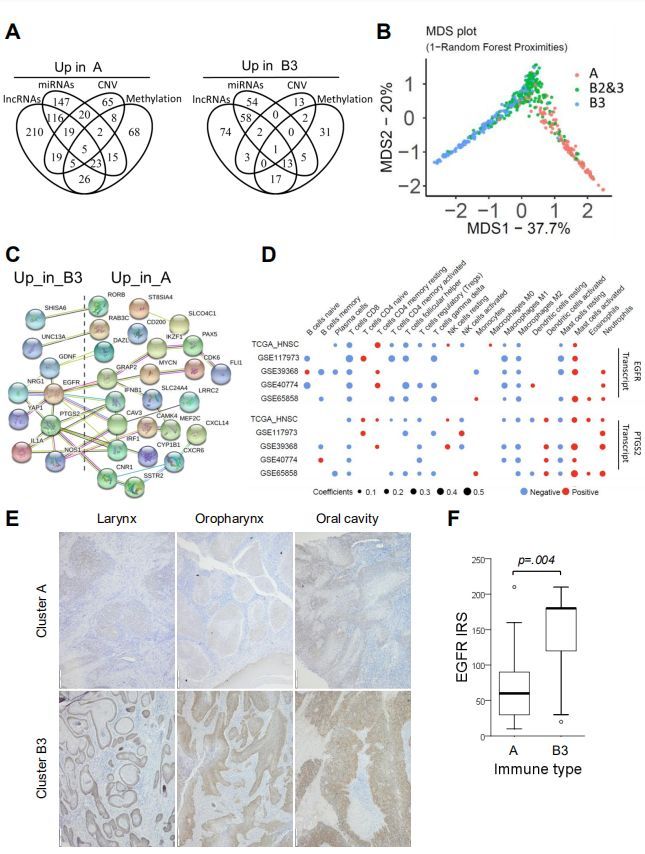
研究假设不同免疫表型的形成和维持是由于不同的遗传和表观遗传调控模式的影响。在以上的3/4分析中识别得到了70个基因,其中54 DEGs在亚型A中上调,16个在亚型B3中上调(图4A)。如预期的一样,TCGA-HNSC队列中亚型A和B3间基于这些基因集的ssGSEA得分有显著差异,在其它癌型中也得到了验证。并且在TCGA-HNSC队列中随机森林印迹支持了这一点(图4B)。STRING数据库进行PPI互作分析显示EGFR、PTGS2是网络中关键的节点。
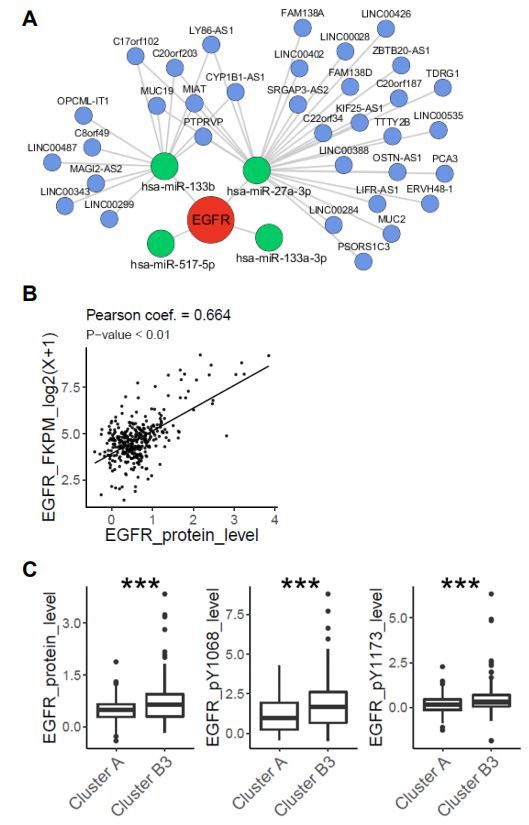
与亚型A比,B3的EGFR表达明显较高。这对于TCGA-HNSC队列几乎是唯一的,其它EGFR成员(ERBB2, ERBB3)在其它TCGA癌型队列亚型之间表达也是差异的。与其它TCGA癌型队列相比,TCGA-HNSC队列亚型间PTGS2有更显著的表达差异。EGFR转录水平可能受7p11染色体拷贝数扩增(图2B)和几个miRs和lncRNAs差异表达的影响。Pearson相关性分析证实了EGFR转录本与蛋白水平之间呈正相关(蛋白组图S15B),与亚型A相比,亚型B3中的EGFR蛋白及磷酸化水平显着更高( 图S15C)。此外,Spearman相关分析证实了EGFR或PTGS2转录水平与TCGA-HNSC和大多数验证队列先前选择的免疫细胞亚型之间显着负相关(图4D)。GSE117973队列的肿瘤切片IHC染色进一步证实与亚型A相比,来自亚型B3的肿瘤癌细胞中的EGFR表达更高,而与肿瘤部位无关(图4E-F)。
图4
图S15
十、EGFR抑制剂影响不同免疫表型相关的DEGs的表达
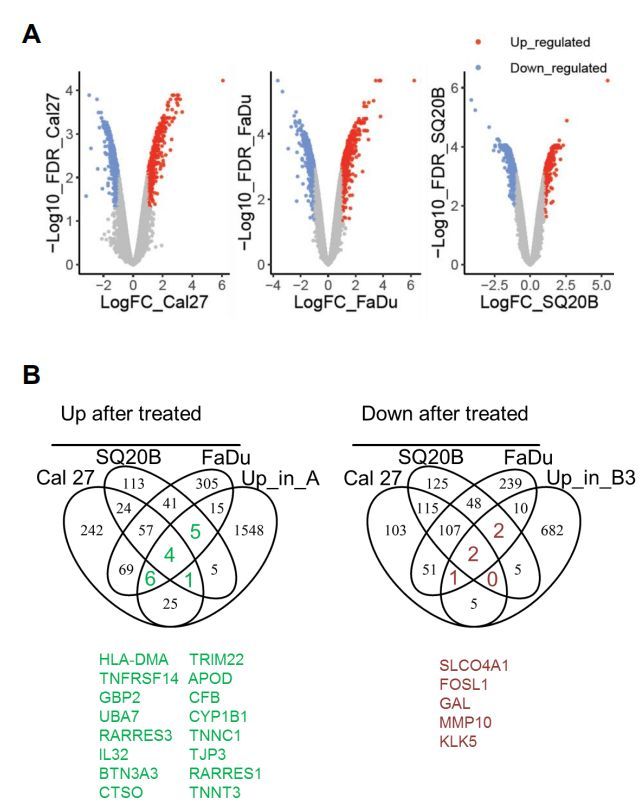
为了证明EGFR信号传导调节亚型A和B3之间的DEGs的表达,研究分析了erlotinib治疗后三种HNSCC细胞系的转录组数据(GSE45891)。发现,在至少三个HNSCC细胞系中的两个中,erlotinib增加了原本在免疫表型A中有较高的表达16个DEGs的表达(lower EGFR expression),而erlotinib减少了原本在亚型B3中5个具有较高表达的DEG的表达(lower EGFR expression)(图S16)。分析结果表明了EGFR表达通过阻止细胞毒性免疫细胞的活化and/or浸润来调节免疫微环境的关键角色。
图S16
十一、免疫表型相关的DEGs影响ICI反应
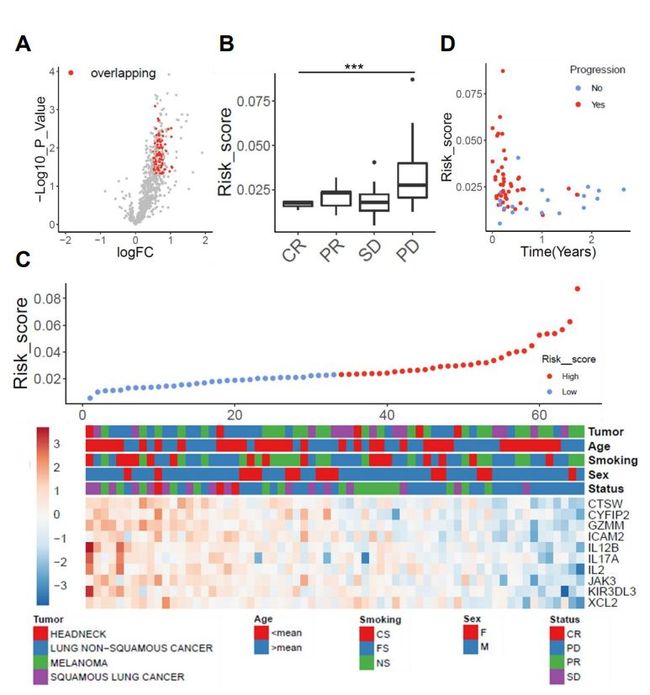
为了探索亚型A和B3之间的DEG是否可作为ICI(免疫检查位点抑制)反应的预测生物标记,研究分析了GSE93157队列,该数据提供了用anti-PD1抗体治疗的非小细胞肺癌(NSCLC),HNSCC和黑色素瘤患者的免疫基因检测结果。与progressive disease(PD)相比,总共有200种免疫基因差异表达(logFC> 0.5,p值<0.05),在Nonprogressive disease(CR,PR和SD)中的表达明显更高。他们中有一半为在TCGA-HNSC队列亚群A和亚群B3的DEGs(图5A)。
使用LASSO Cox回归算法对最相关的预后候选基因进行优先排序,并选择了前10构建回归风险模型(图S18A-C)。在四组反应样本中,PD的风险评分显着高于所有其他组(图5B)。正如预期的那样,增加的风险评分与不良的无进展生存期相关(图5C-D)。
图5
至此,全文赏析就结束了。该研究在不同的数据集TCGA-HNSC、GEO-HNSCC队列中识别了免疫细胞浸润的两个特殊的免疫亚型A、B3。并且在TCGA其它癌型中也发现了同样的规律,接着分析了两个亚型在mRNA表达、体细胞突变、拷贝数变异、DNA甲基化、miRNA表达、lncRNA表达、免疫治疗、蛋白表达等方面的全面的研究了两个免疫亚型的分子机制。数据队列多、组学全、分析广使得文章内容丰富。