前沿
表观遗传学,包括组蛋白共价修饰(covalent histone modification)、DNA甲基化修饰(DNA methylation)、RNA甲基化修饰(RNA methylation)、基因组印记(genomic imprinting)、基因沉默(gene silencing)、RNA编辑(RNA editing)及非编码RNA(noncoding RNA)等,是指在核苷酸序列不发生改变的情况下,生物表型或基因表达发生了稳定的可遗传变化。
RNA甲基化作为表观遗传学研究的重要内容之一,是指发生在RNA分子上不同位置的甲基化修饰现象,6-甲基腺嘌呤(N6-methyladenosine,m6A)和5-甲基胞嘧啶(C5-methylcytidine,m5C)是真核生物中最常见的两种RNA转录后修饰。RNA甲基化在调控基因表达、剪接、RNA编辑、RNA稳定性、控制mRNA寿命和降解等方面可能扮演重要角色。 相对于DNA甲基化,RNA甲基化更加复杂、种类繁多、普遍存在于各种高级生物中。
已知绝大部分真核生物中,mRNA在5’ Cap处存在甲基化修饰,作用包括维持mRNA稳定性、 mRNA前体剪切、多腺苷酸化、 mRNA运输与翻译起始等。而3’ polyA发生的修饰有助于出核转运、翻译起始以及与polyA结合蛋白⼀起维持mRNA的结构稳定。但是这些修饰只发生mRNA的头部和尾部,关于RNA的内部修饰(internal modification)在许多种类的RNA中都有发⽣。无论是mRNA还是lncRNA,都大量存在m6A修饰。m6A能够加速mRNA前体的加工时间,加快mRNA在细胞中的转运速度和出核速度。主要学习研究较多的m6A。RNA的m6A甲基化⼀共有大三类酶参与:Writers、 Erasers和Readers,需要相关研究的可以学习相关文献。
如何检测m6A
检测m6A的方法非常多,如包括MeRIPseq、 miCLIP-seq、 SCARLET、 LC-MS/MS等。2012年之后,两篇发表于Nature和Cell上的论⽂可以说是第⼀次从转录水平上,大范围高通量地鉴定了人和小鼠m6A的甲基化水平(Dominissini 2012和Meyer 2012)。这两篇独立发表的论文采用的核心方法就是将m6A抗体与带有m6A的mRNA片段相结合后进行高通量测序。通过对下机数据的分析,来鉴定mRNA上m6A程度较高的区域,分辨率约为100nt。这种方法我们称之为MeRIP-seq(methylated RNA immunoprecipitation sequencing)或m6A-seq。
MeRIP-seq建库步骤:
1. 提取total RNAs,并用Oligo-dT磁珠对total RNAs带有polyA的mRNA进行富集(通常要求Total RNA 300ug,人鼠可以做微量2ug 但结果可能会出现map率低dup率高 建库步骤与常量也有区别);
2. 用磁珠进行富集,得到带有polyA的mRNA。之后加入片段化试剂,将完整的mRNA进行片段化。或者使用超声波仪直接进行片段化;
3. 将片段化后的RNA分成两份。⼀份加入带有m6A抗体的免疫磁珠,对含有m6A甲基化的mRNA片段进⾏富集。另⼀份作为control,直接构建类似常规的转录组测序文库(这一步就是IP步骤,片段化程度、抗体抓取效率都会影响到后期实验结果;这里的control通常称为Input);
4. 对m6A抗体免疫磁珠进行富集,带有m6A的mRNA片段进行回收后,按照转录组的建库流程构建常规的测序文库;
5. 分别将构建好的2个测序文库,即m6A-seq library和RNA-seq library分别进行高通量测序。测序平台保持一致,推荐Hiseq X ten或Novaseq;
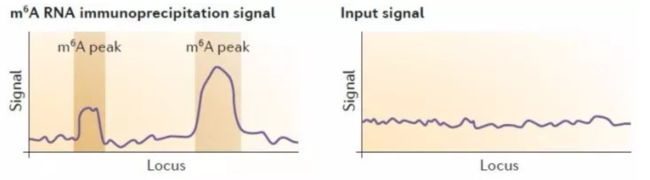
6. 对下机数据进行生物信息学分析,对发生m6A甲基化程度较高的区域进行peak calling。由于不能做到单个碱基的分辨率,所以只能对大致的区域进行分析。从下图中我们可以发现,与右侧常规的转录组测序结果相比,在基因上有两处区域存在非常明显的高甲基化峰;
7.接下来会进行一些常规分析,如peak区域基因注释,差异peak分析。
以上就是关于m6A-seq的标准步骤,现在是不是对m6A-seq有了一个非常直观的认识呢? 再次强调下,这种测序方法只能鉴定高甲基化的区域,并不能做到单碱基的分辨率。
m6A研究思路
思路1 老数据挖掘
第一步:先从原有的转录组数据中,挖掘到差异表达的甲基化酶;
第二步:对挖掘到的甲基化酶如METTL3或FTO等进⾏qPCR验证,并进行m6A-seq分析哪些基因甲基化水平发生改变;
第三步:在细胞(动物模型可选)中对这些酶进行敲低和过表达,进行常规的qPCR和WB检测相关酶表达情况,并用LC-MS/MS法检测RNA整体m6A水平;
第四步:继续对这些敲低和过表达的细胞进行转录组测序/小RNA测序或表达谱芯片/小RNA芯片,分析哪些基因出现差异表达变化和可变剪切变化;
第五步:找到甲基化酶调控的靶基因,进行敲低和过表达,看甲基化酶缺陷的细胞或动物模型表型能否补救;
第六步:在确定上一步靶基因确实受到甲基化酶调控后,对靶基因上的motif进行点突变后进行验证;
第七步:鉴定新型的甲基化酶(可选)。
思路2 研究甲基化修饰差异基因
第一步:直接进行m6A-seq和转录组测序,找到时间顺序或差异表达的基因并用qPCR、 WB等⽅法验证,此外找到m6A有差异的基因;
第二步:对甲基化酶进行敲低和过表达,检测RNA整体的m6A水平,之后可进行转录组或小RNA测序等方法检验甲基化酶敲低和过表达对mRNA或miRNA整体的影响,并着重研究第⼀步中感兴趣的m6A有差异的靶基因;
第三步:对靶基因进行敲低或过表达,是否能够对甲基化酶异常表达后的表型进⾏恢复;
第四步:对靶基因上motif进行点突变后进⼀步确认直接受到甲基化酶调控;
第五步:鉴定新型的甲基化酶(可选)。
当然根据不同的研究目的还有许多其他的研究思路,可根据自身实验设计进行延申和拓展。m6A相关SCI论文根据不同实验手段IF2~20不等,实验手段:m6A-seq、转录组测序/表达谱芯片、 LC-MS/MS 或 m6A 比色法、小RNA 测序/小RNA芯片、qPCR、 WB、敲降/过表达、靶基因验证、动物实验、临床实验/药物实验等。
学习资源来源网络,侵删。
参考学习:
1、高通量RNA甲基化测序数据处理与分析研究进展
2、RNA修饰检测技术
Roundtree, Ian A et al. “Dynamic RNA Modifications in Gene Expression Regulation.” Cell vol. 169,7 (2017): 1187-1200. doi:10.1016/j.cell.2017.05.045
Helm, M, & Y. Motorin. "Detecting RNA modifications in the epitranscriptome: predict and validate.” Nature Reviews Genetics 18.5 (2017):275.