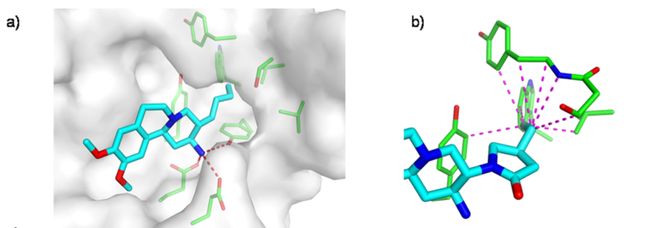
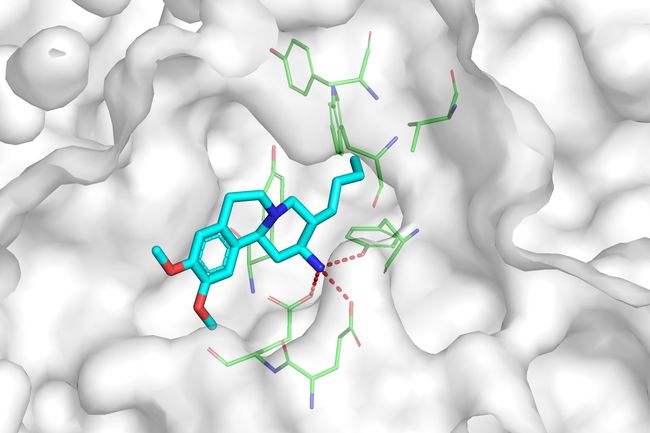
在本文中,我们尝试模仿绘制文献中的Figure 2a,所选文献为J. Med. Chem. 2016, 59, 4087−4102,原图如下所示:
本例中使用的PDB code: 3oc0
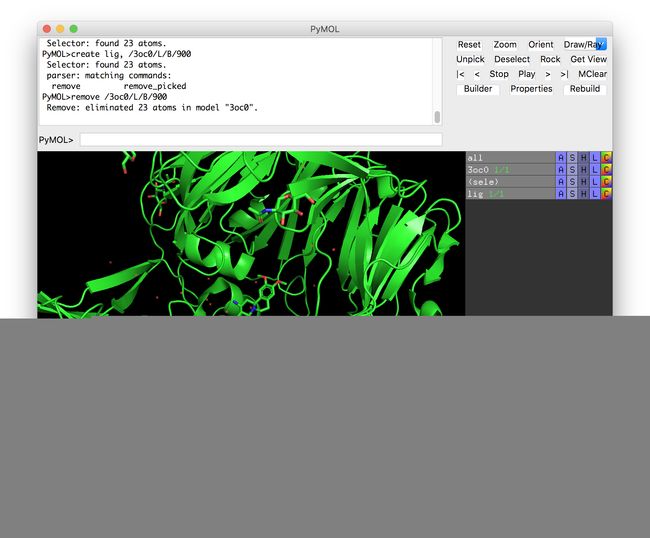
首先加载大分子,使用fetch命令可直接从PDB数据库下载。为了方便操作,我们先把大分子和小分子分成两个对象保存:
fetch 3oc0
create lig, /3oc0/G/A/900
remove /3oc0/G/A/900
现在就可以看到两个对象,一个是除去配体的大分子,另一个是小分子:
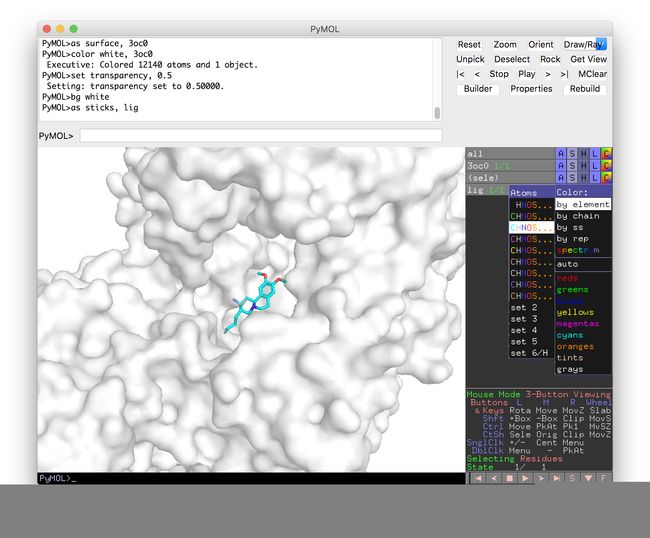
改变大分子和配体的显示样式:
bg white //背景改为白色
as surface, 3oc0 //显示大分子表面
color white, 3oc0 //将颜色改为白色
set transparency, 0.5 //设置透明度为50%
点击右侧控制台的lig对象的C项,选择by element,碳原子为cyan。
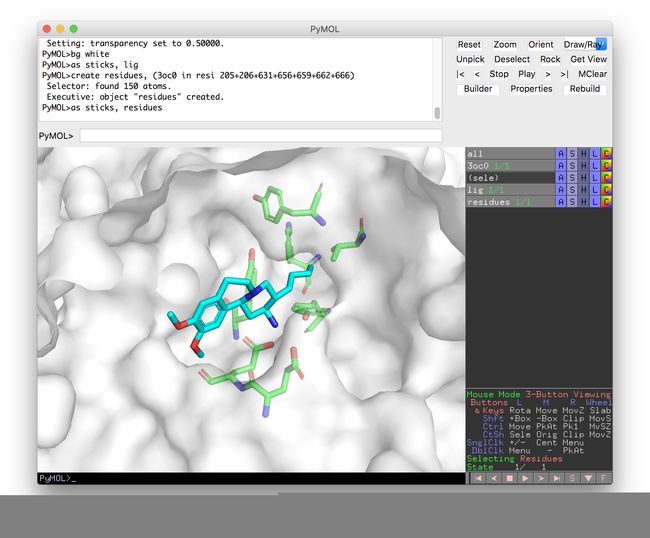
将我们需要的氨基酸残基复制到新对象中
create residues, (3oc0 in resi 205+206+631+656+659+662+666)
此处resi意为residue identifier,即残基的编号。
改变残基显示样式
as sticks, residues
与lig相同,颜色选择by element,不过这次的碳原子选green。
现在你可以看到,已经有文献里图片的内味了。
文献中的残基要比默认值更细,接下来修改粗细:
set_bond stick_radius, 0.1, residues
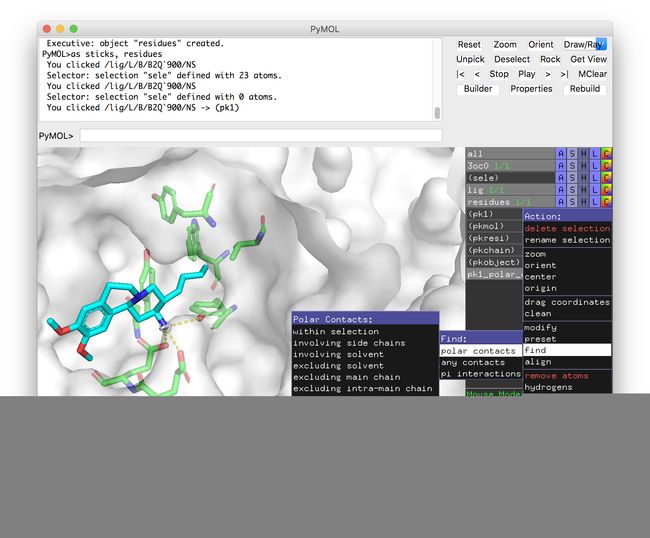
显示氢键:可以看到,文献中的图片只有一个氨基的氢键显示了出来,因此我们需要按住control键,再用鼠标中键单机该氮原子,现在右侧可以看到(pk1)对象,在其A项中选择find -> polar contacts -> to any atoms
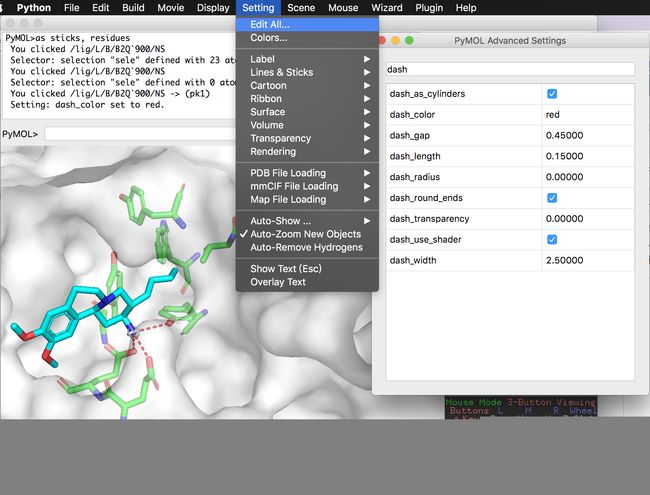
修改氢键样式:在上方菜单栏选择Setting -> Edit All,输入dash,将dash_color修改为red,按回车键确认修改。
最后将视图调整到合适的位置,就可以渲染输出了:
set_view (\
0.596928358, 0.462212354, 0.655772686,\
0.506054461, -0.851173997, 0.139291167,\
0.622557700, 0.248710677, -0.742000937,\
-0.000598469, 0.000388961, -60.677524567,\
54.435737610, 52.480457306, 29.589189529,\
57.677524567, 549.791076660, 20.000000000 )
ray 1200,800
png Figure1.png