scATAC分析神器ArchR初探-简介(1)
scATAC分析神器ArchR初探-ArchR进行doublet处理(2)
scATAC分析神器ArchR初探-创建ArchRProject(3)
scATAC分析神器ArchR初探-使用ArchR降维(4)
scATAC分析神器ArchR初探--使用ArchR进行聚类(5)
scATAC分析神器ArchR初探-单细胞嵌入(6)
scATAC分析神器ArchR初探-使用ArchR计算基因活性值和标记基因(7)
scATAC分析神器ArchR初探-scRNA-seq确定细胞类型(8)
scATAC分析神器ArchR初探-ArchR中的伪批次重复处理(9)
scATAC分析神器ArchR初探-使用ArchR-peak-calling(10)
scATAC分析神器ArchR初探-使用ArchR识别标记峰(11)
scATAC分析神器ArchR初探-使用ArchR进行主题和功能丰富(12)
scATAC分析神器ArchR初探-利用ArchR丰富ChromVAR偏差(13)
scATAC分析神器ArchR初探-使用ArchR进行足迹(14)
scATAC分析神器ArchR初探-使用ArchR进行整合分析(15)
scATAC分析神器ArchR初探-使用ArchR进行轨迹分析(16)
6-单单元嵌入
在ArchR中,嵌入(例如统一流形近似和投影(UMAP)或t分布的随机邻居嵌入(t-SNE))用于可视化缩小维度空间中的单个单元。这些嵌入都有各自的优点和缺点。我们之所以称它们为“嵌入”,是因为它们严格用于可视化集群,而不是用于识别集群(如前几章所述)是在LSI子空间中完成的。UMAP和t-SNE之间的主要区别是细胞或簇之间距离的解释。t-SNE旨在保留数据中的本地结构,而UMAP旨在保留本地和大部分全局数据数据中的结构。从理论上讲,这意味着两个聚类之间的距离在t-SNE中不提供信息,而在UMAP中提供信息。例如,基于观察到群集A在t-SNE上比群集C更靠近群集B,因此t-SNE不允许您说群集A与群集B相比,群集A与群集B更相似。另一方面,UMAP旨在允许进行这种类型的比较,尽管值得注意的是,UMAP是一种足够新的方法,至今仍在文献中被淘汰。
重要的是要注意,t-SNE和UMAP都不是自然确定性的(相同的输入总是提供完全相同的输出)。但是,与UMAP相比,t-SNE在同一输入的多个重复之间显示出更多的随机性。此外,uwot当使用相同的random时,在包中实现的UMAP 是有效的确定性seed。选择使用UMAP还是使用t-SNE的选择很细微,但是在我们看来,UMAP对于各种应用程序都非常有效,这是我们针对scATAC-seq数据的标准选择。UMAP的性能也比t-SNE快。也许最重要的是,使用UMAP可以创建嵌入并将新样本投影到该嵌入中,而t-SNE则无法实现,因为数据的拟合和预测同时发生。
无论选择哪种方法,输入参数都可能对生成的嵌入产生巨大影响。因此,重要的是要了解各种输入参数,并进行调整以最好地满足您自己数据的需求。ArchR实现了适用于大多数应用程序的默认输入参数集,但实际上,没有单个参数集可以为单元格数量,复杂性和质量差异很大的数据集产生所需的结果。
6.1均匀流形逼近和投影(UMAP)
要在ArchR中运行UMAP,请使用以下addUMAP()功能:
projHeme2 <- addUMAP(
ArchRProj = projHeme2,
reducedDims = "IterativeLSI",
name = "UMAP",
nNeighbors = 30,
minDist = 0.5,
metric = "cosine"
)
您可以使用插槽提取运算符embeddings在中列出可用的对象:ArchRProject``@
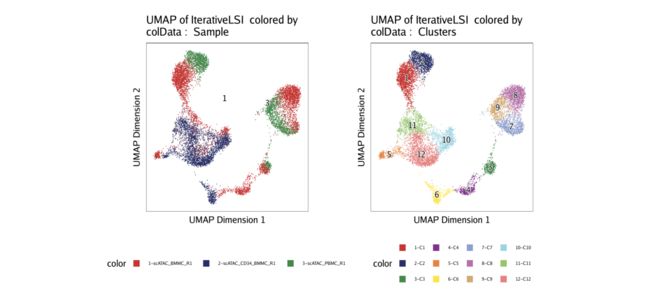
为了绘制UMAP结果,我们使用plotEmbedding()函数并传递刚刚生成的UMAP嵌入的名称(“ UMAP”)。我们可以通过结合使用colorBy哪种组合来告诉ArchR如何使用哪种矩阵来查找提供给的指定元数据列,从而告诉ArchR如何为单元着色name。
p1 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "UMAP")
代替上面的“样本”进行着色,我们可以使用上一章中确定的“簇”进行着色。
p2 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Clusters", embedding = "UMAP")
我们可以使用ggAlignPlots()函数并排显示这两个图,并使用指定水平方向type = "h"。
ggAlignPlots(p1, p2, type = "h")
plotPDF(p1,p2, name = "Plot-UMAP-Sample-Clusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)
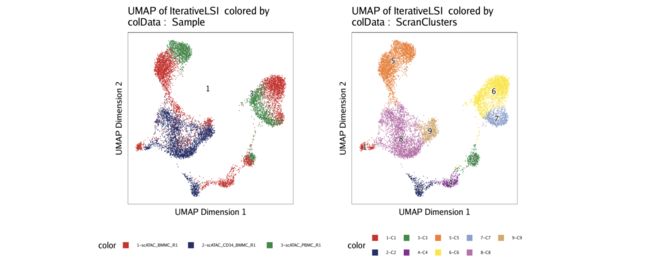
我们还可以使用plotEmbedding()来可视化聚类的结果scran:
p1 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "UMAP")
p2 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "ScranClusters", embedding = "UMAP")
ggAlignPlots(p1, p2, type = "h")
要保存此图的可编辑矢量化版本,请使用plotPDF()。
plotPDF(p1,p2, name = "Plot-UMAP-Sample-ScranClusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)
6.2 t-Stocastic邻居嵌入(t-SNE)
要在ArchR中运行t-SNE,请使用以下addTSNE()功能:
projHeme2 <- addTSNE(
ArchRProj = projHeme2,
reducedDims = "IterativeLSI",
name = "TSNE",
perplexity = 30
)
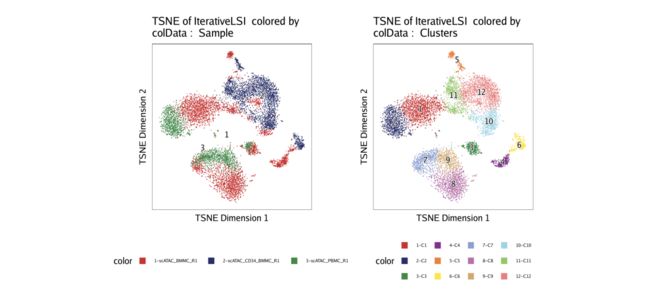
与UMAP类似,我们可以使用绘制t-SNE嵌入plotEmbedding()。相同的参数适用于colorBy和name正在被使用的包埋的类型而不管哪个。
p1 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "TSNE")
p2 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Clusters", embedding = "TSNE")
ggAlignPlots(p1, p2, type = "h")
要保存此图的可编辑矢量化版本,请使用plotPDF()。
plotPDF(p1,p2, name = "Plot-TSNE-Sample-Clusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)
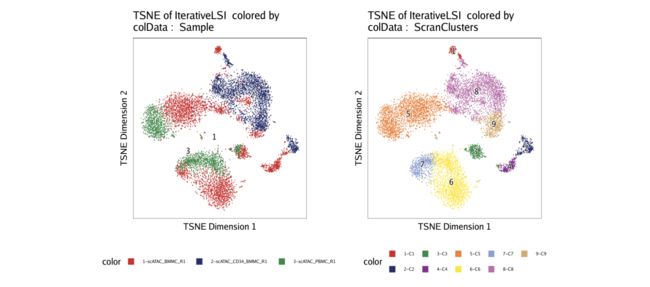
就像我们使用UMAP一样,我们可以将的聚类结果Seurat::FindClusters()与clusering的结果进行比较scran:
p1 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "TSNE")
p2 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "ScranClusters", embedding = "TSNE")
ggAlignPlots(p1, p2, type = "h")
要保存此图的可编辑矢量化版本,请使用plotPDF()。
plotPDF(p1,p2, name = "Plot-tSNE-Sample-ScranClusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)
6.3调和后的降维
在上一章中,我们通过addHarmony()函数使用Harmony进行了批量校正,创建了一个reducedDims名为“ Harmony” 的对象。我们可以通过使用UMAP或t-SNE可视化嵌入并将其与之前的LSI迭代可视化嵌入进行比较,从而评估Harmony的效果。
重复使用相同参数但针对“ Harmony” reducedDims对象的UMAP嵌入:
projHeme2 <- addUMAP(
ArchRProj = projHeme2,
reducedDims = "Harmony",
name = "UMAPHarmony",
nNeighbors = 30,
minDist = 0.5,
metric = "cosine"
)
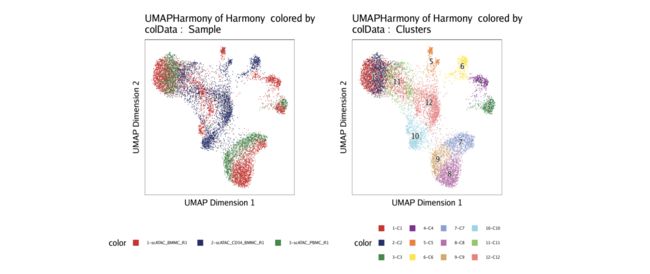
p3 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "UMAPHarmony")
p4 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Clusters", embedding = "UMAPHarmony")
ggAlignPlots(p3, p4, type = "h")
要保存此图的可编辑矢量化版本,请使用plotPDF()。
plotPDF(p1,p2,p3,p4, name = "Plot-UMAP2Harmony-Sample-Clusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)
对于t-SNE同样如此:
projHeme2 <- addTSNE(
ArchRProj = projHeme2,
reducedDims = "Harmony",
name = "TSNEHarmony",
perplexity = 30
)
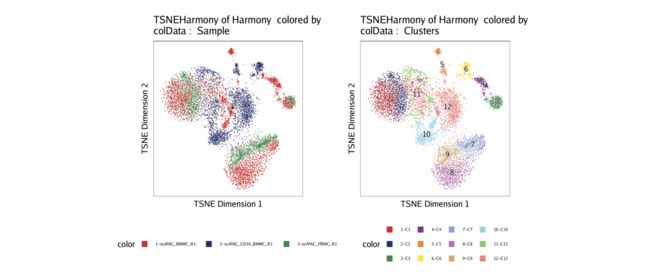
p3 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Sample", embedding = "TSNEHarmony")
p4 <- plotEmbedding(ArchRProj = projHeme2, colorBy = "cellColData", name = "Clusters", embedding = "TSNEHarmony")
ggAlignPlots(p3, p4, type = "h")
要保存此图的可编辑矢量化版本,请使用plotPDF()。
plotPDF(p1,p2,p3,p4, name = "Plot-TSNE2Harmony-Sample-Clusters.pdf", ArchRProj = projHeme2, addDOC = FALSE, width = 5, height = 5)
参考材料:
https://www.archrproject.com/