有监督学习指为了实现根据一样本的预测变量来预测相对应的二分类结果(class),而基于一组大量、已有的包含预测变量值和输出变量值(即二分类结果)的样本单元数据进行建模、验证。
思路为将所有样本单元数据分为一个训练集(较多)和一个验证集(较少)。先用训练集建立预测模型,再用验证集验证模型的准确性。得到一个有效的预测模型后,就可去真实场景根据预测变量预测二分类结果(class)了。具体方法有逻辑回归、决策树、随机森林、支持向量机等。一起来学习R语言的有关操作吧~
实验示例及数据预处理
实验数据来自UCI机器学习数据库,为699个乳腺癌样本观测的11个医学指标(第11列为class列)。其中458个观测为良性样本单元;241个观测为恶性样本单元。(有16个样本单元含有缺失值数据,用?表示)
1、读取数据到R
loc <- "http://archive.ics.uci.edu/ml/machine-learning-databases/"
ds <- "breast-cancer-wisconsin/breast-cancer-wisconsin.data"
url <- paste(loc, ds, sep="")
#上述组成完整的数据获取网址
breast <- read.table(url, sep=",", header=FALSE, na.strings="?")
#利用read.table()读取
2、数据表修饰
breast
查看原始数据,有一些需要修饰的地方。并且数据有如下特征:
- 预测变量数据经过处理,转化为特征得分;从1(最接近良性)到10(最接近病变)
- 最后一列类别变量为输出变量:2为良性;4为恶性。
names(breast) <- c("ID", "clumpThickness", "sizeUniformity",
"shapeUniformity", "maginalAdhesion",
"singleEpithelialCellSize", "bareNuclei",
"blandChromatin", "normalNucleoli", "mitosis", "class")
#添加列/变量名
df <- breast[-1]
#删除第一列Id值
df$class <- factor(df$class, levels=c(2,4),
labels=c("benign", "malignant"))
#将最后一列转化为因子,并贴上标签
3、抽取训练集与验证集
train <- sample(nrow(df), 0.7*nrow(df))
#3/7分
df.train <- df[train,]
#抽取70%为训练集(489)
df.validate <- df[-train,]
#剩余的为验证集(210)
table(df.train$class)
table(df.validate$class)
- 训练集:良性样本323个;恶性样本166个。
- 验证集:良性样本135个;恶性样本75个。
法一:逻辑回归(logistic regression)
即为原先学的广义线性模型的一种(详见第十二节),使用基础函数glm()
- 1、拟合
fit.logit <- glm(class~., data=df.train, family=binomial())
# 注意表达式里的点 . 表示表格里除了因变量(class)里的其它所有预测变量,方便的一种写法。
summary(fit.logit)
检查模型,观察下预测变量系数是否有未通过显著性检验(p值大于1)的,后期可以考虑优化一下。
- 2、验证
prob <- predict(fit.logit, df.validate, type="response")
#type="response"得到预测肿瘤为恶性的概率
logit.pred <- factor(prob > .5, levels=c(FALSE, TRUE),
labels=c("benign", "malignant"))
#概率大于0.5为TRUE,被贴上恶性的标签
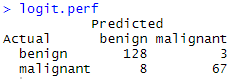
- 3、交叉表结果
logit.perf <- table(df.validate$class, logit.pred,
dnn=c("Actual", "Predicted"))
logit.perf
最终得到预测与实际情况对比的交叉表(混淆矩阵,confusion matrix),基于此可判断拟合建模质量如何
如果想去除显著性低的预测变量,从而减小误差,可使用代码
logit.fit.reduced <- step(fit.logic),然后再进行验证。(我试了下,没有太大作用好像,参看第七节的学习)
法二:决策树(classical decision tree)
经典决策树
1、算法--基于纯度
(1)选定一个最佳预测变量,判定阈值标准,将全部样本单元分割为两类,使得两类的纯度最大化(一类里良的尽量多,另一类里恶的尽量多)
(2)对每一个子类别继续执行步骤(1)
(3)重复步骤(1)与(2)、直到集中的子类别所含的样本单元过少、或者没有分类法能将不纯度降低到一定标准。最终集中的子类别就是终端节点。根据每一个终端节点中样本单元的类别数众数来判断这一终端节点的所属类别。
(4)对于其它样本,从头执行决策树,对应到某一终端节点,从而进行类别判断。
- 综上通常会得到一颗过大的树,出现过拟合,普适性弱,需要采用10折交叉验证法选择误差较小的树。
2、具体步骤
(1)生成初始树
library(rpart)
dtree <- rpart(class ~ ., data=df.train, method="class",
parms=list(split="information"))
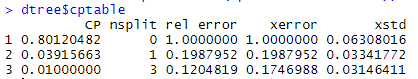
(2)剪枝
dtree$cptable
返回的信息有--
- CP为复杂度参数,为后面剪枝的重要依据;
- nsplit为分支数,终端节点比分支数多一个;
- rel error 训练集中各种树对应的误差;
- xerror 为交叉验证误差;
- xstd 为交叉验证误差的标准差。
选择最优的树的原则基于两个条件:一是其交叉验证误差在返回的最小的交叉验证误差(0.17)一个标准差范围内(0.03)内;二是终端节点最少。
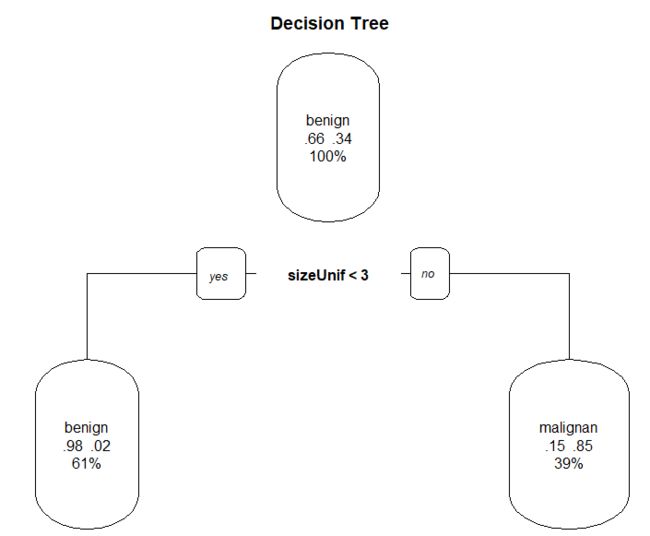
基于上图结果,即选取原则, 这次做的结果就直接推荐仅两个分支,基于一个变量标准的小小树,还是很诧异的,看看验证结果如何吧
dtree.pruned <- prune(dtree, cp=.0392)
library(rpart.plot)
prp(dtree.pruned, type = 2, extra = 104,
fallen.leaves = TRUE, main="Decision Tree")
#绘制决策树
关于prp()函数绘制决策图的参数说明
- type = 2 画出每个标签下分隔的标签(变量)
- extra = 104 画出每一类的总概率(样本数/总样本数)及该节点出良/恶的比例;
- fallen.leaves = TRUE 在图的底端显示出终端节点
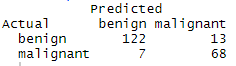
(3)验证与交叉表
dtree.pred <- predict(dtree.pruned, df.validate, type="class")
dtree.perf <- table(df.validate$class, dtree.pred,
dnn=c("Actual", "Predicted"))
dtree.perf
竟然只有一个变量标准准确率还有90%以上。要么是这个变量很重要,要么是数据太少了~ 总之我觉得就是怪怪的。我之前做的是建议分成5个终端节点,书上的结果是建议4个,看来抽样对于建树影响挺大的。
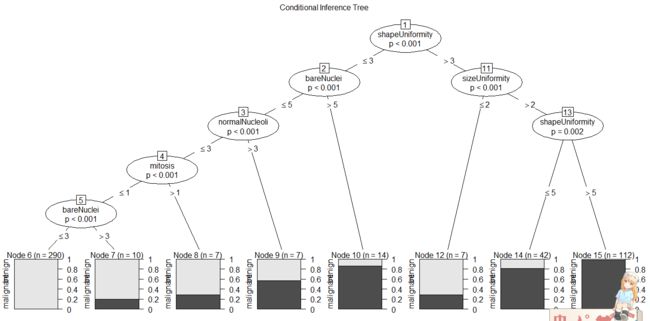
条件推断树(conditional inference tree)
1、算法--基于显著性检验
(1)对输出变量与每个预测变量间的关系计算p值;
(2)选取p值最小的变量;(p值小,不表明最相关,而是表明最有可能正确)
(3)在因变量(应该指class值)与被选中的变量间尝试所有可能的二元分割(通过排列检验),并选取最显著的分割;
(4)将数据集分成两群,并对每个子群重复上述步骤;
(5)重复至所有分割都不显著或已达到最小节点为止。
2、具体步骤
(1)生成树并绘制--不必剪枝了
library(party)
fit.ctree <- ctree(class~., data=df.train)
plot(fit.ctree, main="Conditional Inference Tree")
#绘制条件推断树,阴影区域代表这个节点的恶性肿瘤比例。
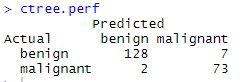
(2)验证与交叉表
ctree.pred <- predict(fit.ctree, df.validate, type="response")
ctree.perf <- table(df.validate$class, ctree.pred,
dnn=c("Actual", "Predicted"))
ctree.perf
- 在本例中条件推断树的验证结果比经典决策树好一些。
法三:随机森林(Random forest)
- 特点:组成式的有监督学习方法----同时生成多个预测模型,并将模型的结果汇总以提升分类准确率。
- 思路:对样本单元和变量进行抽样,从而生成大量决策树。对于每个样本来说,所有决策树依次对其进行分类。所有决策树预测类别中的众数类别即为随机森林所预测的这一样本单元的类别。(详见p368)
- randomForest包中的randomForest()函数用于生成随机变量,默认生成500颗树(经典决策树),并且默认在每个节点处抽取sqrt(M)个变量,最小节点为1。
party()包的cforest()函数可基于条件推断树生成随机森林。
- 具体步骤如下:
(1)生成随机森林
library(randomForest)
fit.forest <- randomForest(class~., data=df.train,
na.action=na.roughfix,
importance=TRUE)
- 默认在每棵树的每个节点随机抽取三个节点,生成500颗树;
- na.action=na.roughfix 将变量中的缺失值替换成对应列的中位数。(若是类别型变量,则替换成众数);
- importance=TRUE 设置评价变量重要性
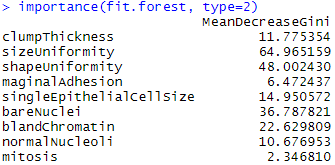
(2)查看变量重要性
importance(fit.forest, type=2)
#type=2 参数设置变量相对重要性的原理,详见p370
由下图可看出,sizeUniformity为最重要的预测变量;mitosis为最不重要的变量。
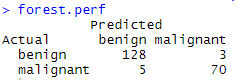
(3)验证与交叉表
forest.pred <- predict(fit.forest, df.validate)
forest.perf <- table(df.validate$class, forest.pred,
dnn=c("Actual", "Predicted"))
forest.perf
- 结果看起来还不错
法四:支持向量机(support vector machine,SVM)
- 一类可用于分类和回归的有监督机器学习模型,这里只学习其在二元分类问题中的应用。
简单来说,SVM旨在多为空间中找到一个能将全部样本单元分成两类的最优平面,这一平面应使两类中距离最近的点的间距(margin)尽可能的大,在间距边上的点被称为支持向量(support vector,它们决定间距),分割的超平面位于间距的中间。对于一个N维空间(N个变量)来说,最优超平面(即线性决策面,linear decision surface)为N-1维。详细原理见p370
1、 默认参数法
library(e1071)
fit.svm <- svm(class~., data=df.train)
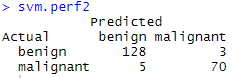
svm.pred <- predict(fit.svm, na.omit(df.validate))
svm.perf <- table(na.omit(df.validate)$class,
svm.pred, dnn=c("Actual", "Predicted"))
svm.perf
结果也还行
2、选择调和参数(原理详见p372)
- 上述用法中gamma默认为预测变量个数的倒数、成本参数默认为1。
- 在建模时,我们也可以尝试变动参数值建立不同的模型,从中选择最佳的组合
tuned <- tune.svm(class~., data=df.train,
gamma=10^(-6:1),
cost=10^(-10:10))
tuned
gamma的8种变化与cost的21种变化,共168个模型,从中选最佳。发现gamma为0.1,cost为1时最优
fit.svm2 <- svm(class~., data=df.train, gamma=.01, cost=1)
svm.pred 2<- predict(fit.svm, na.omit(df.validate))
svm.perf 2<- table(na.omit(df.validate)$class,
svm.pred, dnn=c("Actual", "Predicted"))
svm.perf2
并未改善,但是一般来说为SVM模型选择调和参数可以得到更好的结果,比如我之前尝试的一次。
评价分类模型
1、评价指标
- 敏感度Sensitivity:所有患者里,有多少被成功预测(实际有病样本为基数);
- 特异性Specificity:所有健康人里,有多少被成功预测(实际没病样本为基数);
- 正例命中率Positive Predictive Value:被预测为有病的人里,有多少是预测正确的(预测有病样本为基数);
- 负例命中率Positive Predictive Value:被预测为没病的人里,有多少是预测正确的(预测没病样本为基数);
- 准确率Accuracy:被正确分类的样本单元所占比重,也叫ACC;
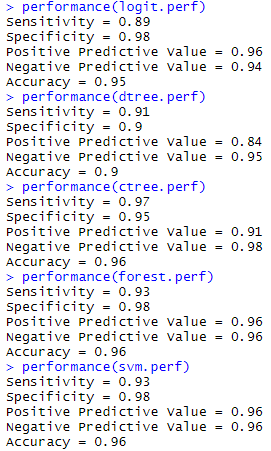
对于癌症等疾病诊断角度来说,敏感度指标格外重要。此外敏感度与特异度是相互权衡,此消彼长的关系,可以通过设定阈值加以调整。P376
2、生成计算上述指标的函数
performance <- function(table, n=2){
if(!all(dim(table) == c(2,2)))
stop("Must be a 2 x 2 table") #一定要是2×2形式的二联表
tn = table[1,1]
fp = table[1,2]
fn = table[2,1]
tp = table[2,2]
#分别对应位置取值
sensitivity = tp/(tp+fn)
#计算敏感度
specificity = tn/(tn+fp)
#计算特异度
ppp = tp/(tp+fp)
#计算正例命中率
npp = tn/(tn+fn)
#计算负例命中率
hitrate = (tp+tn)/(tp+tn+fp+fn)
#计算准确率
result <- paste("Sensitivity = ", round(sensitivity, n) , #原来n是设置了保留的位数
"\nSpecificity = ", round(specificity, n), # /n 为换行符
"\nPositive Predictive Value = ", round(ppp, n),
"\nNegative Predictive Value = ", round(npp, n),
"\nAccuracy = ", round(hitrate, n), "\n", sep="")
cat(result)
}
3、评价获得的模型结果
performance(logit.perf)
performance(dtree.perf)
performance(ctree.perf)
performance(forest.perf)
performance(svm.perf)
此外,教材中还介绍了利用rattel()包提供图像式交互界面来做上述以及更多的数据分析。由于相关软件安装失败,未能实际演练,以后有机会的吧。详见p376
数据挖掘一般先尝试相对简单的方法(逻辑回归、决策树)和一些复杂的方法(随机森林、支持向量机)。如果与简单的方法相比,复杂方法在预测效果方面没有显著提升,则倾向于选择简单的方法。
参考教材《R语言实战(第二版)》