上一节我们对原始数据进行了质量检测,对于质量比较好的数据我们可以直接进行后续分析,而对于一般质量的,或者质量较差的,我们就需要对数据进行进一步的清洗过滤了。
前文我们提到,下机数据中可能包含以下几类不合格数据:
- 测序质量低 :低质量碱基(即质量分数为小于10的碱基)占整条序列的30%以上。
- 序列含接头 :接头序列未去除干净或者由于测序读长较短测穿了的数据。
- 含N比例较高:由于机器设备或者其他原因,导致碱基未测出的。
- 测序错误的序列等
这块比较好的几款软件都在 Github (程序员搞基大本营) 上建立了各自的开源项目。
1. 几款常用软件 (排序不分先后)
1.1 fastp
业内同行海普洛斯小伙伴们的一个开源项目,这里要向海普洛斯 CTO 陈实富博士(推广到了我们生信人员大本营)致敬。
该程序由 C++开发,支持多线程,能实现多个功能,操作简便,支持较多,开发也较为活跃,个人推荐使用:
- 可以过滤低质量数据 (如较低的质量分数,较短的序列,含 N 较多的序列等等)
- 可以实现剪切序列的首尾两端
- 可以通过对滑动窗口的平均质量的评价,对 5' 和 3' 端序列进行剪切(这个功能与Trimmomatic 软件功能相似,但是速度却更快)
- 可以自动检测街头序列并做切除
- 对于PE数据中的overlap区间中不一致的碱基对,依据质量值进行校正
- 支持三代测序或四代测序的long reads (nanopore / pacbio的数据)
- 生成 html/json 报告,有助于直观的查看,json 格式更利于一些大型流程的串写
- 其他等一系列功能(软件还在开发阶段,可以直接和作者团队交流,提一些相关建议)
题外话:软件安装不外乎两种方式(主要针对Linux,其他系统大都相似):
- 二进制程序直接下载使用 (建议初学者使用)
- 下载源代码进行编译
## 软件安装:
## fastp 有编译好的二进制可执行文件版本,我们直接下载使用
wget http://opengene.org/fastp/fastp
chmod a+x ./fastp
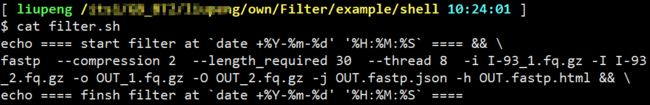
## 使用起来也比较简单
fastp -i in.fq -o out.fq
参数不少,但是说明给的还比较详细。具体可以查看 网页。
会输出过滤后的结果,fq/fq.gz 格式,同时还有 json 和 html 格式的报告,具体内容见 示例报告
1.2 AfterQC
fastp 前身,python语言写的。输出内容比较丰富,但是速度较慢,开发团队推荐 使用 fastp 。
## 软件安装(由于是 python包,推荐使用 bioconda安装)
conda install afterqc
## 也可以直接下载
wget -c https://github.com/OpenGene/AfterQC/archive/master.zip
## 解压后使用
unzip master.zip
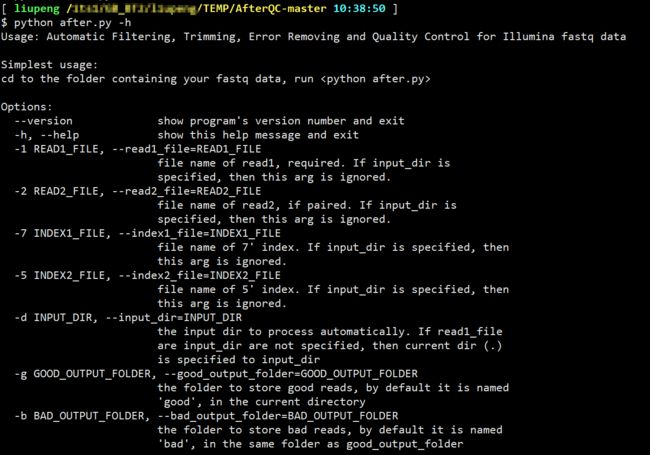
## 用法比较简单
# SE
python after.py -1 R1.fq
# PE
python after.py -1 R1.fq -2 R2.fq
生成的报告比较丰富:
Example: http://opengene.org/AfterQC/report.html
1.3 SOAPnuke
华大看家工具集 SOAP 系列之一。适用于多种数据类型 ( mRNA,small RNA,DEG... )
## 软件下载安装
git clone https://github.com/BGI-flexlab/SOAPnuke.git
cd SOAPnuke/src
cmake .
make
./SOAPnuke
## 软件使用
SOAPnuke filter --cut 0 --qualRate 0.5 --index --lowQual 5 --nRate 0.1 -Q 2 -5 1 -1 ZJ-2-1-A_1.fq.gz -2 ZJ-2-1-A_2.fq.gz -o output/ZJ1Rep1 -C ZJ1Rep1_1.fq -D ZJ1Rep1_2.fq
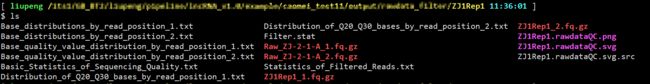
结果展示如下:
这些文件详细的展示了序列过滤的具体情况。
1.4 seqtk
李恒大神(不知道此人的请自行百度)写的一款过滤软件。
四个字点评:短小精悍!
## 下载安装
git clone https://github.com/lh3/seqtk.git;
cd seqtk;
make
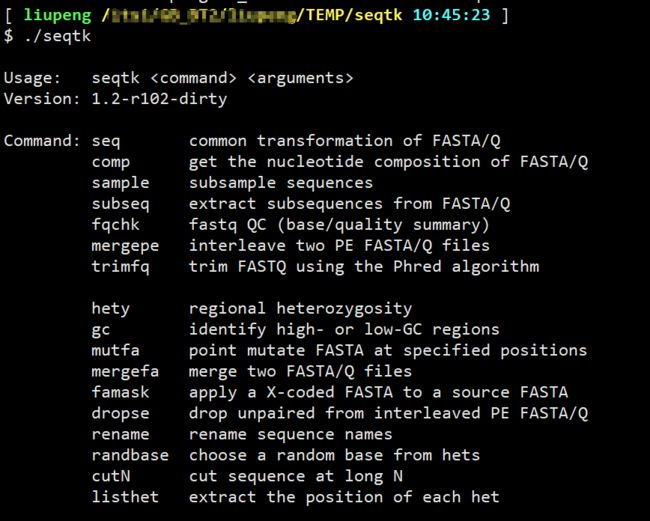
该软件功能丰富,具体示例详见说明文档:https://github.com/lh3/seqtk
我们重点关注 fqchk 与trimfq 功能:
1.5 Trimmomatic
老牌软件啦,java 写的,说明文档:manual。
## 下载安装
wget -c http://www.usadellab.org/cms/uploads/supplementary/Trimmomatic/Trimmomatic-0.36.zip
## 解压软件包
unzip Trimmomatic-0.36.zip
## 软件使用示例
# PE
java -jar trimmomatic-0.35.jar PE -phred33 input_forward.fq.gz input_reverse.fq.gz output_forward_paired.fq.gz output_forward_unpaired.fq.gz output_reverse_paired.fq.gz output_reverse_unpaired.fq.gz ILLUMINACLIP:TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
# SE
java -jar trimmomatic-0.35.jar SE -phred33 input.fq.gz output.fq.gz ILLUMINACLIP:TruSeq3-SE:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
参数说明:
PE/SE 设定对Paired-End或Single-End的reads进行处理,其输入和输出参数稍有不一样。
-threads 设置多线程运行数
-phred33 设置碱基的质量格式,可选pred64
ILLUMINACLIP:TruSeq3-PE.fa:2:30:10 切除adapter序列。参数后面分别接adapter序列的fasta文件:允许的最大 mismatch 数:palindrome 模式下匹配碱基数阈值:simple模式下的匹配碱基数阈值。
LEADING:3 切除首端碱基质量小于3的碱基
TRAILING:3 切除尾端碱基质量小于3的碱基
SLIDINGWINDOW:4:15 从5'端开始进行滑动,当滑动位点周围一段序列(window)的平均碱基低于阈值,则从该处进行切除。Windows的size是4个碱基,其平均碱基
质量小于15,则切除。
MINLEN:50 最小的reads长度
CROP: 保留reads到指定的长度
HEADCROP: 在reads的首端切除指定的长度
TOPHRED33 将碱基质量转换为pred33格式
TOPHRED64 将碱基质量转换为pred64格式
2. 其他数据过滤软件:
QcReads
由中科院昆明动物所开发的一个快速、高效的高通量测序reads去接头和去低质量序列工具。
cutadapt
从名字可以很直观的看出来这是一款去接头的软件啦~
fastx_toolkit
二代测序经典数据质控过滤软件,只是 2010 年后就没有维护更新啦~
喜欢折腾的可以去官网或者 github 上逛逛:
官网
GitHub
SOAPnuke 软件编译这块稍显复杂,之前华大网站有放出编译好的 SOAPnuke 程序,可惜找不到了。文中所列软件,如果有需要的话,各位下伙伴可以后台回复索取(Linux 版本)。
欢迎补充讨论。
下节我们详细讨论各个软件生成的报告。
3. 小尾巴.命令详解
今天的小尾巴是 chmod.
chmod命令用来变更文件或目录的权限。在UNIX系统家族里,文件或目录权限的控制分别以读取、写入、执行3种一般权限来区分,另有3种特殊权限可供运用。用户可以使用chmod指令去变更文件与目录的权限,设置方式采用文字或数字代号皆可。符号连接的权限无法变更,如果用户对符号连接修改权限,其改变会作用在被连接的原始文件。
语法:
chmod [-cfvR] [--help] [--version] mode file...
权限范围的表示法如下:
u User,即文件或目录的拥有者;
g Group,即文件或目录的所属群组;
o Other,除了文件或目录拥有者或所属群组之外,其他用户皆属于这个范围;
a All,即全部的用户,包含拥有者,所属群组以及其他用户;
r 读取权限,数字代号为“4”;
w 写入权限,数字代号为“2”;
x 执行或切换权限,数字代号为“1”;
- 不具任何权限,数字代号为“0”;
s 特殊功能说明:变更文件或目录的权限。
来自: http://man.linuxde.net/chmod
参考资料
http://journal.embnet.org/index.php/embnetjournal/article/view/200/479
http://hannonlab.cshl.edu/fastx_toolkit/index.html
http://not.farbox.com/page/9
https://zhuanlan.zhihu.com/p/28924793
https://disease.novogene.org/topic/305/%E4%BD%BF%E7%94%A8trimmomatic%E8%BF%87%E6%BB%A4%E4%BD%8E%E8%B4%A8%E9%87%8F%E5%BA%8F%E5%88%97?page=1
http://not.farbox.com/post/fastx_toolkit