scATAC分析神器ArchR初探-简介(1)
scATAC分析神器ArchR初探-ArchR进行doublet处理(2)
scATAC分析神器ArchR初探-创建ArchRProject(3)
scATAC分析神器ArchR初探-使用ArchR降维(4)
scATAC分析神器ArchR初探--使用ArchR进行聚类(5)
scATAC分析神器ArchR初探-单细胞嵌入(6)
scATAC分析神器ArchR初探-使用ArchR计算基因活性值和标记基因(7)
scATAC分析神器ArchR初探-scRNA-seq确定细胞类型(8)
scATAC分析神器ArchR初探-ArchR中的伪批次重复处理(9)
scATAC分析神器ArchR初探-使用ArchR-peak-calling(10)
scATAC分析神器ArchR初探-使用ArchR识别标记峰(11)
scATAC分析神器ArchR初探-使用ArchR进行主题和功能丰富(12)
scATAC分析神器ArchR初探-利用ArchR丰富ChromVAR偏差(13)
scATAC分析神器ArchR初探-使用ArchR进行足迹(14)
scATAC分析神器ArchR初探-使用ArchR进行整合分析(15)
scATAC分析神器ArchR初探-使用ArchR进行轨迹分析(16)
9- ArchR中的伪批次重复处理
因为scATAC-seq数据本质上是二进制的-意味着任何单个位点都是可访问的或不可访问的-我们经常发现自己想要执行在单个单元格上不可能进行的分析。此外,我们要执行的许多分析都需要重复进行,以获得具有统计意义的度量。在单单元数据中,我们通过创建伪批量复制来解决这些问题。术语“ 伪批量”是指单个单元格的分组,其中来自每个单个单元格的数据被组合成一个单个的伪样本,该样本类似于批量ATAC-seq实验。对于每个所需的细胞分组,ArchR都会制作多个此类伪大量样本,因此术语“ 伪大量”是重复的。在此过程中的基本假设是,被分组在一起的单个单元格足够相似,因此我们无需理会它们之间的差异。这些单元分组几乎总是从对应于已知单元类型的单个群集或群集的超集派生的。本章介绍了ArchR生成这些伪批次重复的过程。
9.1 ArchR如何进行伪批量复制?
为了创建伪批量复制,ArchR采用了分层优先级方法。用户指定(i)所需的最小和最大重复数,(ii)每个重复的最小和最大细胞数,以及(iii)如果特定组缺乏足够的细胞来进行所需的重复,则使用的采样率。例如,采样率为0.8意味着可以对每个单元进行采样而无需替换,最多可替换每个重复的单元总数的80%(这将导致在重复之间进行替换采样)。在这种情况下,多个重复可能包含一些相同的单元格,但是如果您要从缺少足够单元格的单元组中生成伪批次重复,这是必要的牺牲。
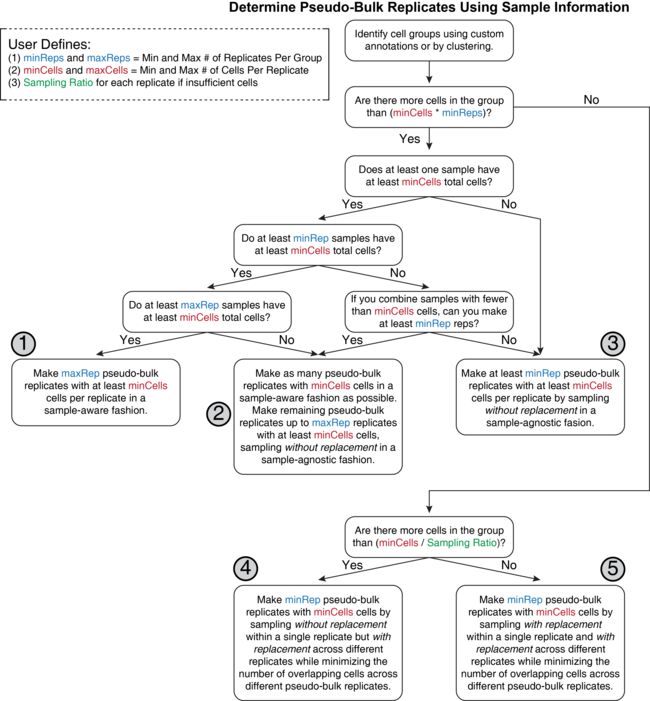
伪批次重复生成的过程可以通过如下所示的决策树来描述。
我们在这里用文字概述了此过程的一些关键考虑因素。首先,用户确定要使用的单元组-这通常是ArchR调用的集群。然后,对于每个单元分组,ArchR尝试创建所需的伪批量重复。理想的伪大量复制将由 单个样本中足够数量的细胞 组成。这样可以保持重复样品之间的样品多样性和生物学差异。这是ArchR力求获得的成果,但实际上在此过程中有5种可能的结果,在ArchR中按优先级排列如下:
- 足够多的不同样本(至少最大数量的重复样本)每个都具有比最小数目更多的细胞,可以以样本感知的方式创建伪批量复制样本,仅将来自同一样本的细胞合并为一个复制样本。
- 一些样本每个都有超过最小数量的单元,以样本感知方式创建伪大量复制。其余所需的重复项是通过合并单元而不用从样本感知伪批量中尚未表示的样本进行替换来创建的。
- 创建样本感知的伪批量复制时,没有任何样本具有超过最小数量的单元格,但有个以上的单元格
minCells * minReps。所有需要的重复通过结合细胞产生无需更换从在样品无关的方式。 - 单元分组中的单元总数少于最小单元数乘以最小重复数,但大于最小单元数除以采样率。通过采样来创建最小数量的复制品,而无需在单个复制品中进行替换,而可以在多个复制品中进行替换,同时最大程度地减少多个伪批量复制品中存在的细胞数量。
- 单元格分组内的单元格总数小于最小单元数除以采样率。这意味着我们必须通过在单个副本内以及不同副本之间进行替换采样来制作副本。这是最坏的情况,用户应谨慎使用下游的这些伪批量复制。可以使用该
minCells参数在其他各种ArchR函数中进行控制。
为了说明此过程,我们将使用以下示例数据集:
Sample Cluster1 Cluster2 Cluster3 Cluster4 Cluster5
A 800 600 900 100 75
B 1000 50 400 150 25
C 600 900 100 200 50
D 1200 500 50 50 25
E 900 100 50 150 50
F 700 200 100 100 25
我们将设置minRep = 3,maxRep = 5,minCells = 300,maxCells = 1000,和sampleRatio = 0.8。
9.1.1群集1
对于Cluster1,我们有6个样本(大于maxRep),所有样本均具有多个minCells单元(300个单元)。这说明了上面的选项#1,我们将以示例感知的方式进行5次伪批量复制,如下所示:
Rep1 = 800 cells from SampleA
Rep2 = 1000 cells from SampleB
Rep3 = 1000 cells from SampleD
Rep4 = 900 cells from SampleE
Rep5 = 700 cells from SampleF
关于这些复制,有两点需要注意:(i)SampleC被省略了,因为我们有足够多的样本来进行maxRep可感知样本的伪批量复制,而SampleC的单元数最少。(ii)SampleD只使用了1000个单元格,因为这是maxCells值。
9.1.2集群2
对于Cluster2,我们有3个样本均具有多个minCells单元格,而另一些样本则没有。这说明了上面的选项2,我们将进行以下伪批量复制:
Rep1 = 600 cells from SampleA
Rep2 = 900 cells from SampleC
Rep3 = 500 cells from SampleD
Rep4 = 350 cells [50 cells from SampleB + 100 from SampleE + 200 from SampleF]
在此示例中,Rep4是通过无须抽样的方式以不可知的方式创建的。
9.1.3群集3
对于Cluster3,我们只有2个样本的minCells单元格数量多于所需数量minReps。但是,如果我们合并其余样本中的单元格,则可以使用多个进行另外的复制minCells。这总共给我们提供了3个伪批量复制,并代表了上述选项3所示的情况。我们将进行以下复制:
Rep1 = 900 cells from SampleA
Rep2 = 400 cells from SampleB
Rep3 = 250 cells [100 cells from SampleC + 50 from SampleD + 50 from SampleE + 50 from SampleF]
与上面的Cluster2相似,Cluster3 Rep3是通过样本不可知的方式创建的,无需采样即可在多个样本之间进行替换。
9.1.4集群4
对于Cluster4,单元总数为750,少于minCells * minReps(900个单元)。在这种情况下,minReps至少minCells没有某种形式的替换样本,我们没有足够的单元来制作。但是,总细胞数仍大于minCells / sampleRatio(375个细胞),这意味着我们只需要在不同的伪大量重复样本中进行替换取样,而不必在单个重复样本中进行替换。这代表了上面选项4中所示的情况,因此我们将进行以下复制:
Rep1 = 300 cells [250 unique cells + 25 cells overlapping Rep2 + 25 cells overlapping Rep3]
Rep2 = 300 cells [250 unique cells + 25 cells overlapping Rep1 + 25 cells overlapping Rep3]
Rep3 = 300 cells [250 unique cells + 25 cells overlapping Rep1 + 25 cells overlapping Rep2]
在这种情况下,ArchR将最大程度减少任何两个伪批量复制之间重叠的单元数量。
9.1.5集群5
对于Cluster5,单元总数为250,小于minCells * minReps(900个单元)且小于minCells / sampleRatio(375个单元)。这意味着我们将不得不在每个样本内以及跨不同的复制品进行替换采样,以进行伪批量复制。这代表了以上选项5中所示的最不理想的情况,因此在下游分析中使用这些伪大量复制品时应谨慎。因此,我们将进行以下复制:
Rep1 = 300 cells [250 unique cells + 25 cells overlapping Rep2 + 25 cells overlapping Rep3]
Rep2 = 300 cells [250 unique cells + 25 cells overlapping Rep1 + 25 cells overlapping Rep3]
Rep3 = 300 cells [250 unique cells + 25 cells overlapping Rep1 + 25 cells overlapping Rep2]
9.2进行伪批量重复
在ArchR中,使用addGroupCoverages()函数进行伪批量重复。此处的关键参数groupBy定义了应进行伪批量重复的组。在这里,我们使用的Clusters2是通过用上一章的scRNA-seq数据定义的细胞类型标记簇来定义的。
projHeme4 <- addGroupCoverages(ArchRProj = projHeme3, groupBy = "Clusters2")
生成这些伪批量重复后,我们现在可以在数据中调用峰。如前所述,我们不想在所有单个像元的合并集合上调用峰,因此通过聚类或其他方式定义这些更细粒度的像元组,为调用峰提供了理想的起点。
参考材料:
https://www.archrproject.com/