典型的聚类算法--学习笔记
目录
- 一. 前言
- 二. 典型的聚类算法
-
- 1. 聚类算法的选择
- 2. K-Means(K均值)
-
- (1)原理
- (2)示意图
- (3)算法优缺点
- (4)注意事项
- (5)示例代码
- (6)算法拓展--Mini Batch K-Means
- (7)模型评估
- 3. Mean-shift(均值偏移)
-
- (1)原理
- (2)示意图
- (3)算法优缺点
- (4)示例代码
- 4. DBSCAN
-
- (1)原理
- (2)示意图
- (3)算法优缺点
- (4)示例代码
- 5. 层次聚类(Hierarchical Clustering)
-
- (1)原理
- (2)示意图
- (3)算法优缺点
- (4)示例代码
- 6. GaussianMixtureModel(高斯混合模型)
-
- (1)原理
- (2)示意图
- (3)算法优缺点
- (4)示例代码
- 7. 谱聚类(Spectral clustering)(仅了解)
-
- (1)原理
- (2)示意图
- (3)背景了解
- (4)示例代码
- 8. Birch(仅了解)
-
- (1)概述
- (2)相关概念
- (3)图解图
- (4)示例代码
- 三. 聚类算法的模型评估
-
- 1. 数据准备、训练、预测
- 2. 模型评估指标
-
- 2.1 非监督式评估(无真实分类结果)
-
- (1)轮廓系数 silhouette_score
- (2)卡林斯基-哈拉巴斯指数 calinski_harabasz_score(越高越好)
- 2.2 监督式评估(有真实分类结果)
-
- (1)(调整的)兰德指数 adjusted_rand_score
- (2)互信息 mutual_info_score
- (3)调整后的互信息 adjusted_mutual_info_score
- (4)同质化得分 homogeneity_score
- (5)完整性得分 completeness_score
- (6)v_measure_score
- 2.3 针对KMeans
-
- (1)inertia_
一. 前言
本文根据以下资料进行整理,对各位大佬表示感谢!
- python数据分析:聚类分析(cluster analysis)
- K-means聚类分析与python实现
- 【聚类】五种主要聚类算法
- [552]python实现聚类算法(6种算法)
二. 典型的聚类算法
常用的聚类算法分为基于划分、层次、密度、网格、统计学、模型等类型的算法,典型算法包括K-Means(经典的聚类算法)、Mean-shift、DBSCAN、层次聚类(主要指“凝聚聚类”,或叫“合并聚类”)、高斯混合、谱聚类、BIRCH等。
1. 聚类算法的选择
(1)高维数据:子空间聚类(如谱聚类);
(2)如果最求更高的分类准确度,那么选择谱聚类将比K-Means准确度更好;
(3)如果数据中存在噪点,那么可以使用基于密度的DBSCAN;
(4)数据量在100万条以内,那么使用K-Means较好;如果数据量超过100万条,那么可以考虑使用Mini Batch K-Means。
2. K-Means(K均值)
(1)原理
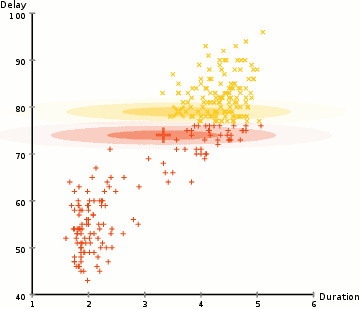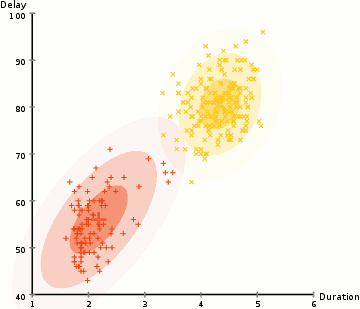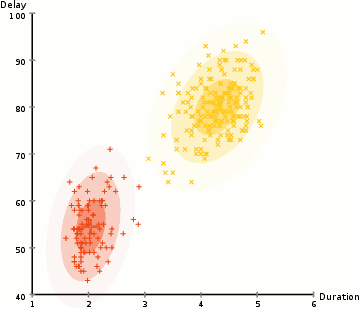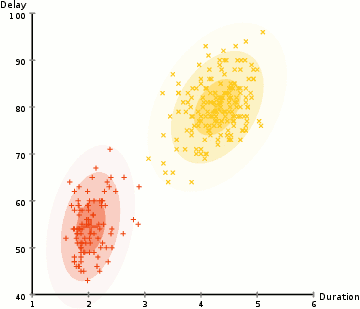
① 首先,我们选择一些类/组来使用并随机地初始化它们各自的中心点。要想知道要使用的类的数量,最好快速地查看一下数据,并尝试识别任何不同的分组。中心点是与每个数据点向量相同长度的向量,在上面的图形中是“X”。
② 每个数据点通过计算点和每个组中心之间的距离进行分类,然后将这个点分类为最接近它的组。
③ 基于这些分类点,我们通过取组中所有向量的均值来重新计算组中心。
④ 对一组迭代重复这些步骤。你还可以选择随机初始化组中心几次,然后选择那些看起来对它提供了最好结果的来运行。
(2)示意图
(3)算法优缺点
优点:
① 对处理大数据集,该算法保持可伸缩性和高效性;
② 算法快速、简单,易于理解(因为我们所做的只是计算 点和群中心之间的距离;它有一个线性复杂度O(n));
缺点:
① 在 K-means 算法中 K 是事先给定的,这个 K 值的选定是非常难以估计的;
② 若簇中含有异常点,将导致均值偏离严重(即:对噪声和孤立点数据敏感);
③ 该算法需要不断地进行样本分类调整,不断地计算调整后的新的聚类中心,因此当数据量非常大时,算法的时间开销是非常大的;
④ 在 K-means 算法中,首先需要根据初始聚类中心来确定一个初始划分,然后对初始划分进行优化。这个初始聚类中心的选择对聚类结果有较大的影响,一旦初始值选择的不好,可能无法得到有效的聚类结果;
⑤ 局部最优解而不是全局优解 (这个和初始点选谁有关)。
(4)注意事项
聚类算法中k-means是最常使用的方法之一,但是k-means要注意数据异常:
① 数据异常值:数据中的异常值能明显改变不同点之间的距离相识度,并且这种影响是非常显著的。因此基于距离相似度的判别模式下,异常值的处理必不可少。
② 数据的异常量纲:不同的维度和变量之间,如果存在数值规模或量纲的差异,那么在做距离之前需要先将变量归一化或标准化。例如跳出率的数值分布区间是[0,1],订单金额可能是[0,10000 000],而订单数量则是[0,1000],如果没有归一化或标准化操作,那么相似度将主要受到订单金额的影响。
(5)示例代码
# coding:utf-8
import random
import imageio
import numpy as np
import pylab as pl
step = 0
color = ['.r', '.g', '.b', '.y'] # 颜色种类
dcolor = ['*r', '*g', '*b', '*y'] # 颜色种类
frames = []
# 计算平面两点的欧氏距离
def distance(a, b):
return (a[0] - b[0]) ** 2 + (a[1] - b[1]) ** 2
# K均值算法
def k_means(x, y, k_count):
# step1: 首先从n个数据对象任意选择k个对象作为初始聚类中心;
count = len(x) # 点的个数
# 从所有点中随机选择K个点
k = random.sample(range(count), k_count)
k_point = [[x[i], [y[i]]] for i in k] # 保证有序
k_point.sort()
global frames
global step
while True:
# step2: 根据每个聚类对象的均值(中心对象),计算每个对象与这些中心对象的距离;并根据最小距离重新对相应对象进行划分;
km = [[] for i in range(k_count)] # 存储每个簇的索引
# 遍历所有点
for i in range(count):
cp = [x[i], y[i]] # 当前点
# 计算cp点到所有质心的距离
_sse = [distance(k_point[j], cp) for j in range(k_count)]
# cp点到那个质心最近
min_index = _sse.index(min(_sse))
# 把cp点并入第i簇
km[min_index].append(i)
# step3: 重新计算每个(有变化)聚类的均值(中心对象);
# 更换质心
step += 1
k_new = []
for i in range(k_count):
_x = sum([x[j] for j in km[i]]) / len(km[i]) # 即每一类中所有点x的平均值
_y = sum([y[j] for j in km[i]]) / len(km[i]) # 即每一类中所有点y的平均值
k_new.append([_x, _y])
k_new.sort() # 排序
# 使用Matplotlab画图
pl.figure()
pl.title("N=%d,k=%d iteration:%d" % (count, k_count, step))
for j in range(k_count):
pl.plot([x[i] for i in km[j]], [y[i] for i in km[j]], color[j % 4])
pl.plot(k_point[j][0], k_point[j][1], dcolor[j % 4])
pl.savefig("1.jpg")
pl.show()
# step4: 循环step2到step3直到每个聚类不再发生变化为止;
frames.append(imageio.imread('1.jpg'))
if (k_new != k_point): # 一直循环直到聚类中心没有变化
k_point = k_new
else:
return km
if __name__ == '__main__':
# x, y = np.loadtxt('2.csv', delimiter=',', unpack=True)
arr = np.random.randint(1000, size=(1000, 1, 2))[:, 0, :]
x = [e[0] for e in arr]
y = [e[1] for e in arr]
k_count = 4
km = k_means(x, y, k_count)
imageio.mimsave('k-means.gif', frames, 'GIF', duration=0.5)
(6)算法拓展–Mini Batch K-Means
K均值在算法稳定性、效率和准确率(相对于真实标签的判别)上表现非常好,并且在应对大量数据时依然如此。它的算法时间复杂度上界为O(nkt),其中n是样本量、k是划分的聚类数、t是迭代次数。当聚类数和迭代次数不变时,K均值的算法消耗时间只跟样本量有关,因此会呈线性增长趋势。
但是当面对海量数据时,k均值算法计算速度慢会产生延时,尤其算法被用于做实时性处理时这种弊端尤为明显。针对K均值的这一问题,很多延伸算法出现了,Mini Batch K-Means就是其中一个典型代表。Mini Batch K-Means使用了一个种名为Mini Batch(分批处理)的方法计算数据点之间的距离。
Mini Batch K-Means的好处是计算过程中不必使用所有的数据样本,而是从不同类别的样本中抽取一部分样本(而非全部样本)作为代表参与聚类算法过程。由于计算样本量少,所以会相应减少运行时间;但另一方面,由于是抽样方法,抽样样本很难完全代表整体样本的全部特征,因此会带来准确度的小幅度下降,但是并不明显。
(7)模型评估
用scikit-learn学习K-Means聚类
3. Mean-shift(均值偏移)
它是一种基于滑动窗口(sliding-window)的算法,它试图找到密集的数据点。而且,它还是一种基于中心的算法,它的目标是定位每一组群/类的中心点,通过更新中心点的候选点来实现滑动窗口中的点的平均值。这些候选窗口在后期处理阶段被过滤,以消除几乎重复的部分,形成最后一组中心点及其对应的组。
(1)原理
核心思想:不断寻找新的圆心坐标,直到密度最大的区域。
以“单滑动窗口的均值偏移聚类”说明均值偏移的原理(结合示意图1,方便理解):
① 以二维空间中的一组点为例,我们从一个以点C(随机选择)为中心的圆形滑窗开始,以半径r为内核。均值偏移是一种爬山算法(hill climbing algorithm),它需要在每个步骤中反复地将这个内核移动到一个更高的密度区域,直到收敛。
② 在每一次迭代中,滑动窗口会移向密度较高的区域,将中心点移动到窗口内的点的平均值(因此得名)。 滑动窗口中的密度与它内部的点的数量成比例。自然地,通过移向窗口中点的平均值,它将逐渐向更高的点密度方向移动。
③ 我们继续根据均值移动滑动窗口,直到没有方向移动可以容纳内核中的更多点。看看上面的图表;我们一直在移动这个圆,直到我们不再增加密度(也就是窗口中的点数)。
④ 步骤①到③的过程是用许多滑动窗口完成的,直到所有的点都位于一个窗口内。当多个滑动窗口重叠的时候,包含最多点的窗口会被保留。然后,数据点根据它们所在的滑动窗口聚类。
(2)示意图
① 单滑动窗口的均值偏移聚类 示意图
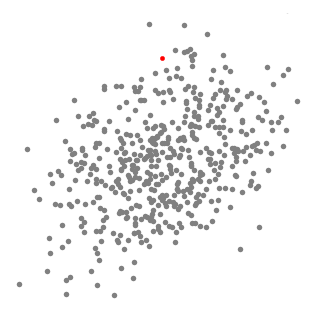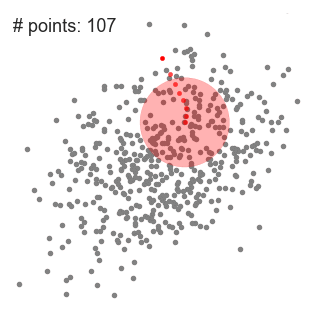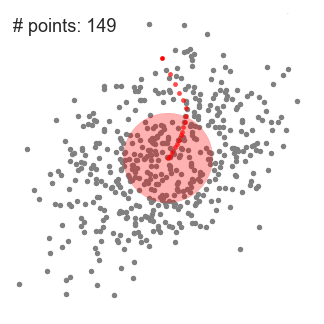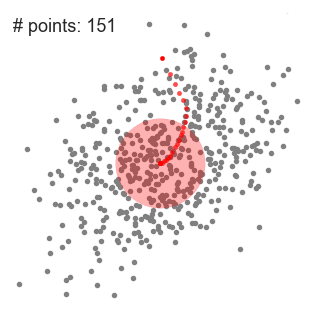
② 下面展示了从端到端所有滑动窗口的整个过程的演示。每个黑点代表一个滑动窗口的质心,每个灰色点都是一个数据点。

(3)算法优缺点
优点:
① 与K-Means聚类相比,均值偏移不需要选择聚类的数量,因为它会自动地发现这一点。这是一个巨大的优势。
② 聚类中心收敛于最大密度点的事实也是非常可取的,因为它非常直观地理解并适合于一种自然数据驱动。
缺点:
选择窗口大小/半径r是非常关键的,所以不能疏忽。
(4)示例代码
# coding:utf-8
from itertools import cycle ##python自带的迭代器模块
import matplotlib.pyplot as plt
import numpy as np
from sklearn.cluster import MeanShift, estimate_bandwidth
from sklearn.datasets.samples_generator import make_blobs
# 产生随机数据的中心
centers = [[1, 1], [-1, -1], [1, -1]]
# 产生的数据个数
n_samples = 10000
# 生产数据
X, _ = make_blobs(n_samples=n_samples, centers=centers, cluster_std=0.6, random_state=0)
# 带宽,也就是以某个点为核心时的搜索半径
bandwidth = estimate_bandwidth(X, quantile=0.2, n_samples=500)
# 设置均值偏移函数
ms = MeanShift(bandwidth=bandwidth, bin_seeding=True)
# 训练数据
ms.fit(X)
# 每个点的标签
labels = ms.labels_
print(labels)
# 簇中心的点的集合
cluster_centers = ms.cluster_centers_
print('cluster_centers:', cluster_centers)
# 总共的标签分类
labels_unique = np.unique(labels)
# 聚簇的个数,即分类的个数
n_clusters_ = len(labels_unique)
print("number of estimated clusters : %d" % n_clusters_)
# 绘图
plt.figure(1)
plt.clf()
colors = cycle('bgrcmykbgrcmykbgrcmykbgrcmyk')
for k, col in zip(range(n_clusters_), colors):
# 根据lables中的值是否等于k,重新组成一个True、False的数组
my_members = labels == k
cluster_center = cluster_centers[k]
# X[my_members, 0] 取出my_members对应位置为True的值的横坐标
print("col: ", col)
plt.plot(X[my_members, 0], X[my_members, 1], col + '.')
plt.plot(cluster_center[0], cluster_center[1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=20)
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
4. DBSCAN
DBSCAN的全称 - - Density-Based Spatial Clustering of Applications with Noise。
中文含义是“基于密度的带有噪声的空间聚类”。
比较有代表性的基于密度的聚类算法,类似于均值转移聚类算法,但它有几个显著的优点。
强调:有异常的数据可以使用DBSCAN聚类方法进行处理。
(1)原理
① DBSCAN以一个从未访问过的任意起始数据点开始。这个点的邻域是用距离ε(所有在ε距离的点都是邻点)来提取的。
② 如果在这个邻域中有足够数量的点(根据 minPoints),那么聚类过程就开始了,并且当前的数据点成为新聚类中的第一个点。否则,该点将被标记为噪声(稍后这个噪声点可能会成为聚类的一部分)。在这两种情况下,这一点都被标记为“访问(visited)”。
③ 对于新聚类中的第一个点,其ε距离附近的点也会成为同一聚类的一部分。这一过程使在ε邻近的所有点都属于同一个聚类,然后重复所有刚刚添加到聚类组的新点。
④ 步骤②和步骤③的过程将重复,直到聚类中的所有点都被确定,就是说在聚类附近的所有点都已被访问和标记。
⑤ 一旦我们完成了当前的聚类,就会检索并处理一个新的未访问点,这将导致进一步的聚类或噪声的发现。这个过程不断地重复,直到所有的点被标记为访问。因为在所有的点都被访问过之后,每一个点都被标记为属于一个聚类或者是噪音。
(2)示意图
(3)算法优缺点
优点:
① 对原始数据分布规律没有明显要求,能适应任意数据集分布形状的空间聚类,因此数据集适用性更广,尤其是对非凸装、圆环形等异性簇分布的识别较好。
② 无需指定聚类数量,对结果的先验要求不高。
③ 由于DBSCAN可区分核心对象、边界点和噪点,因此对噪声的过滤效果好,能有效应对数据噪点。
缺点:
它对整个数据集进行操作且聚类时使用了一个全局性的表征密度的参数,因此也存在比较明显的弱点:
① 对于高维度问题,基于半径(距离ε)和密度(minPoints)的定义成问题。
② 当簇的密度变化太大时,聚类结果较差。
③ 当数据量增大时,要求较大的内存支持,I/O消耗也很大。
(4)示例代码
# coding:utf-8
from sklearn.datasets.samples_generator import make_blobs
from sklearn.cluster import DBSCAN
import numpy as np
import matplotlib.pyplot as plt
# from itertools import cycle ##python自带的迭代器模块
# from sklearn.preprocessing import StandardScaler
# 产生随机数据的中心
centers = [[1, 1], [-1, -1], [1, -1]]
# 产生的数据个数
n_samples = 750
# 生产数据:此实验结果受cluster_std的影响,或者说受eps 和cluster_std差值影响
X, lables_true = make_blobs(n_samples=n_samples, centers=centers, cluster_std=0.4,
random_state=0)
# 设置分层聚类函数
db = DBSCAN(eps=0.3, min_samples=10)
# eps--扫描半径;min_samples--作为核心点的话邻域(即以其为圆心,eps为半径的圆,含圆上的点)中的最小样本数(包括点本身)
# 训练数据
db.fit(X)
# 初始化一个全是False的bool类型的数组
core_samples_mask = np.zeros_like(db.labels_, dtype=bool)
'''
这里是关键点(针对这行代码:xy = X[class_member_mask & ~core_samples_mask]):
db.core_sample_indices_ 表示的是某个点在寻找核心点集合的过程中暂时被标为噪声点的点(即周围点
小于min_samples),并不是最终的噪声点。在对核心点进行联通的过程中,这部分点会被进行重新归类(即标签
并不会是表示噪声点的-1),也可也这样理解,这些点不适合做核心点,但是会被包含在某个核心点的范围之内
'''
core_samples_mask[db.core_sample_indices_] = True
# 每个数据的分类
lables = db.labels_
# 分类个数:lables中包含-1,表示噪声点
n_clusters_ = len(np.unique(lables)) - (1 if -1 in lables else 0)
# 绘图
unique_labels = set(lables)
'''
1)np.linspace 返回[0,1]之间的len(unique_labels) 个数
2)plt.cm 一个颜色映射模块
3)生成的每个colors包含4个值,分别是rgba
4)其实这行代码的意思就是生成4个可以和光谱对应的颜色值
'''
colors = plt.cm.Spectral(np.linspace(0, 1, len(unique_labels)))
plt.figure(1)
plt.clf()
for k, col in zip(unique_labels, colors):
# -1表示噪声点,这里的k表示黑色
if k == -1:
col = 'k'
# 生成一个True、False数组,lables == k 的设置成True
class_member_mask = (lables == k)
# 两个数组做&运算,找出即是核心点又等于分类k的值 markeredgecolor='k',
xy = X[class_member_mask & core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', c=col, markersize=14)
'''
1)~优先级最高,按位对core_samples_mask 求反,求出的是噪音点的位置
2)& 与运算之后,求出虽然刚开始是噪音点的位置,但是重新归类却属于k的点
3)对核心分类之后进行的扩展
'''
xy = X[class_member_mask & ~core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', c=col, markersize=3)
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
5. 层次聚类(Hierarchical Clustering)
分为两类:自上而下(很少用)或自下而上。
自下而上的算法在一开始就将每个数据点视为一个单一的聚类,然后依次合并(或聚集)类,直到所有类合并成一个包含所有数据点的单一聚类。因此,自下而上的层次聚类称为合成聚类或HAC。合成聚类的层次结构用一棵树(或树状图)表示。树的根是收集所有样本的唯一聚类,而叶子是只有一个样本的聚类。
以下主要讲合成聚类(“凝聚聚类”)
(1)原理
① 首先将每个数据点作为一个单独的聚类进行处理。如果我们的数据集有X个数据点,那么我们就有了X个聚类。然后我们选择一个度量两个聚类之间距离的距离度量。作为一个示例,我们将使用平均连接(average linkage)聚类,它定义了两个聚类之间的距离,即第一个聚类中的数据点和第二个聚类中的数据点之间的平均距离。
② 在每次迭代中,我们将两个聚类合并为一个。将两个聚类合并为具有最小平均连接的组。比如说根据我们选择的距离度量,这两个聚类之间的距离最小,因此是最相似的,应该组合在一起。
③ 重复步骤②直到我们到达树的根。我们只有一个包含所有数据点的聚类。通过这种方式,我们可以选择最终需要多少个聚类,只需选择何时停止合并聚类,也就是我们停止建造这棵树的时候!
(2)示意图
(3)算法优缺点
优点:
① 不要求我们指定聚类的数量,我们甚至可以选择哪个聚类看起来最好。
② 该算法对距离度量的选择不敏感;它们的工作方式都很好,而对于其他聚类算法,距离度量的选择是至关重要的。
③ 层次聚类方法的一个特别好的用例是,当底层数据具有层次结构时,你可以恢复层次结构;而其他的聚类算法无法做到这一点。
缺点:
层次聚类的优点是以低效率为代价的,因为它具有O(n³)的时间复杂度,与K-Means和高斯混合模型的线性复杂度不同。
(4)示例代码
# coding:utf-8
# 层次聚类
from sklearn.datasets.samples_generator import make_blobs
from sklearn.cluster import AgglomerativeClustering
import matplotlib.pyplot as plt
from itertools import cycle ##python自带的迭代器模块
# 产生随机数据的中心
centers = [[1, 1], [-1, -1], [1, -1]]
# 产生的数据个数
n_samples = 3000
# 生产数据
X, lables_true = make_blobs(n_samples=n_samples, centers=centers, cluster_std=0.6,
random_state=0)
# 设置分层聚类函数
linkages = ['ward', 'average', 'complete']
# linkage:指定层次聚类判断相似度的方法,有以下三种:
# ward:组间距离等于两组对象之间的最小距离。(即single-linkage聚类)
# average:组间距离等于两组对象之间的平均距离。(average-linkage聚类)
# complete:组间距离等于两组对象之间的最大距离。(complete-linkage聚类)
n_clusters_ = 3
ac = AgglomerativeClustering(linkage=linkages[0], n_clusters=n_clusters_)
# 训练数据
ac.fit(X)
# 每个数据的分类
lables = ac.labels_
# 绘图
plt.figure(1)
plt.clf()
colors = cycle('bgrcmykbgrcmykbgrcmykbgrcmyk')
for k, col in zip(range(n_clusters_), colors):
# 根据lables中的值是否等于k,重新组成一个True、False的数组
my_members = lables == k
# X[my_members, 0] 取出my_members对应位置为True的值的横坐标
plt.plot(X[my_members, 0], X[my_members, 1], col + '.')
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
6. GaussianMixtureModel(高斯混合模型)
基本思想:任意形状的概率分布都可以用多个高斯分布函数去近似,也就是说GMM是由多个单高斯密度分布组成的,每个Gaussian叫一个"Component"(即一个聚类),这些"Component"线性加成在一起就组成了 GMM 的概率密度函数,也就是下面的函数:

k - - 模型的个数,即Component的个数(聚类的个数);
πk - - 第k个高斯的权重;
p(x|k) - - 第k个高斯概率密度,其均值为μk,方差为σk 。
上述参数,除了k是直接给定之外,其他参数都是通过EM算法估算出来的。
为了找到每个聚类的高斯分布的参数(例如平均值和标准差)我们将使用一种叫做期望最大化(EM)的优化算法。看看下面的示意图,就可以看到高斯混合模型是被拟合到聚类上的。然后,我们可以继续进行期望的过程 - - 使用高斯混合模型实现最大化聚类。
(1)原理
使用高斯混合模型来期望最大化聚类
① 我们首先选择聚类的数量(如K-Means所做的那样),然后随机初始化每个聚类的高斯分布参数。通过快速查看数据,可以尝试为初始参数提供良好的猜测。注意,在下面的示意图中可以看到,这并不是100%的必要,因为高斯开始时的表现非常不好,但是很快就被优化了。
② 给定每个聚类的高斯分布,计算每个数据点属于特定聚类的概率。一个点离高斯中心越近,它就越有可能属于那个聚类。这应该是很直观的,因为有一个高斯分布,我们假设大部分的数据都离聚类中心很近。
③ 基于这些概率,我们为高斯分布计算一组新的参数,这样我们就能最大程度地利用聚类中的数据点的概率。我们使用数据点位置的加权和来计算这些新参数,权重是属于该特定聚类的数据点的概率。为了解释这一点,我们可以看一下下面的图,特别是黄色的聚类作为例子。分布在第一次迭代中是随机的,但是我们可以看到大多数的黄色点都在这个分布的右边。当我们计算一个由概率加权的和,即使在中心附近有一些点,它们中的大部分都在右边。因此,自然分布的均值更接近于这些点。我们还可以看到,大多数点都是“从右上角到左下角”。因此,标准差的变化是为了创造一个更符合这些点的椭圆,从而使概率的总和最大化。
④ 步骤②和③被迭代地重复,直到收敛,在那里,分布不会从迭代到迭代这个过程中变化很多。
(2)示意图
(3)算法优缺点
优点:
① 高斯混合模型在聚类协方差方面比K-Means要灵活得多;根据标准差参数,聚类可以采用任何椭圆形状,而不是局限于圆形。K-Means实际上是高斯混合模型的一个特例,每个聚类在所有维度上的协方差都接近0。
② 根据高斯混合模型的使用概率,每个数据点可以有多个聚类。因此,如果一个数据点位于两个重叠的聚类的中间,通过说X%属于1类,而y%属于2类,我们可以简单地定义它的类。
(4)示例代码
# coding:utf8
import matplotlib.pyplot as plt
from sklearn.datasets.samples_generator import make_blobs
from sklearn.mixture import GaussianMixture
# X为样本特征,Y为样本簇类别, 共1000个样本,每个样本2个特征,共4个簇,簇中心在[-1,-1], [0,0],[1,1], [2,2]
X, y = make_blobs(n_samples=1000, n_features=2, centers=[[-1, -1], [0, 0], [1, 1], [2, 2]],cluster_std=[0.4, 0.3, 0.4, 0.3], random_state=0)
# 设置gmm函数
# covariance_type: 通过EM算法估算参数时使用的协方差类型,默认是"full"
# full:每个模型使用自己的一般协方差矩阵
# tied:所用模型共享一个一般协方差矩阵
# diag:每个模型使用自己的对角线协方差矩阵
# spherical:每个模型使用自己的单一方差
# n_components:高斯模型的个数,即聚类的目标个数
gmm = GaussianMixture(n_components=4, covariance_type='full').fit(X)
# 训练数据
y_pred = gmm.predict(X)
# 绘图
plt.scatter(X[:, 0], X[:, 1], c=y_pred)
plt.show()
7. 谱聚类(Spectral clustering)(仅了解)
是一种基于图论的聚类方法,它能够识别任意形状的样本空间且收敛于全局最有解,其基本思想是利用样本数据的相似矩阵进行特征分解后得到的特征向量进行聚类。它与样本特征无关而只与样本个数有关。
(1)原理
基本思路: 将样本看作顶点,样本间的相似度看作带权的边,从而将聚类问题转为图分割问题:找到一种图分割的方法使得连接不同组的边的权重尽可能低(这意味着组间相似度要尽可能低),组内的边的权重尽可能高(这意味着组内相似度要尽可能高)。
(2)示意图
(3)背景了解
在大数据背景下,有很多高维度数据场景,如电子商务交易数据、web文本数据日益丰富。高维数据聚类时耗时长、聚类结果准确性和稳定性都不尽如人意。因为,在高维数据,基于距离的相似度计算效率极低;特征值过多在所有维度上存在簇的可能性非常低;由于稀疏性和紧邻特性,基于距离的相似度几乎为0,导致高维空间很难出现数据簇。这时我们可以选着使用子空间聚类,或是降维处理。
子空间聚类算法是在高维数据空间中对传统聚类算法的一种扩展,其思想是选取与给定簇密切相关的维,然后在对应的子空间进行聚类。 比如谱聚类就是一种子空间聚类方法,由于选择相关维的方法以及评估子空间的方法需要自定义,因此这种方法对操作者的要求较高。
(4)示例代码
# coding:utf8
# 谱聚类
from sklearn.datasets.samples_generator import make_blobs
from sklearn.cluster import spectral_clustering
import numpy as np
import matplotlib.pyplot as plt
from sklearn import metrics
from itertools import cycle # python自带的迭代器模块
# 产生随机数据的中心
centers = [[1, 1], [-1, -1], [1, -1]]
# 产生的数据个数
n_samples = 3000
# 生产数据
X, lables_true = make_blobs(n_samples=n_samples, centers=centers, cluster_std=0.6, random_state=0)
print("X -- ", X)
print("X.shape -- ", X.shape)
# 变换成矩阵,输入必须是对称矩阵
metrics_metrix = (-1 * metrics.pairwise.pairwise_distances(X)).astype(np.int32) # metrics.pairwise.pairwise_distances计算行向量之间的距离
print(metrics_metrix)
print(metrics_metrix.min()) # -6
metrics_metrix += -1 * metrics_metrix.min()
# 设置谱聚类函数
n_clusters_ = 4
lables = spectral_clustering(metrics_metrix, n_clusters=n_clusters_)
print(metrics_metrix.shape)
print(metrics_metrix)
# 绘图
plt.figure(1)
plt.clf()
colors = cycle('bgrcmykbgrcmykbgrcmykbgrcmyk')
for k, col in zip(range(n_clusters_), colors):
# 根据lables中的值是否等于k,重新组成一个True、False的数组
my_members = lables == k
# X[my_members, 0] 取出my_members对应位置为True的值的横坐标
plt.plot(X[my_members, 0], X[my_members, 1], col + '.')
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
8. Birch(仅了解)
(1)概述
就是通过聚类特征(CF)形成一个聚类特征树,root层的CF个数就是聚类个数。
(2)相关概念
聚类特征(CF):每一个CF是一个三元组,可以用(N,LS,SS)表示。其中N代表了这个CF中拥有的样本点的数量;LS代表了这个CF中拥有的样本点各特征维度的和向量,SS代表了这个CF中拥有的样本点各特征维度的平方和。
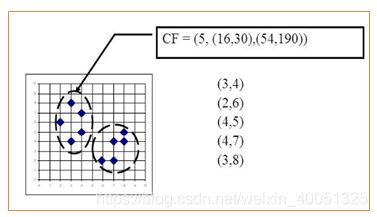
如上图所示:N = 5
LS=(3+2+4+4+3,4+6+5+7+8)=(16,30)
SS =(32+22+42+42+32,42+62+52+72+82)=(54,190)
(3)图解图
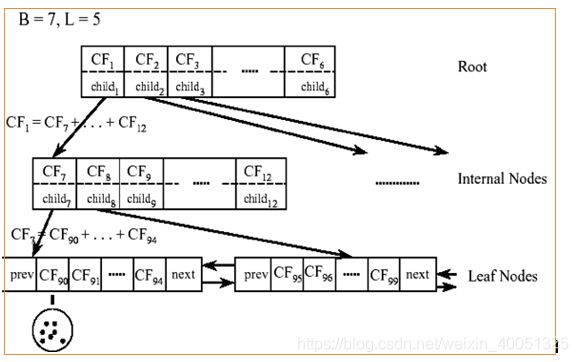
对于上图中的CF Tree,限定了B=7,L=5, 也就是说内部节点最多有7个CF(CF90下的圆),而叶子节点最多有5个CF(CF90到CF94)。叶子节点是通过双向链表连通的。
(4)示例代码
# coding:utf8
import matplotlib.pyplot as plt
from sklearn.cluster import Birch
from sklearn.datasets.samples_generator import make_blobs
# X为样本特征,Y为样本簇类别, 共1000个样本,每个样本2个特征,共4个簇,簇中心在[-1,-1], [0,0],[1,1], [2,2]
X, y = make_blobs(n_samples=1000, n_features=2, centers=[[-1, -1], [0, 0], [1, 1], [2, 2]],
cluster_std=[0.4, 0.3, 0.4, 0.3], random_state=9)
# 设置birch函数
# birch = Birch(n_clusters=None)
birch = Birch(n_clusters=4)
# 训练数据
y_pred = birch.fit_predict(X)
# 绘图
plt.scatter(X[:, 0], X[:, 1], c=y_pred)
plt.show()
三. 聚类算法的模型评估
(以K-Means算法为例)
1. 数据准备、训练、预测
import numpy as np
import pandas as pd
from sklearn.cluster import KMeans
from sklearn import metrics
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
# 数据准备
data = make_blobs(n_samples=2000, centers=[[1,1],[-1,-1]], cluster_std=0.7, random_state=1)
X = data[0]
y = data[1]
# 设置聚类数量
n_clusters = 2
# 设置聚类模型对象
kmeans = KMeans(n_clusters=n_clusters, random_state=2018)
# 训练聚类模型
kmeans.fit(X)
# 预测聚类模型
pred_y = kmeans.predict(X)
2. 模型评估指标
2.1 非监督式评估(无真实分类结果)
(1)轮廓系数 silhouette_score
① 定义: 结合内聚度和分离度两种因素,其最高值为1,最差值为-1,0附近的值表示重叠的聚类,负值通常表示样本已被分配到错误的集群。可以用来在相同原始数据的基础上用来评价不同算法、或者算法不同运行方式对聚类结果所产生的影响。
以下内容转载参考: 聚类评估算法-轮廓系数(Silhouette Coefficient )
② 原理/方法:
step1. 计算样本i到同簇其他样本的平均距离ai。ai 越小,说明样本i越应该被聚类到该簇,将ai 称为样本i的簇内不相似度。 簇C中所有样本的ai 均值称为簇C的簇不相似度。
step2. 计算样本i到其他某簇Cj 的所有样本的平均距离bij,称为样本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:bi =min{bi1, bi2, …, bik}。bi越大,说明样本i越不属于其他簇。
step3. 根据样本i的簇内不相似度a i 和簇间不相似度b i ,定义样本i的轮廓系数:
 step4. 判断:
step4. 判断:
si接近1,则说明样本i聚类合理;
si接近-1,则说明样本i更应该分类到另外的簇;
若si 近似为0,则说明样本i在两个簇的边界上。
所有样本的s i 的均值称为聚类结果的轮廓系数,是该聚类是否合理、有效的度量。
③ 代码示例:
silhouette_s = metrics.silhouette_score(X, pred_y, metric='euclidean')
print("silhouette_s---", silhouette_s) # 0.58674
(2)卡林斯基-哈拉巴斯指数 calinski_harabasz_score(越高越好)
① 定义: 该分数定义为簇间离散与簇内离散的比值,它是一种非监督式评估指标。
② 代码示例:
calinski_harabaz_s = metrics.calinski_harabasz_score(X, pred_y)
print("calinski_harabaz_s---", calinski_harabaz_s) # 4474.03821
2.2 监督式评估(有真实分类结果)
(1)(调整的)兰德指数 adjusted_rand_score
② 代码示例:
ajusted_rand_s = metrics.adjusted_rand_score(y, pred_y)
print("ajusted_rand_s---", ajusted_rand_s) # 0.91389
(2)互信息 mutual_info_score
① 定义: 互信息(Mutual Information, MI),互信息是一个随机变量中包含的关于另一个随机变量的信息量,在这里指的是相同数据的两个标签之间的相似度的量度,结果是非负值。
② 代码示例:
mutual_info_s = metrics.mutual_info_score(y, pred_y)
print("mutual_info_s---", mutual_info_s) # 0.58751
(3)调整后的互信息 adjusted_mutual_info_score
① 定义: (Adjusted Mutual Information, AMI),调整后的互信息是对互信息评分的调整得分。它考虑到对于具有更大数量的聚类群,通常MI较高,而不管实际上是否有更多的信息共享,它通过调整聚类群的概率来纠正这种影响。当两个聚类集相同(即完全匹配)时,AMI返回值为1;随机分区(独立标签)平均预期AMI约为0,也可能为负数。
② 代码示例:
adjusted_mutual_info_s = metrics.adjusted_mutual_info_score(y, pred_y)
print("adjusted_mutual_info_s---", adjusted_mutual_info_s) # 0.84755
(4)同质化得分 homogeneity_score
① 定义: (Homogeneity),如果所有的聚类都只包含属于单个类的成员的数据点,则聚类结果将满足同质性。其取值范围[0,1],值越大意味着聚类结果与真实情况越吻合。
② 代码示例:
homogeneity_s = metrics.homogeneity_score(y, pred_y)
print("homogeneity_s---", homogeneity_s) # 0.84760
(5)完整性得分 completeness_score
① 定义: 如果作为给定类的成员的所有数据点是相同集群的元素,则聚类结果满足完整性。其取值范围[0,1],值越大意味着聚类结果与真实情况越吻合。
② 代码示例:
completeness_s = metrics.completeness_score(y, pred_y)
print("completeness_s---", completeness_s) # 0.84761
(6)v_measure_score
① 定义: 它是同质化和完整性之间的谐波平均值,v = 2 (均匀性 * 完整性)/(均匀性+完整性)。其取值范围[0,1],值越大意味着聚类结果与真实情况越吻合。
② 代码示例:
v_measure_s = metrics.v_measure_score(y, pred_y)
print("v_measure_s---", v_measure_s) # 0.84760
2.3 针对KMeans
(1)inertia_
① 定义: inertia_(含义“惯性、惰性”)是K-Means模型对象的属性,表示样本点距离最近聚类中心的距离的总和,它是作为在没有真实分类结果标签下的非监督式评估指标。该值越小越好,值越小证明样本在类间的分布越集中,即类内的距离越小。
② 代码示例:
inertias = kmeans.inertia_
print("inertias---", inertias) # 1874.5899