CORD-19数据集以及相关分析代码介绍
写在前面
最近发现了一个有关新冠疫情的数公开据集,CORD-19。CORD-19是有关COVID-19和相关历史冠状病毒研究的不断增长的科学论文资源。 CORD-19旨在通过其丰富的元数据和结构化全文本来促进文本挖掘和信息检索系统的开发。 自发布以来,CORD-19已下载超过75,000次,并已成为许多COVID-19文本挖掘和发现系统的基础。 在本文中,我们描述了数据集构建的机制,重点介绍了挑战和关键设计决策,概述了如何使用CORD-19,并预览了围绕数据集构建的工具和即将出现的共享任务。 我们希望该资源将继续使计算机界,生物医学专家和决策者聚集在一起,以寻求有效的COVID-19治疗和管理政策。 一经发布在kaggle平台上就引发了研究的热潮,这里贴上网址:https://www.kaggle.com/allen-institute-for-ai/CORD-19-research-challenge。这份数据集包括csv元数据和json文件,可以用于信息检索,信息抽取,知识图谱,问答对,预训练模型,摘要,推荐,蕴含,辅助文献综述,增强阅读等多个方向,适合广大机器学习爱好者学习研究,以寻求有效的COVID-19治疗和管理政策。
博主这里介绍一份kaggle上聚类分析的代码,虽然这份代码没有用现在流行的一些预训练模型进行分类任务,但是它处理这些数据的思路以及对聚类相关工具的熟练使用值得借鉴。网址:https://www.kaggle.com/maksimeren/covid-19-literature-clustering
目标
鉴于大量文献和COVID-19的迅速普及,卫生专业人员很难掌握有关该病毒的新信息。我们通过使用聚类进行标记和降维以进行可视化,可以通过散点图来表示文献的集合。在此图上,主题高度相似的出版物将共享一个标签,并且彼此靠近绘制。为了找到群集中的含义,将执行主题建模以查找每个群集的关键字。
通过使用Bokeh,该图将是交互式的。用户可以选择查看整体图,也可以按群集过滤数据。如果需要缩小范围,该图还将具有搜索功能,该功能会将输出限制为仅包含搜索项的论文。将鼠标悬停在绘图上的点上将提供基本信息,例如标题,作者,期刊和摘要。单击某个点将显示一个带有URL的菜单,该URL可用于访问完整出版物。
这是一个艰难的时期,医护人员,环卫人员和许多其他重要人员无法离开世界。在遵守隔离协议的同时,Kaggle CORD-19竞赛为我们提供了机会,以计算机科学系学生的最佳方式提供帮助。但是,应该指出的是,我们不是流行病学家,也不是我们评估这些论文重要性的地方。创建该工具是为了帮助受过培训的专业人员更轻松地筛选与该病毒有关的许多出版物,并找到自己的决定。
方法
-
使用自然语言处理(NLP)从每个文档的正文中解析文本。
-
使用TF-IDF将每个文档实例di转换为特征向量Xi。
-
使用t分布随机邻近嵌入(t-SNE)将降维应用于每个特征向量Xi,以将相似的研究文章聚类在二维平面XY1中。
-
使用主成分分析(PCA)将X的尺寸投影到多个尺寸,这些尺寸将保持0.95的方差,同时消除嵌入Y2时的噪声和离群值。
-
在Y2上应用k均值聚类,其中k为20,以标记Y1上的每个聚类。
-
使用潜在狄利克雷分配(LDA)在X上应用主题建模,以从每个集群中发现关键字。
-
在情节上可视地调查聚类,根据需要缩小到特定文章,并使用随机梯度下降(SGD)进行分类。
1 加载数据
加载csv数据
import numpy as np # linear algebra
import pandas as pd # data processing, CSV file I/O (e.g. pd.read_csv)
import glob
import json
import matplotlib.pyplot as plt
plt.style.use('ggplot')
metadata_path = 'data/metadata.csv'
meta_df = pd.read_csv(metadata_path, dtype={
'pubmed_id': str,
'Microsoft Academic Paper ID': str,
'doi': str
})
meta_df.head()
加载json文件
辅助函数
构建一些辅助用的函数,读取json文件,在字符长度达到一定数量时在每个单词后添加断点。 这是用于交互式绘图的,因此悬停工具适合屏幕。
class FileReader:
def __init__(self, file_path):
with open(file_path) as file:
content = json.load(file)
self.paper_id = content['paper_id']
self.abstract = []
self.body_text = []
# Abstract
for entry in content['abstract']:
self.abstract.append(entry['text'])
# Body text
for entry in content['body_text']:
self.body_text.append(entry['text'])
self.abstract = '\n'.join(self.abstract)
self.body_text = '\n'.join(self.body_text)
def __repr__(self):
return f'{self.paper_id}: {self.abstract[:200]}... {self.body_text[:200]}...'
first_row = FileReader(all_json[0])
print(first_row)def get_breaks(content, length):
data = ""
words = content.split(' ')
total_chars = 0
# add break every length characters
for i in range(len(words)):
total_chars += len(words[i])
if total_chars > length:
data = data + "
" + words[i]
total_chars = 0
else:
data = data + " " + words[i]
return data将json文件转换为DataFrame
dict_ = {'paper_id': [], 'doi':[], 'abstract': [], 'body_text': [], 'authors': [], 'title': [], 'journal': [], 'abstract_summary': []}
for idx, entry in enumerate(all_json):
if idx % (len(all_json) // 10) == 0:
print(f'Processing index: {idx} of {len(all_json)}')
try:
content = FileReader(entry)
except Exception as e:
continue # invalid paper format, skip
# get metadata information
meta_data = meta_df.loc[meta_df['sha'] == content.paper_id]
# no metadata, skip this paper
if len(meta_data) == 0:
continue
dict_['abstract'].append(content.abstract)
dict_['paper_id'].append(content.paper_id)
dict_['body_text'].append(content.body_text)
# also create a column for the summary of abstract to be used in a plot
if len(content.abstract) == 0:
# no abstract provided
dict_['abstract_summary'].append("Not provided.")
elif len(content.abstract.split(' ')) > 100:
# abstract provided is too long for plot, take first 100 words append with ...
info = content.abstract.split(' ')[:100]
summary = get_breaks(' '.join(info), 40)
dict_['abstract_summary'].append(summary + "...")
else:
# abstract is short enough
summary = get_breaks(content.abstract, 40)
dict_['abstract_summary'].append(summary)
# get metadata information
meta_data = meta_df.loc[meta_df['sha'] == content.paper_id]
try:
# if more than one author
authors = meta_data['authors'].values[0].split(';')
if len(authors) > 2:
# if more than 2 authors, take them all with html tag breaks in between
dict_['authors'].append(get_breaks('. '.join(authors), 40))
else:
# authors will fit in plot
dict_['authors'].append(". ".join(authors))
except Exception as e:
# if only one author - or Null valie
dict_['authors'].append(meta_data['authors'].values[0])
# add the title information, add breaks when needed
try:
title = get_breaks(meta_data['title'].values[0], 40)
dict_['title'].append(title)
# if title was not provided
except Exception as e:
dict_['title'].append(meta_data['title'].values[0])
# add the journal information
dict_['journal'].append(meta_data['journal'].values[0])
# add doi
dict_['doi'].append(meta_data['doi'].values[0])
df_covid = pd.DataFrame(dict_, columns=['paper_id', 'doi', 'abstract', 'body_text', 'authors', 'title', 'journal', 'abstract_summary'])一些特征工程
df_covid['abstract_word_count'] = df_covid['abstract'].apply(lambda x: len(x.strip().split())) # word count in abstract
df_covid['body_word_count'] = df_covid['body_text'].apply(lambda x: len(x.strip().split())) # word count in body
df_covid['body_unique_words']=df_covid['body_text'].apply(lambda x:len(set(str(x).split()))) # number of unique words in body
df_covid.drop_duplicates(['abstract', 'body_text'], inplace=True)
2 数据预处理
取前10000条数据为例
df = df_covid.sample(10000, random_state=42)
del df_covid
## 去除空值
df.dropna(inplace=True)
df.info()处理多种语言问题
from tqdm import tqdm
from langdetect import detect
from langdetect import DetectorFactory
# set seed
DetectorFactory.seed = 0
# hold label - language
languages = []
# go through each text
for ii in tqdm(range(0,len(df))):
# split by space into list, take the first x intex, join with space
text = df.iloc[ii]['body_text'].split(" ")
lang = "en"
try:
if len(text) > 50:
lang = detect(" ".join(text[:50]))
elif len(text) > 0:
lang = detect(" ".join(text[:len(text)]))
# ught... beginning of the document was not in a good format
except Exception as e:
all_words = set(text)
try:
lang = detect(" ".join(all_words))
# what!! :( let's see if we can find any text in abstract...
except Exception as e:
try:
# let's try to label it through the abstract then
lang = detect(df.iloc[ii]['abstract_summary'])
except Exception as e:
lang = "unknown"
pass
# get the language
languages.append(lang)看一下语言分布:
把不是英语的扔掉df = df[df['language'] == 'en']
使用spacy处理一下正文文本(body text):
# Download the spacy bio parser
from IPython.utils import io
with io.capture_output() as captured:
!pip install https://s3-us-west-2.amazonaws.com/ai2-s2-scispacy/releases/v0.2.4/en_core_sci_lg-0.2.4.tar.gz
#NLP
import spacy
from spacy.lang.en.stop_words import STOP_WORDS
import en_core_sci_lg # model downloaded in previous step
import string
punctuations = string.punctuation
stopwords = list(STOP_WORDS)
stopwords[:10]
custom_stop_words = [
'doi', 'preprint', 'copyright', 'peer', 'reviewed', 'org', 'https', 'et', 'al', 'author', 'figure',
'rights', 'reserved', 'permission', 'used', 'using', 'biorxiv', 'medrxiv', 'license', 'fig', 'fig.',
'al.', 'Elsevier', 'PMC', 'CZI', 'www'
]
for w in custom_stop_words:
if w not in stopwords:
stopwords.append(w)
# Parser
parser = en_core_sci_lg.load(disable=["tagger", "ner"])
parser.max_length = 7000000
def spacy_tokenizer(sentence):
mytokens = parser(sentence)
# 大写转小写
mytokens = [word.lemma_.lower().strip() if word.lemma_ != "-PRON-" else word.lower_ for word in mytokens]
# 去除停用词
mytokens = [word for word in mytokens if word not in stopwords and word not in punctuations]
mytokens = " ".join([i for i in mytokens])
return mytokens
tqdm.pandas()
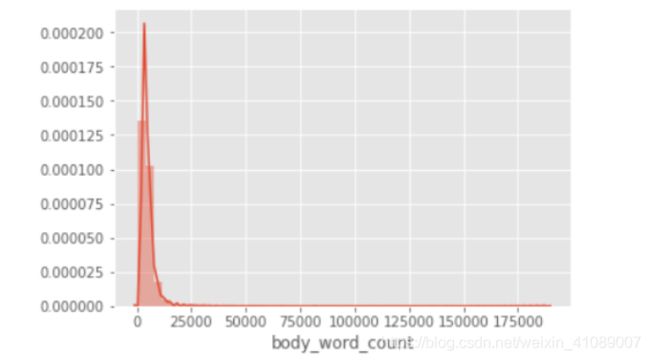
df["processed_text"] = df["body_text"].progress_apply(spacy_tokenizer)看看正文文本的字数统计。
这幅图使我们对正在处理的内容有了很好的了解。 大多数论文的长度约为5000字。 两个图中的长尾巴都是由异常值引起的。 实际上,约有98%的论文篇幅少于20,000个字,而精选的论文则超过200,000个!
3 向量化
现在我们已经对数据进行了预处理,是时候将其转换为可以由我们的算法处理的格式了。 为此,我们将使用tf-idf。 这会将字符串格式的数据转换为衡量每个单词对整个文献中实例的重要性的度量。
from sklearn.feature_extraction.text import TfidfVectorizer
def vectorize(text, maxx_features):
vectorizer = TfidfVectorizer(max_features=maxx_features)
X = vectorizer.fit_transformer(text)
return X向量化我们的数据。 我们将基于正文文本的内容进行聚类。特征的最大数量将受到限制, 仅将使用前个特征,从而可以用作噪声过滤器。 此外,更多特征会导致痛苦的长时间运行。
text = df['processed_text'].values
X = vectorize(text, 2**12)
## X.shape = (9087,4096)4 PCA&聚类
让我们看看在保持95%的差异的同时我们可以减少多少维度。 我们将对我们的矢量化数据应用主成分分析(PCA)。这样做的原因是,通过使用PCA保留大量维数,不会破坏很多信息,同时可以从数据中消除一些噪声/离群值,并使k均值的聚类问题更加容易。请注意,X_reduced仅用于k-均值,t-SNE仍将使用通过tf-idf对NLP处理的文本生成的原始特征向量X。
from slearn.decomposition import PCA
pca = PCA(n_components=0.95, random_state=42)
X_reduced = pca.fit_transform(X.toarray())
## X_reduced.shape = (9087,2107)为了分开文献,将在矢量化文本上运行k均值。 给定簇数k,k均值将通过取到随机初始化质心的平均距离的方式对每个向量进行分类,质心随着迭代更新。那么具体选择多少类呢,为了找到最佳k值,我们将研究不同k值下的失真。失真计算从每个点到其指定中心的平方距离之和。当对k绘制失真图时,将有一个k值,在该值之后失真的减少是最小的。这是所需的群集数。
from sklearn.cluster import KMeans
from sklearn import metrics
from scipy.spatial.distance import cdist
# run kmeans with many different k
distortions = []
K = range(2, 50)
for k in K:
kmeans = KMeans(n_clusters=k, random_state=42)
kmeans.fit(X_reduce)
distortions.append(sum(np.min(cdist(X_reduced, k_means.cluster_centers_, 'euclidean'), axis=1)) / X.shape[0])
X_line = [K[0], K[-1]]
Y_line = [distortions[0], distortions[-1]]
# Plot the elbow
plt.plot(K, distortions, 'b-')
plt.plot(X_line, Y_line, 'r')
plt.xlabel('k')
plt.ylabel('Distortion')
plt.title('The Elbow Method showing the optimal k')
plt.show()在该图中,我们可以看到更好的k值在18-25之间。 此后,失真的降低就不那么明显了。 为简单起见,我们将使用k = 20。
k = 20
kmeans = KMeans(n_clusters=k, random_state=42)
y_pred = kmeans.fit_predict(X_reduced)
df['y'] = y_pred5 使用t-SNE降维
使用t-SNE,我们可以将高维特征向量缩减为2维。 通过使用2维作为x,y坐标,可以绘制正文文本。t分布随机邻近嵌入(t-SNE)降低了维数,同时尝试使相似实例聚集而异质实例分开。它主要用于可视化,尤其是可视化高维空间中的实例簇。
from sklearn.manifold impor TSNE
tsne = TSNE(verbose=1, perplexity=100, random_state=42)
X_embedded = tsne.fit_transform(X.toarray())让我们来看看我们的数据压缩到2维时是什么样子的。
from matplotlib import pyplot as plt
import seaborn as sns
# sns settings
sns.set(rc={'figure.figsize':(15,15)})
# colors
palette = sns.color_palette("bright", 1)
# plot
sns.scatterplot(X_embedded[:,0], X_embedded[:,1], palette=palette)
plt.title('t-SNE with no Labels')
plt.savefig("t-sne_covid19.png")
plt.show()
 可以看到, t-SNE在降低尺寸方面做得很好,但是现在我们需要一些标签,不然中心的簇类难以分辨。 让我们使用k均值发现的聚类作为标签。 这将有助于从视觉上区分主题的不同集中。
可以看到, t-SNE在降低尺寸方面做得很好,但是现在我们需要一些标签,不然中心的簇类难以分辨。 让我们使用k均值发现的聚类作为标签。 这将有助于从视觉上区分主题的不同集中。
%matplotlib inline
from matplotlib import pyplot as plt
import seaborn as sns
# sns settings
sns.set(rc={'figure.figsize':(15,15)})
# colors
palette = sns.hls_palette(20, l=.4, s=.9)
# plot
sns.scatterplot(X_embedded[:,0], X_embedded[:,1], hue=y_pred, legend='full', palette=palette)
plt.title('t-SNE with Kmeans Labels')
plt.savefig("improved_cluster_tsne.png")
plt.show()
带标签的图表可以更好地了解文章的分组方式。有趣的是,即使k-均值和t-SNE能够独立运行,它们也能够在某些集群上达成共识。通过t-SNE确定每篇文章在图中的位置,而通过k均值确定标签(颜色)。
6 每个集群中的主题模型
现在,我们将尝试在每个聚类中找到最重要的词。 K均值将文章聚类,但未标记主题。 通过主题建模,我们将发现每个集群最重要的术语是什么。 通过提供关键字以快速识别群集的主题,这将为群集增加更多含义。
对于主题建模,我们将使用LDA(潜在Dirichlet分配)。 在LDA中,每个文档都可以通过主题分布来描述,每个主题都可以通过单词分布来描述。这次采用CountVectorizer来将文本向量化。
from sklearn.decomposition import LatentDirichletAllocation
from sklearn.feature_extraction.text import CountVectorizer
## First we will create 20 vectorizers, one for each of our cluster labels
vectorizers = []
for i in range(0,20):
vectorizers.append(CountVectorizer(min_df=5,max_df=0.9,stop_words='english', lowercase=True, token_pattern='[a-zA-Z\-][a-zA-Z\-]{2,}'))
vectorized_data = []
for current_cluster, cvec in enumerate(vectorizers):
try:
vectorized_data.append(cvec.fit_transform(df.loc[df['y'] == current_cluster, 'processed_text']))
except Exception as e:
print("Not enough instances in cluster: " + str(current_cluster))
vectorized_data.append(None)主题建模将通过使用潜在狄利克雷分配(LDA)进行。 这是一个生成统计模型,可以通过一个共享的主题来解释单词集。
# number of topics per cluster
NUM_TOPICS_PER_CLUSTER = 20
lda_models = []
for ii in range(0, 20):
# Latent Dirichlet Allocation Model
lda = LatentDirichletAllocation(n_components=NUM_TOPICS_PER_CLUSTER, max_iter=10, learning_method='online',verbose=False, random_state=42)
lda_models.append(lda)
clusters_lda_data = []
for current_cluster, lda in enumerate(lda_models):
# print("Current Cluster: " + str(current_cluster))
if vectorized_data[current_cluster] != None:
clusters_lda_data.append((lda.fit_transform(vectorized_data[current_cluster])))从每个集群中提取关键字。将单个群集的关键字列表追加到长度为NUM_TOPICS_PER_CLUSTER的2D列表中。
# Functions for printing keywords for each topic
def selected_topics(model, vectorizer, top_n=3):
current_words = []
keywords = []
for idx, topic in enumerate(model.components_):
words = [(vectorizer.get_feature_names()[i], topic[i]) for i in topic.argsort()[:-top_n - 1:-1]]
for word in words:
if word[0] not in current_words:
keywords.append(word)
current_words.append(word[0])
keywords.sort(key = lambda x: x[1])
keywords.reverse()
return_values = []
for ii in keywords:
return_values.append(ii[0])
return return_values
all_keywords = []
for current_vectorizer, lda in enumerate(lda_models):
# print("Current Cluster: " + str(current_vectorizer))
if vectorized_data[current_vectorizer] != None:
all_keywords.append(selected_topics(lda, vectorizers[current_vectorizer]))
'''
all_keywords[0][:10]:
['antibody','response','target','gene','culture','autophagy','infection','viral','expression','ifitm']
'''
7 分类
尽管是任意的,但在运行kmeans之后,现在已对数据进行“标记”。 这意味着我们现在使用监督学习来了解聚类的推广程度。 这只是评估聚类的一种方法。如果k均值能够在数据中找到有意义的拆分,则应该有可能训练分类器以预测给定实例应属于哪个群集。
# function to print out classification model report
def classification_report(model_name, test, pred):
from sklearn.metrics import precision_score, recall_score
from sklearn.metrics import accuracy_score
from sklearn.metrics import f1_score
print(model_name, ":\n")
print("Accuracy Score: ", '{:,.3f}'.format(float(accuracy_score(test, pred)) * 100), "%")
print(" Precision: ", '{:,.3f}'.format(float(precision_score(test, pred, average='macro')) * 100), "%")
print(" Recall: ", '{:,.3f}'.format(float(recall_score(test, pred, average='macro')) * 100), "%")
print(" F1 score: ", '{:,.3f}'.format(float(f1_score(test, pred, average='macro')) * 100), "%")
from sklearn.model_selection import train_test_split
# test set size of 20% of the data and the random seed 42 <3
X_train, X_test, y_train, y_test = train_test_split(X.toarray(),y_pred, test_size=0.2, random_state=42)
## X_train size: 7269, X_test size: 1818
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import cross_val_predict
from sklearn.linear_model import SGDClassifier
# SGD instance
sgd_clf = SGDClassifier(max_iter=10000, tol=1e-3, random_state=42, n_jobs=4)
# train SGD
sgd_clf.fit(X_train, y_train)
# cross validation predictions
sgd_pred = cross_val_predict(sgd_clf, X_train, y_train, cv=3, n_jobs=4)
# print out the classification report
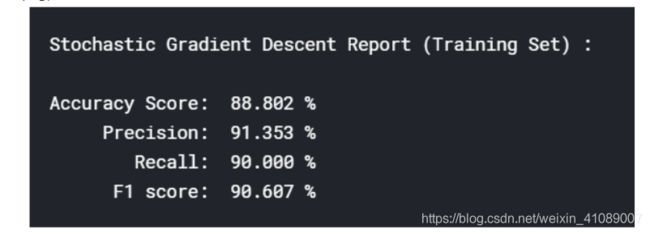
classification_report("Stochastic Gradient Descent Report (Training Set)", y_train, sgd_pred)# cross validation predictions
sgd_pred = cross_val_predict(sgd_clf, X_test, y_test, cv=3, n_jobs=4)
# print out the classification report
classification_report("Stochastic Gradient Descent Report (Training Set)", y_test, sgd_pred)
这说明我们的聚类还是比较合理的。
8 数据可视化
前面的步骤为我们提供了聚类标签和减少到二维的论文数据集。 通过将其与Bokeh配对,我们可以创建这些论文的互动交互图。Bokeh会将实际论文与其在t-SNE图上的位置配对。通过这种方法,将更容易看到论文如何组合在一起,从而可以探索数据集和评估聚类。
import os
# change into lib directory to load plot python scripts
main_path = os.getcwd()
lib_path = '/kaggle/input/kaggle-resources'
os.chdir(lib_path)
# required libraries for plot
from call_backs import input_callback, selected_code # file with customJS callbacks for bokeh
# github.com/MaksimEkin/COVID19-Literature-Clustering/blob/master/lib/call_backs.py
import bokeh
from bokeh.models import ColumnDataSource, HoverTool, LinearColorMapper, CustomJS, Slider, TapTool, TextInput
from bokeh.palettes import Category20
from bokeh.transform import linear_cmap, transform
from bokeh.io import output_file, show, output_notebook
from bokeh.plotting import figure
from bokeh.models import RadioButtonGroup, TextInput, Div, Paragraph
from bokeh.layouts import column, widgetbox, row, layout
from bokeh.layouts import column
# go back
os.chdir(main_path)启动。
# show on notebook
output_notebook()
# target labels
y_labels = y_pred
# data sources
source = ColumnDataSource(data=dict(
x= X_embedded[:,0],
y= X_embedded[:,1],
x_backup = X_embedded[:,0],
y_backup = X_embedded[:,1],
desc= y_labels,
titles= df['title'],
authors = df['authors'],
journal = df['journal'],
abstract = df['abstract_summary'],
labels = ["C-" + str(x) for x in y_labels],
links = df['doi']
))
# hover over information
hover = HoverTool(tooltips=[
("Title", "@titles{safe}"),
("Author(s)", "@authors{safe}"),
("Journal", "@journal"),
("Abstract", "@abstract{safe}"),
("Link", "@links")
],
point_policy="follow_mouse")
# map colors
mapper = linear_cmap(field_name='desc',
palette=Category20[20],
low=min(y_labels) ,high=max(y_labels))
# prepare the figure
plot = figure(plot_width=1200, plot_height=850,
tools=[hover, 'pan', 'wheel_zoom', 'box_zoom', 'reset', 'save', 'tap'],
title="Clustering of the COVID-19 Literature with t-SNE and K-Means",
toolbar_location="above")
# plot settings
plot.scatter('x', 'y', size=5,
source=source,
fill_color=mapper,
line_alpha=0.3,
line_color="black",
legend = 'labels')
plot.legend.background_fill_alpha = 0.6风格尺寸设置。
# Keywords
text_banner = Paragraph(text= 'Keywords: Slide to specific cluster to see the keywords.', height=45)
input_callback_1 = input_callback(plot, source, text_banner, topics)
# currently selected article
div_curr = Div(text="""Click on a plot to see the link to the article.""",height=150)
callback_selected = CustomJS(args=dict(source=source, current_selection=div_curr), code=selected_code())
taptool = plot.select(type=TapTool)
taptool.callback = callback_selected
# WIDGETS
slider = Slider(start=0, end=20, value=20, step=1, title="Cluster #", callback=input_callback_1)
keyword = TextInput(title="Search:", callback=input_callback_1)
# pass call back arguments
input_callback_1.args["text"] = keyword
input_callback_1.args["slider"] = slider
# STYLE
slider.sizing_mode = "stretch_width"
slider.margin=15
keyword.sizing_mode = "scale_both"
keyword.margin=15
div_curr.style={'color': '#BF0A30', 'font-family': 'Helvetica Neue, Helvetica, Arial, sans-serif;', 'font-size': '1.1em'}
div_curr.sizing_mode = "scale_both"
div_curr.margin = 20
text_banner.style={'color': '#0269A4', 'font-family': 'Helvetica Neue, Helvetica, Arial, sans-serif;', 'font-size': '1.1em'}
text_banner.sizing_mode = "scale_both"
text_banner.margin = 20
plot.sizing_mode = "scale_both"
plot.margin = 5
r = row(div_curr,text_banner)
r.sizing_mode = "stretch_width"展示。
# LAYOUT OF THE PAGE
l = layout([
[slider, keyword],
[text_banner],
[div_curr],
[plot],
])
l.sizing_mode = "scale_both"
# show
output_file('t-sne_covid-19_interactive.html')
show(l)
如何使用图解?图中的每个点代表一篇研究文章。它们用kmeans找到的簇号进行颜色编码。默认情况下,未选择任何群集,并且显示所有群集。要选择一个不同的群集,请将滑块设置为所需的群集编号。选择单个群集时,在该群集上方将显示在该群集中找到的关键字列表。您还可以在单个群集内或全部群集中按特定关键字搜索文章。使用例子就不写了,感兴趣的朋友可以自行前往原网站阅读。