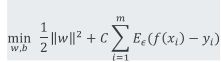机器学习--支持向量机
支持向量机(SVM)和支持向量机回归(SVR) - 知乎
机器学习工程师面试宝典-第一章-绪论 - 知乎
支持向量机基本概念
支持向量机原理
基本概念:
场景1
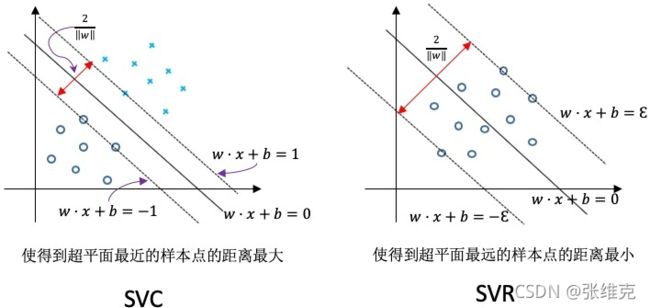
叫做 硬间隔 。
场景2
场景3
如何画出如下两个类别的超平面?
•显然,只使用之前的线性超平面是无法正确分类的
SVM通过引入额外的特征来解决以上问题。
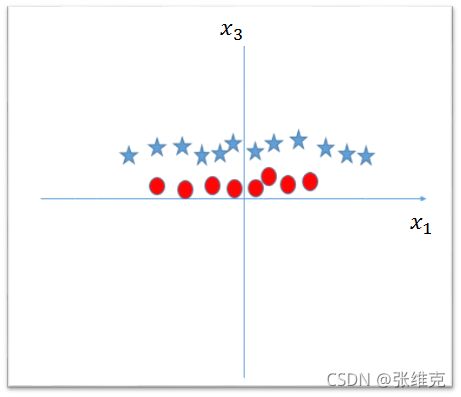
•添加一个新特征3=12+22x_3=x_1^2+x_2^2。
•绘制轴1x_1和3x_3上的数据点。
引入新特征3x_3需要注意的是:
•3x_3的所有值都是正的,因为3x_3是1x_1和2x_2的平方和。
•在原图中,红色圆圈出现在靠近1x_1和2x_2轴原点的位置,
导致3x_3值比较低。星形相对远离原点,导致3x_3值较高。
对于非线性分类问题,我们是否需要手动添加一个特征以获得超平面?
SVM有一种称为核技巧的技术。
•通过引入核函数把低维度的输入空间转换为更高维度的空间,来解决非线性分离问题
核函数
•线性核函数
•方案首选,奥卡姆剃刀原理
•简单,求解快
•可解释性强:可以轻易知道哪些feature是重要的
•多项式核
•基本原理:依靠升维使得原本线性不可分的数据线性可分
•可解决非线性问题
•对于大数量级的幂数,不太适用
•需要选择的参数较多
•高斯核
优点:
•可以映射到无限维
•决策边界更为多样
•只有一个参数,相比多项式核容易选择
•Sigmoid核
主要用在神经网络的设计中
在实战中通常只在线性核与高斯核之间做选择
•数据量特别大,或者数据特征维数很高,选择线性核
•数据量中等,特征数少,选择高斯核
三种支持向量机
在python下使用SVM
sklearn.svm.SVC(只考虑分类问题)
参数详解:
•C: float参数 默认值为1.0
•错误项的惩罚系数。C越大,即对分错样本的惩罚程度越大,因此在训练样本中准确率越高,但是泛化能力降低,也就是对测试数据的分类准确率降低。相反,减小C的话,容许训练样本中有一些误分类错误样本,泛化能力强。对于训练样本带有噪声的情况,一般采用后者,把训练样本集中错误分类的样本作为噪声。
•kernel: str参数默认为‘rbf’:高斯核函数,其他可选核函数如下:
•‘linear’:线性核函数
•‘poly’:多项式核函数
•‘sigmod’:sigmod核函数
•‘precomputed’:核矩阵
–precomputed表示自己提前计算好核函数矩阵,这时候算法内部就不再用核函数去计算核矩阵,而是直接用你给的核矩阵。
•degree: int型参数,默认为3
•这个参数只对多项式核函数有用,是指多项式核函数的阶数n。如果给的核函数参数是其他核函数,则会自动忽略该参数。
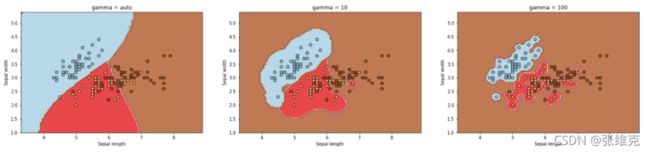
•gamma: float参数,或者{ scaler,auto},默认为scaler ,
•核函数系数,只对‘rbf’,‘poly’,‘sigmod’有效。控制这些核函数的拟合能力,gamma越大拟合能力越强,越容易过拟合。反之,拟合能力越弱,越容易欠拟合。
•如果gamma为auto,代表其值为样本特征数的倒数,即1/n_features。
•如果为scaler,gamma=1/(n_features*X.var())
•coef0:float参数,默认为0.0
•核函数中的独立项,只有对‘poly’和‘sigmod’核函数有用,是指其中的参数c。
•probability:bool参数,默认为False
•是否启用概率估计。 这必须在调用fit()之前启用,并且会使fit()方法速度变慢。
•shrinking:bool参数,默认为True
•是否采用启发式收缩方式。
•tol:float参数,默认为1e^-3
•svm停止训练的误差精度。
•cache_size:float参数,默认为200
•指定训练所需要的内存,以MB为单位,默认为200MB。
•class_weight:字典类型或者‘balance’字符串。默认为None
•给每个类别分别设置不同的惩罚参数C,如果没有给,则会给所有类别都给C=1,即前面参数指出的参数C.
•如果给定参数‘balance’,则使用y的值自动调整与输入数据中的类频率成反比的权重。
•verbose:bool参数,默认为False
•是否启用详细输出。 此设置利用libsvm中的每个进程运行时设置,如果启用,可能无法在多线程上下文中正常工作。一般情况都设为False。
•max_iter:int参数,默认为-1
•最大迭代次数,如果为-1,表示不限制。
•random_state:int型参数,默认为None
•伪随机数发生器的种子,在混洗数据时用于概率估计。
SVC的属性:
•svc.n_support_:各类各有多少个支持向量
•svc.support_:各类的支持向量在训练样本中的索引
•svc.support_vectors_:各类所有的支持向量
莺尾花实例
import numpy as np
import matplotlib.pyplot as plt
from sklearn import svm,datasetsiris = datasets.load_iris()
X=iris.data[:,:2]
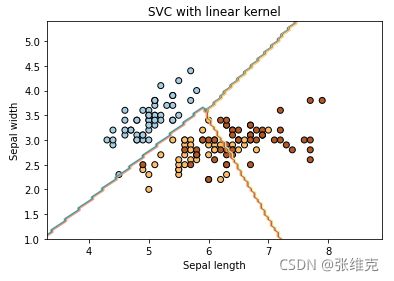
y=iris.targetsvc = svm.SVC(kernel='linear',C=1).fit(X,y)#x_min,x_max = X[:,0].min()-1,X[:,0].max()+1
y_min,y_max = X[:,1].min()-1,X[:,1].max()+1xx,yy = np.meshgrid(np.linspace(x_min,x_max,100),np.linspace(y_min,y_max,100))Z = svc.predict(np.c_[xx.ravel(), yy.ravel()]) #对应的类别值
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap=plt.cm.Paired, alpha=0.8)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Paired, edgecolors='k')
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('SVC with linear kernel')
plt.show()
Z = svc.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contour(xx, yy, Z, cmap=plt.cm.Paired, alpha=0.8)
# plt.contour(xx, yy, Z, cmap=plt.cm.hot, alpha=0.8)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Paired, edgecolors='k')
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('SVC with linear kernel')
plt.show()svc = svm.SVC(kernel='rbf',C=1).fit(X,y)Z = svc.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap=plt.cm.Paired, alpha=0.8)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Paired, edgecolors='k')
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('SVC with rbf kernel')
plt.show()
plt.figure(figsize=(25,5))
plt.subplot(1,3,1)
svc = svm.SVC(kernel ='rbf',C = 1, gamma='auto').fit(X,y)
Z = svc.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap=plt.cm.Paired, alpha=0.8)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Paired, edgecolors='k')
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('gamma = auto')
plt.subplot(1,3,2)
svc = svm.SVC(kernel ='rbf',C = 1, gamma=10).fit(X,y)
Z = svc.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap=plt.cm.Paired, alpha=0.8)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Paired, edgecolors='k')
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('gamma = 10')
plt.subplot(1,3,3)
svc = svm.SVC(kernel ='rbf',C = 1, gamma=100).fit(X,y)
Z = svc.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap=plt.cm.Paired, alpha=0.8)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Paired, edgecolors='k')
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('gamma = 100')
plt.show()
plt.figure(figsize=(25,5))
plt.subplot(1,3,1)
svc = svm.SVC(kernel ='rbf',C = 1).fit(X,y)
Z = svc.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap=plt.cm.Paired, alpha=0.8)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Paired, edgecolors='k')
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
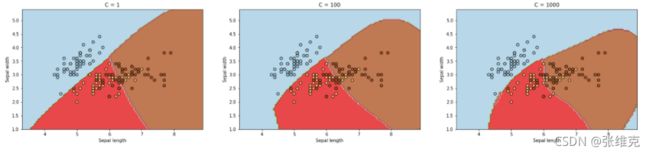
plt.title('C = 1')
plt.subplot(1,3,2)
svc = svm.SVC(kernel ='rbf',C = 10).fit(X,y)
Z = svc.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap=plt.cm.Paired, alpha=0.8)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Paired, edgecolors='k')
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('C = 100')
plt.subplot(1,3,3)
svc = svm.SVC(kernel ='rbf',C = 1000).fit(X,y)
Z = svc.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap=plt.cm.Paired, alpha=0.8)
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Paired, edgecolors='k')
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.title('C = 1000')
plt.show()支持向量机解决回归问题
SVR算法
SVR的优化问题:
SVR只计算数据点落入以f(x)为中心,±ϵ范围区域外的数据点
(虚线所围成的区域外),其中(z)可以理解为点到f(x)的ϵ区域距离
类似软间隔分类时引入的松弛因子。这里引入两个松弛因子ξ和ξ ̂,目的是尽可能让红色区域拟合数据又不容易受异常值影响。
利用一条固定宽度的条带,覆盖尽可能多的样本点,从而使得总误差尽可能的小。
引入两个松弛因子ξ和ξ ̂,表示对ϵ区域两边进行松弛,目的是尽可能让红色区域拟合数据又不容易受异常值影响。
利用一条固定宽度的条带,覆盖尽可能多的样本点,从而使得总误差尽可能的小。
SVR优化问题可以重写为:
SVM的优缺点
优点
缺点:
拥有大量的数据集时,SVM表现并不好,因为计算量很大,所需要的训练时间长
数据集的噪音过多时,表现不好,需要非常仔细的数据预处理和调参
核函数选择难度较大