- element实现动态路由+面包屑
软件技术NINI
vue案例vue.js前端
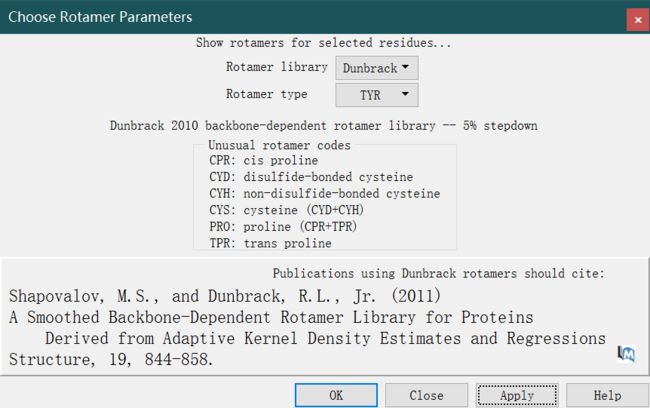
el-breadcrumb是ElementUI组件库中的一个面包屑导航组件,它用于显示当前页面的路径,帮助用户快速理解和导航到应用的各个部分。在Vue.js项目中,如果你已经安装了ElementUI,就可以很方便地使用el-breadcrumb组件。以下是一个基本的使用示例:安装ElementUI(如果你还没有安装的话):你可以通过npm或yarn来安装ElementUI。bash复制代码npmi
- Python中os.environ基本介绍及使用方法
鹤冲天Pro
#Pythonpython服务器开发语言
文章目录python中os.environos.environ简介os.environ进行环境变量的增删改查python中os.environ的使用详解1.简介2.key字段详解2.1常见key字段3.os.environ.get()用法4.环境变量的增删改查和判断是否存在4.1新增环境变量4.2更新环境变量4.3获取环境变量4.4删除环境变量4.5判断环境变量是否存在python中os.envi
- linux中sdl的使用教程,sdl使用入门
Melissa Corvinus
linux中sdl的使用教程
本文通过一个简单示例讲解SDL的基本使用流程。示例中展示一个窗口,窗口里面有个随机颜色快随机移动。当我们鼠标点击关闭按钮时间窗口关闭。基本步骤如下:1.初始化SDL并创建一个窗口。SDL_Init()初始化SDL_CreateWindow()创建窗口2.纹理渲染存储RGB和存储纹理的区别:比如一个从左到右由红色渐变到蓝色的矩形,用存储RGB的话就需要把矩形中每个点的具体颜色值存储下来;而纹理只是一
- 直返最高等级与直返APP:无需邀请码的返利新体验
古楼
随着互联网的普及和电商的兴起,直返模式逐渐成为一种流行的商业模式。在这种模式下,消费者通过购买产品或服务,获得一定的返利,并可以分享给更多的人。其中,直返最高等级和直返APP是直返模式中的重要概念和工具。本文将详细介绍直返最高等级的概念、直返APP的使用以及与邀请码的关系。【高省】APP(高佣金领导者)是一个自用省钱佣金高,分享推广赚钱多的平台,百度有几百万篇报道,运行三年,稳定可靠。高省APP,
- 2020-04-12每天三百字之连接与替代
冷眼看潮
不知道是不是好为人师,有时候还真想和别人分享一下我对某些现象的看法或者解释。人类社会不断发展进步的过程,就是不断连接与替代的过程。人类发现了火并应用火以后,告别了茹毛饮血的野兽般的原始生活(火烧、烹饪替代了生食)人类用石器代替了完全手工,工具的使用使人类进步一大步。类似这样的替代还有很多,随着科技的发展,有更多的原始的事物被替代,代之以更高效、更先进的技术。在近现代,汽车替代了马车,高速公路和铁路
- pyecharts——绘制柱形图折线图
2224070247
信息可视化pythonjava数据可视化
一、pyecharts概述自2013年6月百度EFE(ExcellentFrontEnd)数据可视化团队研发的ECharts1.0发布到GitHub网站以来,ECharts一直备受业界权威的关注并获得广泛好评,成为目前成熟且流行的数据可视化图表工具,被应用到诸多数据可视化的开发领域。Python作为数据分析领域最受欢迎的语言,也加入ECharts的使用行列,并研发出方便Python开发者使用的数据
- Python爬虫解析工具之xpath使用详解
eqa11
python爬虫开发语言
文章目录Python爬虫解析工具之xpath使用详解一、引言二、环境准备1、插件安装2、依赖库安装三、xpath语法详解1、路径表达式2、通配符3、谓语4、常用函数四、xpath在Python代码中的使用1、文档树的创建2、使用xpath表达式3、获取元素内容和属性五、总结Python爬虫解析工具之xpath使用详解一、引言在Python爬虫开发中,数据提取是一个至关重要的环节。xpath作为一门
- 2.2.6 通知类控件 Toast、Menu
常思行
本文例程下载:WillFlow_Toast、WillFlowMenu一、什么是Toast?Toast也被叫做吐司,是Android系统提供的一种非常好的提醒方式,在程序中可以使用它将一些短小的信息通知给用户,它有如下两个特点:Toast是没有焦点的Toast显示的时间有限过一定的时间就会自动消失所以一般来讲Toast的使用并不会影响我们的正常操作,并且它通常不会占用太大的屏幕空间,有着良好的用户体
- 第二十 python基础--语句
九樱MOL
目录具体内容1:if语句的使用格式判断语句2:if-else的使用格式3:if-elif-else的使用格式4:if嵌套1:while循环的格式循环语句2:while循环嵌套3:for循环的格式一、判断语句在程序中如果某些条件满足,才能做某件事情,而不满足时不允许做,这就是所谓的判断1.1if语句的使用格式if要判断的条件:条件成立时,要做的事情案例:判断年纪,如果age大于18,输入成年age=
- Python神器!WEB自动化测试集成工具 DrissionPage
亚丁号
python开发语言
一、前言用requests做数据采集面对要登录的网站时,要分析数据包、JS源码,构造复杂的请求,往往还要应付验证码、JS混淆、签名参数等反爬手段,门槛较高。若数据是由JS计算生成的,还须重现计算过程,体验不好,开发效率不高。使用浏览器,可以很大程度上绕过这些坑,但浏览器运行效率不高。因此,这个库设计初衷,是将它们合而为一,能够在不同须要时切换相应模式,并提供一种人性化的使用方法,提高开发和运行效率
- 如何用matlab灵活控制feko的求解
NingrLi
matlab开发语言
https://bbs.rfeda.cn/read.php?tid=3778Feko中的模型和求解设置等都可以通过editfeko进行设置,其文件存储为.pre文件,该文件可以用文本打开,因此,我们可以通过VB、VC、matlab等工具对.pre文件进行读写操作,以达到更灵活的使用feko。同样,对于.out文件,我们也可以进行读操作。熟练使用对.pre文件和.out文件的操作后,我们可以方便的计
- 怎么做才能真正限制塑料袋的使用?
BalNews
Environmentalpollutionisalwaysamajorlivelihoodissue.Morethanadecadeago,ourgovernmenthadintroducedapolicyaboutrestrictionsontheuseofplasticbags,wecallitrestrictionsontheuseofplasticbags.Butmorethan10ye
- 入门MySQL——查询语法练习
K_un
前言:前面几篇文章为大家介绍了DML以及DDL语句的使用方法,本篇文章将主要讲述常用的查询语法。其实MySQL官网给出了多个示例数据库供大家实用查询,下面我们以最常用的员工示例数据库为准,详细介绍各自常用的查询语法。1.员工示例数据库导入官方文档员工示例数据库介绍及下载链接:https://dev.mysql.com/doc/employee/en/employees-installation.h
- 4款毕业论文参考文献格式生成器(附加详细步骤)
小猪包333
写论文人工智能深度学习计算机视觉AI写作
在撰写毕业论文时,参考文献的格式规范是至关重要的。为了帮助学生和学者们更高效地生成符合要求的参考文献格式,本文将详细介绍四款推荐的参考文献格式生成器,并提供详细的使用步骤。1.千笔-AIPassPaper千笔-AIPassPaper是一款先进的AI辅助论文写作工具,不仅能够自动生成大纲、开题报告,还能一键生成参考文献。AI论文,免费大纲,10分钟3万字https://www.aipaperpass
- 字节二面
Redstone Monstrosity
前端面试
1.假设你是正在面试前端开发工程师的候选人,面试官让你详细说出你上一段实习过程的收获和感悟。在上一段实习过程中,我获得了宝贵的实践经验和深刻的行业洞察,以下是我的主要收获和感悟:一、专业技能提升框架应用熟练度:通过实际项目,我深入掌握了React、Vue等前端框架的使用,不仅提升了编码效率,还学会了如何根据项目需求选择合适的框架。问题解决能力:在实习期间,我遇到了许多预料之外的技术难题。通过查阅文
- 史上最全git命令,git回滚,git命令大全
騒周
其他git
git命令大全一、Git整体理解二、由暂存区本地仓库三、由本地仓->远程仓库四、冲突处理五、Git分支操作六、bug的分支七、feature分支八、暂存的使用九、远程仓的操作十、标签的使用十一、Git配置全局信息十二、Linux的一些简单操作和一些符号的解释十三、符号解释十四、显示安装详细信息十五、gitconfig十六、Gitclone十七、Gitinit十八、gitstatus十九、gitre
- Nginx的使用场景:构建高效、可扩展的Web架构
张某布响丸辣
nginx前端架构
Nginx,作为当今最流行的Web服务器和反向代理软件之一,凭借其高性能、稳定性和灵活性,在众多Web项目中扮演着核心角色。无论是个人博客、中小型网站,还是大型企业级应用,Nginx都能提供强大的支持。本文将探讨Nginx的几个主要使用场景,帮助读者理解如何在实际项目中充分利用Nginx的优势。1.静态文件服务对于包含大量静态文件(如HTML、CSS、JavaScript、图片等)的网站,Ngin
- 什么是最好的时间管理
江千钰
源自罗辑思维1.最近我看到一个观点,说时间管理这个事,一般方法的入手点,可能都错了。为啥?因为都是想站在自己和时间的外面,规范对时间的使用。这管理得越狠,就越是分秒必争,那样就会让自己的生活越绷越紧,最后谁都受不了。2.那真正的时间管理的秘诀是什么呢?其实秘诀就是四个字。头两个字是“沉浸”。沉浸在自己做的事里面。你可能会说,不对啊,我就是沉浸在刷手机、打游戏里面,所以才浪费时间,才需要时间管理嘛。
- svg图片兼容性和用法优缺点
独行侠_ef93
svg图片的使用方法第一次来认认真真的研究了下svg图片,之前只是在网上见过,但都是一晃而过也没当回事,最近网站改版看到同事有用到svg格式的图片,想想自己干了几年的重构也没用过,这些细节的知识是应该好好研究研究了。暂时还没研究得完全透切,先记下目前为止所看到的吧不然又给忘了。svg可缩放矢量图形(ScalableVectorGraphics),顾名思义就是任意改变其大小也不会变形,是基于可扩展标
- 基于STM32F103C8T6定时器的PWM通道的重映射
—你的鼬先生
stm32嵌入式硬件单片机
在我们平时的的使用中,我们最常使用的是TIM2和TIM3的PWM通道,但是由于C8T6的IO口有限,所以可能会出现PWM通道的资源不够的情况,从而我们可能会使用PWM4的PWM通道,但是TIM4的PWM通道并不能直接使用,它需要进行一个重映射,不然可能会导致PWM波不能正常发送。以下就是对PWM4的PWM通道进行一个重映射#include"stm32f10x.h"//Deviceheadervoi
- Lombok:Java开发者的代码简化神器【后端 17】
终末圆
Java后端java开发语言mysql数据库后端springbootpython
Lombok:Java开发者的代码简化神器在Java开发中,我们经常需要编写大量的样板代码,如getter、setter、equals、hashCode、toString等方法。这些代码虽然基础且必要,但往往占据了大量开发时间,且容易在属性变更时引发错误。幸运的是,Lombok这个Java库通过注解的方式,为我们提供了一种高效的解决方案。本文将详细介绍Lombok的使用及其优势。什么是Lombok
- 深入理解AOP(面向切面编程)及其应用
自身就是太阳
java开发语言spring
目录AOP的核心概念AOP的实现方式1.定义DAO接口和实现类2.定义通知类3.开启AOP注解驱动切入点表达式通配符的使用:AOP通知类型案例分析:测量业务层接口的执行效率结论概述:AOP(Aspect-OrientedProgramming,面向切面编程)是一种编程范式,主要用于将共性功能从具体的业务逻辑中分离出来,实现松耦合的代码设计。其作用是在不修改原始代码的情况下,对现有方法进行增强,广泛
- Vector和Stack的用法
蟹道人
JavaSejava
/***作者:*日期:*功能:vector的用法*/packagecom.cg;importjava.util.*;publicclassDemo5{publicstaticvoidmain(String[]args){//Vector的使用Vectorvec=newVector();Empemp=newEmp("2011",25,"zhang");vec.add(emp);for(inti=0;
- 如何使用并提高批判性思考能力?
颍水书生
学会一个技能的最好办法就是不断的使用,学英语如此、学游泳如此、学吉他也是如此。作人类最核心的技能,学会思考的最重要的方法不是学,而是不断的对心智的积极运用。如何使用心智,则是一个相对不容易的事情,因为思考结果的没有明显的反馈,不像游泳,换气、泳姿、游的速度这些都是可以量化,水平的高低,很容易考评,甚至一眼就能看出来。不过不容易也未必是无章可循,就像我们在读书时那样,可以先来个摸底考试,看看自己水平
- 9.15初识指针
西科Monesy
c语言开发语言
初识指针什么是指针?指针是一种数据类型,它存储了变量的内存地址。通过指针,程序可以直接访问和操作内存中的数据,而不是通过变量的名称。这使得C语言在内存管理和性能优化方面具有很大的灵活性。内存是什么?内存是电脑上的存储器,计算机中程序的运行都是在内存中进行的。程序中如果有数据需要存储也会申请内存空间。为了有效的使用内存,就把内存划分成一个小小的内存单元,每个内存单元的大小是一个字节。为了能够有效的访
- 深入探讨:如何在Python中通过LangChain技术精准追踪大型语言模型(LLM)的Token使用情况
m0_57781768
pythonlangchain语言模型
深入探讨:如何在Python中通过LangChain技术精准追踪大型语言模型(LLM)的Token使用情况在现代的人工智能开发中,大型语言模型(LLM)已经成为了不可或缺的工具,无论是用于自然语言处理、对话生成,还是其他复杂的文本生成任务。然而,随着这些模型的广泛应用,开发者面临的一个重要挑战是如何有效地追踪和管理Token的使用情况,特别是在生产环境中,Token的使用直接影响着API调用的成本
- Java中四种常用的数组复制的方法copyOf(),arraycop(),clone()和copyOfRange()的使用与区别
方九九
java知识点总结java
所谓复制数组,是指将一个数组中的元素在另一个数组中进行复制。本文主要介绍关于Java里面的数组复制(拷贝)的几种方式和用法。在Java中实现数组复制分别有以下4种方法:1.Arrays类的copyOf()方法2.Arrays类的copyOfRange()方法3.System类的arraycopy()方法4.Object类的clone()方法下面来详细介绍这4种方法的使用。使用copyOf()方法和
- 年底了,积分也要清零了,该兑就兑了吧,清零就可惜了
遇见yh
年底了,许多平台的积分也要清零了,信用卡、电话卡的积分也攒了不少,该兑就得兑了,如果不兑,到年底也都会被清零。所以如果你的使用的平台,现在还有积分的话,就赶快去兑换一下,如果清零就太可惜了。前几天我登录联通官网一看,上面也有1000多个积分,就去兑换了一件麦芯粉。还有支付宝积分,也可以兑换自己需要的东西,我昨天就兑了一盒脱骨鸡爪。之前还兑过一箱全麦面包,品质都不错。这些东西都是平台送的,你不去兑换
- 笔记:Python顺序结构 练习题
辞言i
笔记python
文章目录前言一、什么是顺序结构?二、练习题1.题目2.填空题3.简答题4.编程题总结前言本次笔记旨在帮助读者加深对Python编程语言中顺序结构和选择题的理解。在学习Python编程过程中,掌握程序的基本结构以及条件语句的使用至关重要。通过本次练习题,读者将有机会测试自己对这些概念的掌握程度,并通过实际应用进一步巩固所学知识。本次练习题将涵盖Python中的顺序结构的练习题,旨在帮助读者提高解决问
- 怎样使用淘宝优惠券?全面解析优惠券使用技巧
氧惠_飞智666999
在数字化的购物时代,淘宝优惠券已经成为我们享受购物优惠的重要途径。然而,许多消费者在领取了优惠券后,却不知如何正确使用,导致优惠券的价值无法得到充分发挥。那么,怎样使用淘宝优惠券呢?本文将为您全面解析优惠券的使用技巧,助您轻松享受购物优惠。首先,我们需要了解优惠券的来源。淘宝优惠券可以通过多种途径获取,如淘宝官方活动页面、店铺首页、品牌官方账号等。同时,一些第三方平台也会提供淘宝优惠券的领取服务。
- ios内付费
374016526
ios内付费
近年来写了很多IOS的程序,内付费也用到不少,使用IOS的内付费实现起来比较麻烦,这里我写了一个简单的内付费包,希望对大家有帮助。
具体使用如下:
这里的sender其实就是调用者,这里主要是为了回调使用。
[KuroStoreApi kuroStoreProductId:@"产品ID" storeSender:self storeFinishCallBa
- 20 款优秀的 Linux 终端仿真器
brotherlamp
linuxlinux视频linux资料linux自学linux教程
终端仿真器是一款用其它显示架构重现可视终端的计算机程序。换句话说就是终端仿真器能使哑终端看似像一台连接上了服务器的客户机。终端仿真器允许最终用户用文本用户界面和命令行来访问控制台和应用程序。(LCTT 译注:终端仿真器原意指对大型机-哑终端方式的模拟,不过在当今的 Linux 环境中,常指通过远程或本地方式连接的伪终端,俗称“终端”。)
你能从开源世界中找到大量的终端仿真器,它们
- Solr Deep Paging(solr 深分页)
eksliang
solr深分页solr分页性能问题
转载请出自出处:http://eksliang.iteye.com/blog/2148370
作者:eksliang(ickes) blg:http://eksliang.iteye.com/ 概述
长期以来,我们一直有一个深分页问题。如果直接跳到很靠后的页数,查询速度会比较慢。这是因为Solr的需要为查询从开始遍历所有数据。直到Solr的4.7这个问题一直没有一个很好的解决方案。直到solr
- 数据库面试题
18289753290
面试题 数据库
1.union ,union all
网络搜索出的最佳答案:
union和union all的区别是,union会自动压缩多个结果集合中的重复结果,而union all则将所有的结果全部显示出来,不管是不是重复。
Union:对两个结果集进行并集操作,不包括重复行,同时进行默认规则的排序;
Union All:对两个结果集进行并集操作,包括重复行,不进行排序;
2.索引有哪些分类?作用是
- Android TV屏幕适配
酷的飞上天空
android
先说下现在市面上TV分辨率的大概情况
两种分辨率为主
1.720标清,分辨率为1280x720.
屏幕尺寸以32寸为主,部分电视为42寸
2.1080p全高清,分辨率为1920x1080
屏幕尺寸以42寸为主,此分辨率电视屏幕从32寸到50寸都有
适配遇到问题,已1080p尺寸为例:
分辨率固定不变,屏幕尺寸变化较大。
如:效果图尺寸为1920x1080,如果使用d
- Timer定时器与ActionListener联合应用
永夜-极光
java
功能:在控制台每秒输出一次
代码:
package Main;
import javax.swing.Timer;
import java.awt.event.*;
public class T {
private static int count = 0;
public static void main(String[] args){
- Ubuntu14.04系统Tab键不能自动补全问题解决
随便小屋
Ubuntu 14.04
Unbuntu 14.4安装之后就在终端中使用Tab键不能自动补全,解决办法如下:
1、利用vi编辑器打开/etc/bash.bashrc文件(需要root权限)
sudo vi /etc/bash.bashrc
接下来会提示输入密码
2、找到文件中的下列代码
#enable bash completion in interactive shells
#if
- 学会人际关系三招 轻松走职场
aijuans
职场
要想成功,仅有专业能力是不够的,处理好与老板、同事及下属的人际关系也是门大学问。如何才能在职场如鱼得水、游刃有余呢?在此,教您简单实用的三个窍门。
第一,多汇报
最近,管理学又提出了一个新名词“追随力”。它告诉我们,做下属最关键的就是要多请示汇报,让上司随时了解你的工作进度,有了新想法也要及时建议。不知不觉,你就有了“追随力”,上司会越来越了解和信任你。
第二,勤沟通
团队的力
- 《O2O:移动互联网时代的商业革命》读书笔记
aoyouzi
读书笔记
移动互联网的未来:碎片化内容+碎片化渠道=各式精准、互动的新型社会化营销。
O2O:Online to OffLine 线上线下活动
O2O就是在移动互联网时代,生活消费领域通过线上和线下互动的一种新型商业模式。
手机二维码本质:O2O商务行为从线下现实世界到线上虚拟世界的入口。
线上虚拟世界创造的本意是打破信息鸿沟,让不同地域、不同需求的人
- js实现图片随鼠标滚动的效果
百合不是茶
JavaScript滚动属性的获取图片滚动属性获取页面加载
1,获取样式属性值
top 与顶部的距离
left 与左边的距离
right 与右边的距离
bottom 与下边的距离
zIndex 层叠层次
例子:获取左边的宽度,当css写在body标签中时
<div id="adver" style="position:absolute;top:50px;left:1000p
- ajax同步异步参数async
bijian1013
jqueryAjaxasync
开发项目开发过程中,需要将ajax的返回值赋到全局变量中,然后在该页面其他地方引用,因为ajax异步的原因一直无法成功,需将async:false,使其变成同步的。
格式:
$.ajax({ type: 'POST', ur
- Webx3框架(1)
Bill_chen
eclipsespringmaven框架ibatis
Webx是淘宝开发的一套Web开发框架,Webx3是其第三个升级版本;采用Eclipse的开发环境,现在支持java开发;
采用turbine原型的MVC框架,扩展了Spring容器,利用Maven进行项目的构建管理,灵活的ibatis持久层支持,总的来说,还是一套很不错的Web框架。
Webx3遵循turbine风格,velocity的模板被分为layout/screen/control三部
- 【MongoDB学习笔记五】MongoDB概述
bit1129
mongodb
MongoDB是面向文档的NoSQL数据库,尽量业界还对MongoDB存在一些质疑的声音,比如性能尤其是查询性能、数据一致性的支持没有想象的那么好,但是MongoDB用户群确实已经够多。MongoDB的亮点不在于它的性能,而是它处理非结构化数据的能力以及内置对分布式的支持(复制、分片达到的高可用、高可伸缩),同时它提供的近似于SQL的查询能力,也是在做NoSQL技术选型时,考虑的一个重要因素。Mo
- spring/hibernate/struts2常见异常总结
白糖_
Hibernate
Spring
①ClassNotFoundException: org.aspectj.weaver.reflect.ReflectionWorld$ReflectionWorldException
缺少aspectjweaver.jar,该jar包常用于spring aop中
②java.lang.ClassNotFoundException: org.sprin
- jquery easyui表单重置(reset)扩展思路
bozch
formjquery easyuireset
在jquery easyui表单中 尚未提供表单重置的功能,这就需要自己对其进行扩展。
扩展的时候要考虑的控件有:
combo,combobox,combogrid,combotree,datebox,datetimebox
需要对其添加reset方法,reset方法就是把初始化的值赋值给当前的组件,这就需要在组件的初始化时将值保存下来。
在所有的reset方法添加完毕之后,就需要对fo
- 编程之美-烙饼排序
bylijinnan
编程之美
package beautyOfCoding;
import java.util.Arrays;
/*
*《编程之美》的思路是:搜索+剪枝。有点像是写下棋程序:当前情况下,把所有可能的下一步都做一遍;在这每一遍操作里面,计算出如果按这一步走的话,能不能赢(得出最优结果)。
*《编程之美》上代码有很多错误,且每个变量的含义令人费解。因此我按我的理解写了以下代码:
*/
- Struts1.X 源码分析之ActionForm赋值原理
chenbowen00
struts
struts1在处理请求参数之前,首先会根据配置文件action节点的name属性创建对应的ActionForm。如果配置了name属性,却找不到对应的ActionForm类也不会报错,只是不会处理本次请求的请求参数。
如果找到了对应的ActionForm类,则先判断是否已经存在ActionForm的实例,如果不存在则创建实例,并将其存放在对应的作用域中。作用域由配置文件action节点的s
- [空天防御与经济]在获得充足的外部资源之前,太空投资需有限度
comsci
资源
这里有一个常识性的问题:
地球的资源,人类的资金是有限的,而太空是无限的.....
就算全人类联合起来,要在太空中修建大型空间站,也不一定能够成功,因为资源和资金,技术有客观的限制....
&
- ORACLE临时表—ON COMMIT PRESERVE ROWS
daizj
oracle临时表
ORACLE临时表 转
临时表:像普通表一样,有结构,但是对数据的管理上不一样,临时表存储事务或会话的中间结果集,临时表中保存的数据只对当前
会话可见,所有会话都看不到其他会话的数据,即使其他会话提交了,也看不到。临时表不存在并发行为,因为他们对于当前会话都是独立的。
创建临时表时,ORACLE只创建了表的结构(在数据字典中定义),并没有初始化内存空间,当某一会话使用临时表时,ORALCE会
- 基于Nginx XSendfile+SpringMVC进行文件下载
denger
应用服务器Webnginx网络应用lighttpd
在平常我们实现文件下载通常是通过普通 read-write方式,如下代码所示。
@RequestMapping("/courseware/{id}")
public void download(@PathVariable("id") String courseID, HttpServletResp
- scanf接受char类型的字符
dcj3sjt126com
c
/*
2013年3月11日22:35:54
目的:学习char只接受一个字符
*/
# include <stdio.h>
int main(void)
{
int i;
char ch;
scanf("%d", &i);
printf("i = %d\n", i);
scanf("%
- 学编程的价值
dcj3sjt126com
编程
发一个人会编程, 想想以后可以教儿女, 是多么美好的事啊, 不管儿女将来从事什么样的职业, 教一教, 对他思维的开拓大有帮助
像这位朋友学习:
http://blog.sina.com.cn/s/articlelist_2584320772_0_1.html
VirtualGS教程 (By @林泰前): 几十年的老程序员,资深的
- 二维数组(矩阵)对角线输出
飞天奔月
二维数组
今天在BBS里面看到这样的面试题目,
1,二维数组(N*N),沿对角线方向,从右上角打印到左下角如N=4: 4*4二维数组
{ 1 2 3 4 }
{ 5 6 7 8 }
{ 9 10 11 12 }
{13 14 15 16 }
打印顺序
4
3 8
2 7 12
1 6 11 16
5 10 15
9 14
13
要
- Ehcache(08)——可阻塞的Cache——BlockingCache
234390216
并发ehcacheBlockingCache阻塞
可阻塞的Cache—BlockingCache
在上一节我们提到了显示使用Ehcache锁的问题,其实我们还可以隐式的来使用Ehcache的锁,那就是通过BlockingCache。BlockingCache是Ehcache的一个封装类,可以让我们对Ehcache进行并发操作。其内部的锁机制是使用的net.
- mysqldiff对数据库间进行差异比较
jackyrong
mysqld
mysqldiff该工具是官方mysql-utilities工具集的一个脚本,可以用来对比不同数据库之间的表结构,或者同个数据库间的表结构
如果在windows下,直接下载mysql-utilities安装就可以了,然后运行后,会跑到命令行下:
1) 基本用法
mysqldiff --server1=admin:12345
- spring data jpa 方法中可用的关键字
lawrence.li
javaspring
spring data jpa 支持以方法名进行查询/删除/统计。
查询的关键字为find
删除的关键字为delete/remove (>=1.7.x)
统计的关键字为count (>=1.7.x)
修改需要使用@Modifying注解
@Modifying
@Query("update User u set u.firstna
- Spring的ModelAndView类
nicegege
spring
项目中controller的方法跳转的到ModelAndView类,一直很好奇spring怎么实现的?
/*
* Copyright 2002-2010 the original author or authors.
*
* Licensed under the Apache License, Version 2.0 (the "License");
* yo
- 搭建 CentOS 6 服务器(13) - rsync、Amanda
rensanning
centos
(一)rsync
Server端
# yum install rsync
# vi /etc/xinetd.d/rsync
service rsync
{
disable = no
flags = IPv6
socket_type = stream
wait
- Learn Nodejs 02
toknowme
nodejs
(1)npm是什么
npm is the package manager for node
官方网站:https://www.npmjs.com/
npm上有很多优秀的nodejs包,来解决常见的一些问题,比如用node-mysql,就可以方便通过nodejs链接到mysql,进行数据库的操作
在开发过程往往会需要用到其他的包,使用npm就可以下载这些包来供程序调用
&nb
- Spring MVC 拦截器
xp9802
spring mvc
Controller层的拦截器继承于HandlerInterceptorAdapter
HandlerInterceptorAdapter.java 1 public abstract class HandlerInterceptorAdapter implements HandlerIntercep