思维导图
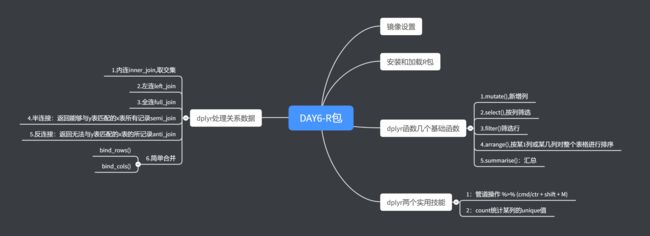
镜像设置
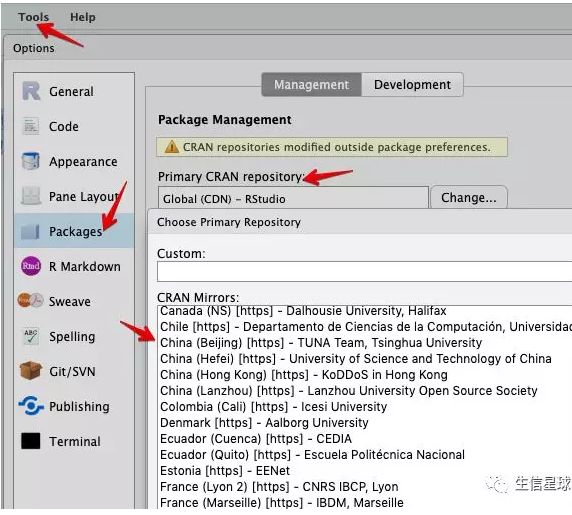
第一种方法:
第二种方法(可以免去下次打开的镜像设置):
file.edit('~/.Rprofile')调出上面的框
.Rprofile就是一个代码文件,如果启动时找到这个文件,那么就替我们先运行一遍(这个过程就是在启动Rstudio时完成的)
在代码文件中输入options函数
# options函数就是设置R运行过程中的一些选项设置
options("repos" = c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/")) #对应清华源
options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/") #对应中科大源
# 当然可以换成其他地区的镜像
最后保存=》重启Rstudio,这时你再运行一下:options()$repos和options()$BioC_mirror就发现已经配置好了
安装和加载R包
安装:install.packages(“包”)或者BiocManager::install(“包”)取决于包是存放在CRAN还是Biocductor网站
加载:library(包)或require(包)在使用包之前都要加载
安装加载代码:
options("repos" = c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")
install.packages("dplyr")
library(dplyr)
dplyr函数几个基础函数
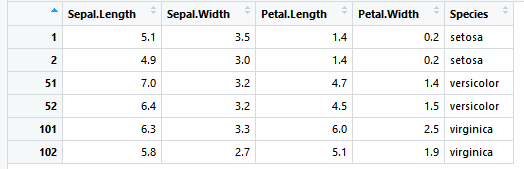
示例数据使用内置数据集:test <- iris[c(1:2,51:52,101:102),]
1.mutate(),新增列mutate(test, new = Sepal.Length * Sepal.Width)
2.select(),按列筛选
(1)按列号筛选
select(test,1) select(test,c(1,5)) select(test,Sepal.Length)
(2)按列名筛选
select(test, Petal.Length, Petal.Width)
vars <- c("Petal.Length", "Petal.Width")
select(test, one_of(vars))
3.filter()筛选行
filter(test, Species == "setosa")
filter(test, Species == "setosa"&Sepal.Length > 5 )
filter(test, Species %in% c("setosa","versicolor"))
4.arrange(),按某1列或某几列对整个表格进行排序
arrange(test, Sepal.Length)#默认从小到大排序
arrange(test, desc(Sepal.Length))#用desc从大到小
5.summarise():汇总
(对数据进行汇总操作,结合group_by使用实用性强)
summarise(test, mean(Sepal.Length), sd(Sepal.Length))# 计算Sepal.Length的平均值和标准差
group_by(test, Species)# 先按照Species分组,再计算每组Sepal.Length的平均值和标准差
summarise(group_by(test, Species),mean(Sepal.Length), sd(Sepal.Length))
dplyr两个实用技能
1:管道操作 %>% (cmd/ctr + shift + M)
test %>%
group_by(Species) %>%
summarise(mean(Sepal.Length), sd(Sepal.Length))
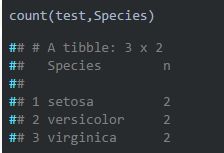
2:count统计某列的unique值
count(test,Species)
dplyr处理关系数据 (即将2个表进行连接,注意:不要引入factor)
options(stringsAsFactors = F)
test1 <- data.frame(x = c('b','e','f','x'),
z = c("A","B","C",'D'),
stringsAsFactors = F)
test1
test2 <- data.frame(x = c('a','b','c','d','e','f'),
y = c(1,2,3,4,5,6),
stringsAsFactors = F)
test2
1.內连inner_join,取交集
inner_join(test1, test2, by = "x")
2.左连left_join
left_join(test1, test2, by = 'x')
left_join(test2, test1, by = 'x')
3.全连full_join
full_join( test1, test2, by = 'x')
4.半连接:返回能够与y表匹配的x表所有记录semi_join
semi_join(x = test1, y = test2, by = 'x')
5.反连接:返回无法与y表匹配的x表的所记录anti_join
anti_join(x = test2, y = test1, by = 'x')
6.简单合并
在相当于base包里的cbind()函数和rbind()函数;注意,bind_rows()函数需要两个表格列数相同,而bind_cols()函数则需要两个数据框有相同的行数
test1 <- data.frame(x = c(1,2,3,4), y = c(10,20,30,40))
test1
test2 <- data.frame(x = c(5,6), y = c(50,60))
test2
test3 <- data.frame(z = c(100,200,300,400))
test3
bind_rows(test1, test2)
bind_cols(test1, test3)