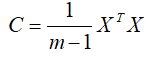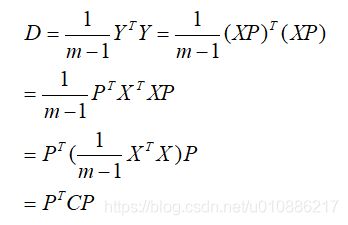机器学习(4):PCA主成分分析法实例
一、简介
1.Principal Component Analysis
2.用途:降维中最常用的一种手段,可用于数据压缩、提取重要信息等领域。
3.目标:基于方差提取最有价值的信息
二、PCA求解原理
1.优化目标
(1)第一个目标:将一组N维向量降为K维(K大于0,小于N),目标是选择K个单位(模为1)正交基,使原始数据变换到这组基上后,各字段两两间协方差为0
(2)第二个目标:每个字段的方差,则尽可能大(在正交的约束下,取最大的K个方差)
2.优化目标和协方差矩阵关系
(1)推倒原始矩阵与基变换矩阵后矩阵的关系。
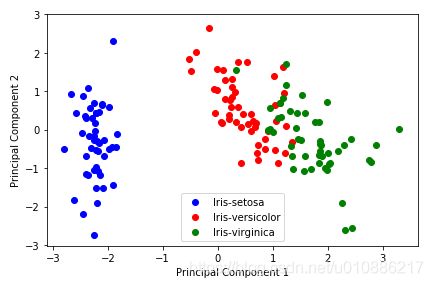
假设原始X=(X1,X2,...,Xn)有N个特征,每个特征有m条数据,即m*n的矩阵。目标将为K(k 即:Y=XP (2)推倒原始矩阵的协方差矩阵C与基变换矩阵后矩阵的协方差矩阵D的关系。 设原始矩阵的协方差矩阵维C,变换基维P,则Y=PX。其中C为 通过基变换降维后的协方差D为 (3)由协方差矩阵推出的优化目标: 寻找一个矩阵P,满足PTCP是一个对角矩阵,并且对角元素按从大到小依次排列,那么P的前K列就是要寻找的基,用X乘以P的前K列组成的矩阵就使得X从N维降到了K维并满足上述优化条件。 (1)协方差矩阵C是一个实对称矩阵,实对称矩阵有性质: (a)实对称矩阵不同特征值对应的特征向量必然正交。 (b)设特征向量λλ重数为r,则必然存在r个线性无关的特征向量对应于λλ,因此可以将这r个特征向量单位正交化 (2)所以针对实对称矩阵C,一定可以找到n个单位正交特征向量,设这n个特征向量为e1,e2,e3,..,en,组成矩阵: P=(e1 e2 e3 ... en),这个P可以使C对角化: 假设有m条n维数据 1.将原始数据按列组成m行n列矩阵X 2.将X的每一列(代表一个属性字段)进行零均值化,即减去这一行的均值 3.求出协方差矩阵 4.求出协方差矩阵的特征值及对应的特征向量 5.将特征向量按对应特征值大小从上到下按行排列成矩阵,取前k列组成矩阵P 6.Y=XP即为降维到k维后的数据(m×k) 实例采用常用的iris.data数据集 结果: (1)方法一:按照定义 (2)方法二:numpy求取协方差的函数cov 注意:输入一定要是n*m,n为特征个数,m为样本数目。即cov输入需要为: (1)降维后矩阵Y=XP (2)降维前原始数据的绘图 结果: (3)降维后的矩阵Y针对不同分类采用不同颜色绘图 结果: (4)结论:由(2)和(3)中图可以看出,经过PCA后,基本核心特征被保留,分类的结果更加明显。 1.PCA的数学原理,作者:张洋,链接:http://blog.codinglabs.org/articles/pca-tutorial.html 3.变换矩阵P的求解原理
三、PCA实现步骤
四、python代码实例(python3.7)
1.读取数据,并且设定第一行名称
import numpy as np
import pandas as pd
df = pd.read_csv('iris.data')
df.head()
df.columns=['sepal_len', 'sepal_wid', 'petal_len', 'petal_wid', 'class']
df.head() sepal_len sepal_wid petal_len petal_wid class
0 4.9 3.0 1.4 0.2 Iris-setosa
1 4.7 3.2 1.3 0.2 Iris-setosa
2 4.6 3.1 1.5 0.2 Iris-setosa
3 5.0 3.6 1.4 0.2 Iris-setosa
4 5.4 3.9 1.7 0.4 Iris-setosa2.将数据集分为数据类X和类别类Y
# split data table into data X and class labels y
X = df.iloc[:,0:4].values
y = df.iloc[:,4].values3.将X的每一列(代表一个属性字段)进行零均值化
from sklearn.preprocessing import StandardScaler
X_std = StandardScaler().fit_transform(X)
print (X_std)4.求取X的协方差矩阵
mean_vec = np.mean(X_std, axis=0)
cov_mat = (X_std - mean_vec).T.dot((X_std - mean_vec)) / (X_std.shape[0]-1)
print('Covariance matrix \n%s' %cov_mat)
结果:
Covariance matrix
[[ 1.00675676 -0.10448539 0.87716999 0.82249094]
[-0.10448539 1.00675676 -0.41802325 -0.35310295]
[ 0.87716999 -0.41802325 1.00675676 0.96881642]
[ 0.82249094 -0.35310295 0.96881642 1.00675676]]print('NumPy covariance matrix: \n%s' %np.cov(X_std.T))
结果:
NumPy covariance matrix:
[[ 1.00675676 -0.10448539 0.87716999 0.82249094]
[-0.10448539 1.00675676 -0.41802325 -0.35310295]
[ 0.87716999 -0.41802325 1.00675676 0.96881642]
[ 0.82249094 -0.35310295 0.96881642 1.00675676]]5.求取协方差矩阵的特征值和特征向量
cov_mat = np.cov(X_std.T)
eig_vals, eig_vecs = np.linalg.eig(cov_mat)
print('Eigenvectors \n%s' %eig_vecs)
print('\nEigenvalues \n%s' %eig_vals)
结果:
Eigenvectors
[[ 0.52308496 -0.36956962 -0.72154279 0.26301409]
[-0.25956935 -0.92681168 0.2411952 -0.12437342]
[ 0.58184289 -0.01912775 0.13962963 -0.80099722]
[ 0.56609604 -0.06381646 0.63380158 0.52321917]]
Eigenvalues
[2.92442837 0.93215233 0.14946373 0.02098259]
6.特征值和特征向量由高到低排序
# Make a list of (eigenvalue, eigenvector) tuples
eig_pairs = [(np.abs(eig_vals[i]), eig_vecs[:,i]) for i in range(len(eig_vals))]
print (eig_pairs)
print ('----------')
# Sort the (eigenvalue, eigenvector) tuples from high to low
eig_pairs.sort(key=lambda x: x[0], reverse=True)
#打印
# Visually confirm that the list is correctly sorted by decreasing eigenvalues
print('Eigenvalues in descending order:')
for i in eig_pairs:
print(i[0])
结果:
[(2.9244283691111144, array([ 0.52308496, -0.25956935, 0.58184289, 0.56609604])), (0.932152330253508, array([-0.36956962, -0.92681168, -0.01912775, -0.06381646])), (0.14946373489813417, array([-0.72154279, 0.2411952 , 0.13962963, 0.63380158])), (0.02098259276427019, array([ 0.26301409, -0.12437342, -0.80099722, 0.52321917]))]
----------
Eigenvalues in descending order:
2.9244283691111144
0.932152330253508
0.14946373489813417
0.020982592764270197.取特征值最大的前2个特征向量,组成转换基P=4×2维
P=np.hstack((eig_pairs[0][1].reshape(4,1),
eig_pairs[1][1].reshape(4,1)))
print('Matrix P:\n', P)
结果:
Matrix P:
[[ 0.52308496 -0.36956962]
[-0.25956935 -0.92681168]
[ 0.58184289 -0.01912775]
[ 0.56609604 -0.06381646]]8.验证结果
Y = X_std.dot(matrix_w)
结果:
array([[-2.10795032, 0.64427554],
[-2.38797131, 0.30583307],
[-2.32487909, 0.56292316],
[-2.40508635, -0.687591 ],
[-2.08320351, -1.53025171],
[-2.4636848 , -0.08795413],
......
[ 1.54200377, 0.90808604],
[ 1.50925493, -0.26460621],
[ 1.3690965 , -1.01583909],
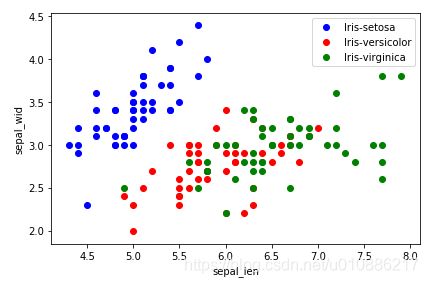
[ 0.94680339, 0.02182097]])plt.figure(figsize=(6, 4))
for lab, col in zip(('Iris-setosa', 'Iris-versicolor', 'Iris-virginica'),
('blue', 'red', 'green')):
plt.scatter(X[y==lab, 0],
X[y==lab, 1],
label=lab,
c=col)
plt.xlabel('sepal_len')
plt.ylabel('sepal_wid')
plt.legend(loc='best')
plt.tight_layout()
plt.show()
plt.figure(figsize=(6, 4))
for lab, col in zip(('Iris-setosa', 'Iris-versicolor', 'Iris-virginica'),
('blue', 'red', 'green')):
plt.scatter(Y[y==lab,0],
Y[y==lab,1],
label=lab,
c=col)
plt.xlabel('Principal Component 1')
plt.ylabel('Principal Component 2')
plt.legend(loc='lower center')
plt.tight_layout()
plt.show()五、参考