Gephi绘制微生物网络图
1 软件下载
链接:https://pan.baidu.com/s/1AdfqBaJVhTOVnNMT_DglRw
提取码:l8ne
需要注意的是,本软件的运行像Cytoscape一样也依赖JAVA,如果你的电脑没有安装较新版本的JRE,可以到官网下载安装。
JRE安装包链接:https://www.java.com/zh_CN/download/
2 数据准备与导入
2.1 软件所需数据介绍
类似于Cytoscape,网络图的数据一般有两个:边文件和点文件。
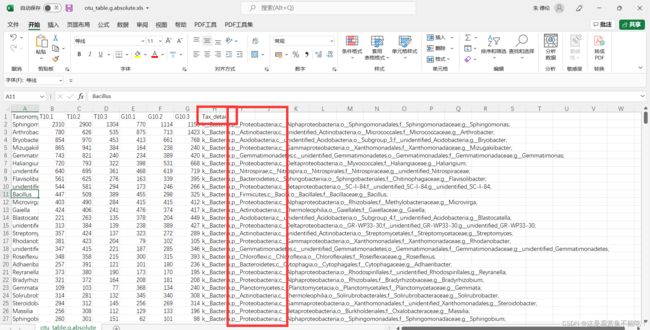
边文件edgeex.csv,如下图,主要是由成对的节点Id构成,Sourse和Target两个点连成一条线(边),边文件中也可以加入边的描述信息,比如边的id、作用类型、权重等。
点文件nodeex.csv,主要是对边文件中的“点”进行描述,是点的属性文件,如下,可添加点的分组信息用于网络图的可视化。
如果大家担心自己准备数据可能会有问题,可参考我这里的表头,你的数据复制到我的文件里即可(在网盘里的edgeex.csv和nodeex.csv文件),没有数据的列,可以空着,最后另存成csv格式(逗号分隔)的文本文件。当然,Excel格式、制表符分隔的文本文件也是支持的,避免最后显示出错,这里仍推荐csv格式。
2.2 数据准备
2.2.1用R分析数据或基迪奥在线分析数据
如2.1所述,绘制网络图我们需要两个文件,edgeex.csv文件和nodeex.csv文件
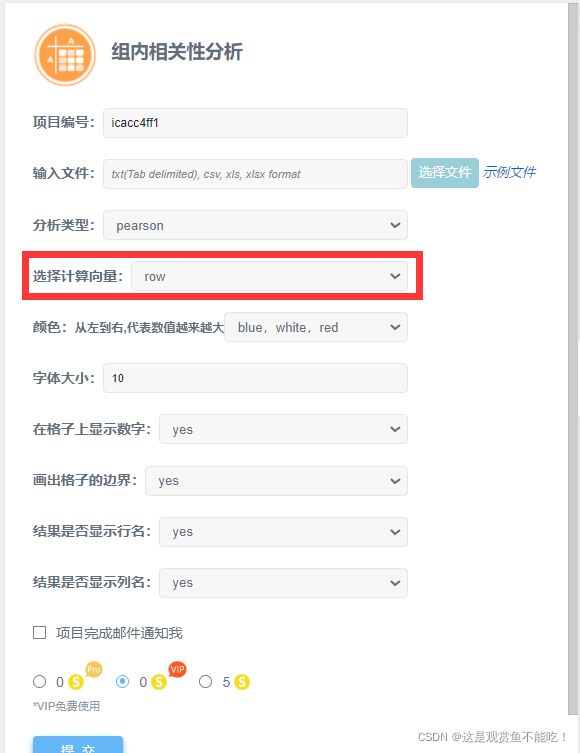
首先,我们把需要做好数据分析,这里使用基迪奥平台的组内相关性分析工具。
OmicShare Tools - 基迪奥生信云工具![]() https://www.omicshare.com/tools/Home/Soft/ica
https://www.omicshare.com/tools/Home/Soft/ica
(组间分析的数据在基迪奥的微生物数据包\16s\result**\result\02.OTUanalysis\taxa_abundance\Absolute中,具体选择哪个要看你要分析们水平还是属水平了。在按照基迪奥平台数据模板上传数据之前我这里把细菌名称改为了n+数字的形式,以便编辑后面的nodeex.csv文件。)
如下如所示,需要注意计算向量为row,然后点提交。
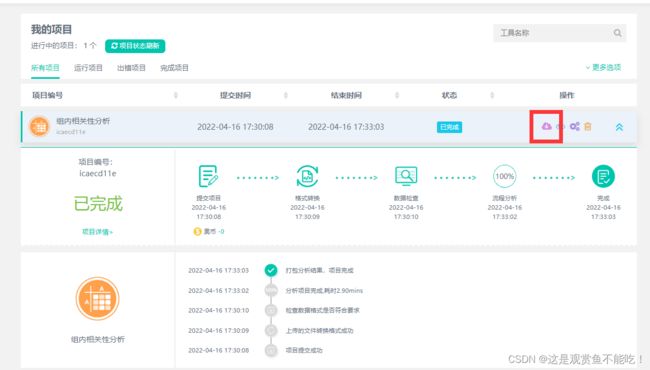
项目完成后点击操作下方下载图标下载文件,解压文件。
2.2.2准备两个数据文件
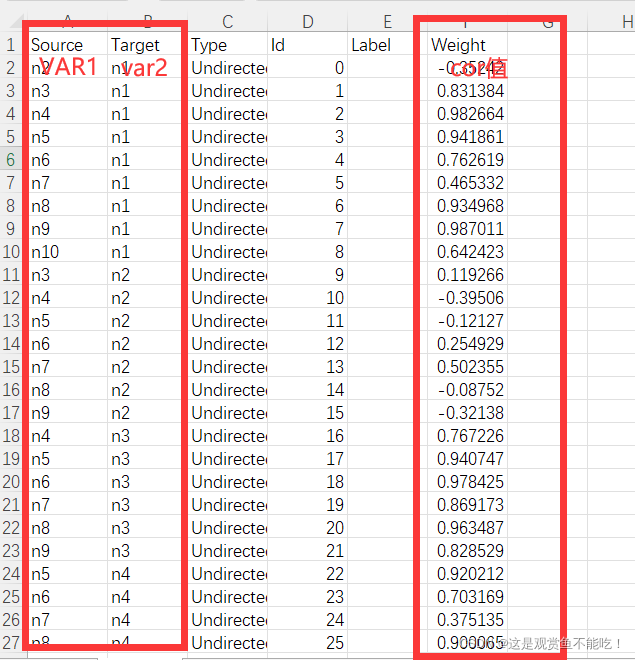
解压文件后打开all.cor_pvalue.list.xls文件
准备edgeex.csv文件
Var1列、Var2列和cor数值对应edgeex.csv边文件中的Source列、Target列和Weight列的数值。,格式如下图。
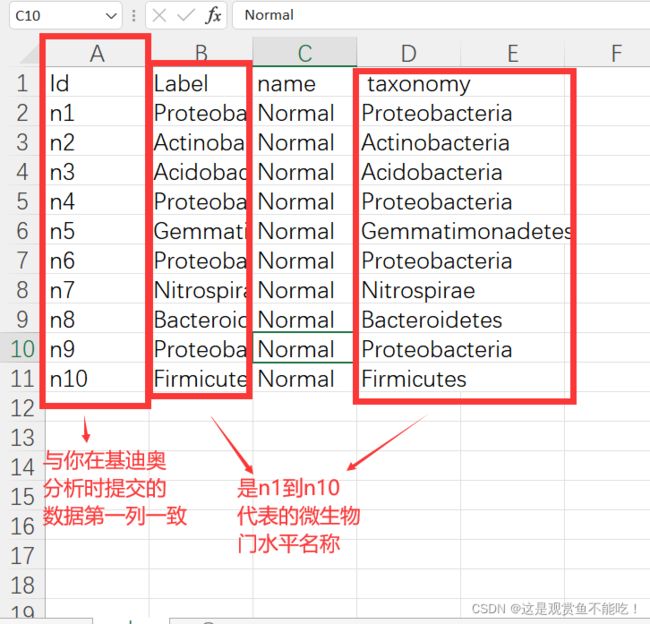
准备nodeex.csv文件,格式如下图。
备注:在填写Label和taxonomy列时可以参基迪奥给的数据包的16s\result**\result\02.OTUanalysis\taxa_abundance\Absolute的文件,这里我选的是.g.属水平的文件表格,找到最后一列,:p*********:这一段就是这个属水平的菌所属的门。
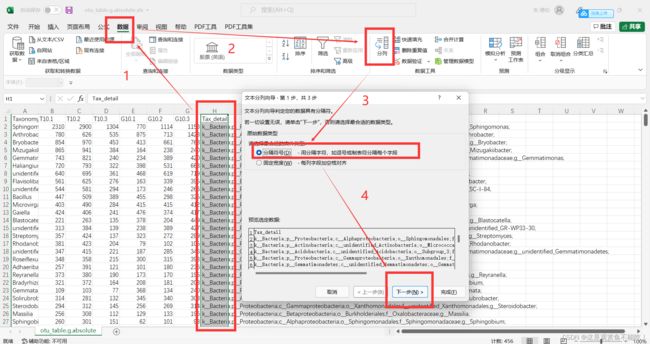
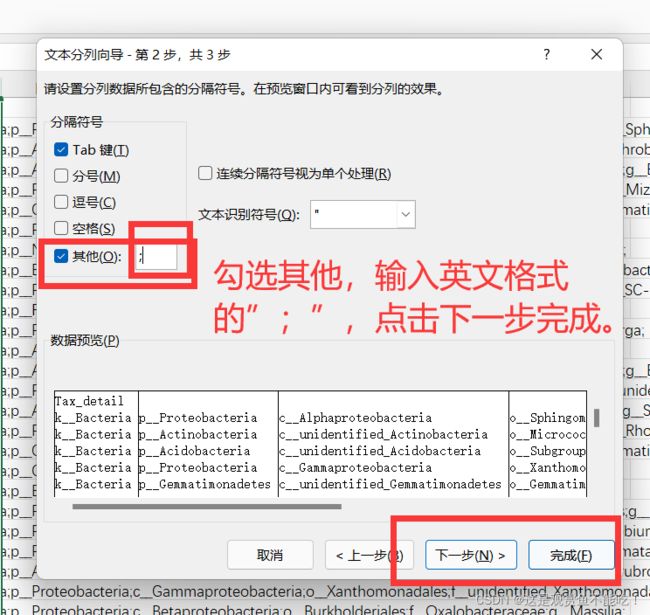
如果你分析的数据太多,可以采用以下办法提取出来。全选这里一列——选择数据中的分列——选下一步。
得出界门纲目科属列,接下来使用替换功能一次性删除”p__“,这样完整的微生物所属的门就可以一次性复制了。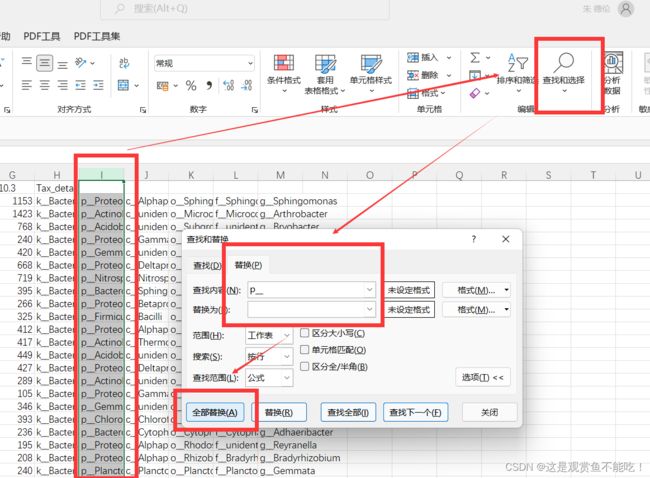
2.3 导入数据
接下来,打开软件,选择文件——导入电子表格,导入边文件表格edgeex.csv边文件表格。
在弹出的窗口中,如下,直接点Next按钮。
接着,一些没有数据的空列可取消勾选,点完成按钮。
新弹窗点OK。
接着,用同样的操作导入点文件表格node.csv,最后一步要注意的是,把点文件信息导入当前workspace中,如下。
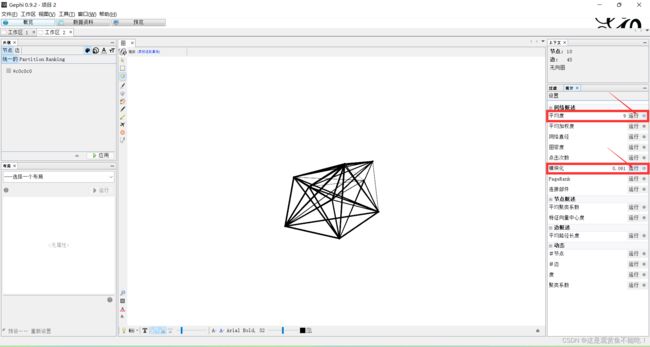
接着在统计选项卡下计算一下网络的属性信息,比如,平均度(Average degree)和模块化指数(Modularity),点击红框的运行,会有弹窗,关闭即可,如下。
3 调整外观
调整颜色
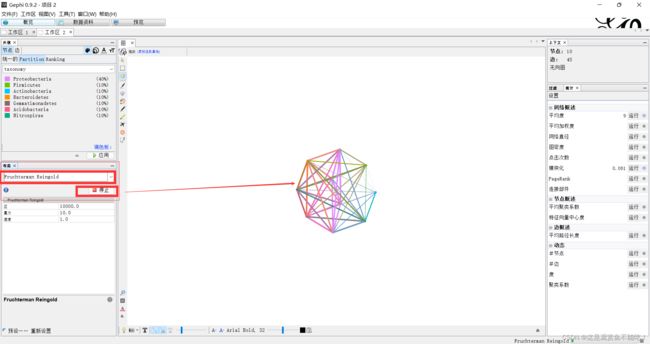
调整形状布局:这里我选择的是Fruchterman Reingold预设布局,点击运行,或者停止。
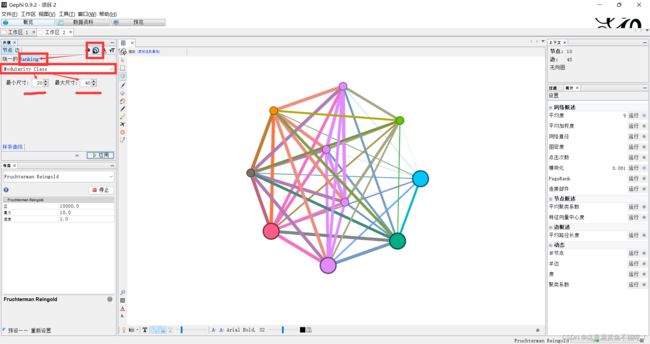
调整节点大小:选择大小图标——选择Ranking——Modularity Class———设置最小最大值,最后点击应用。
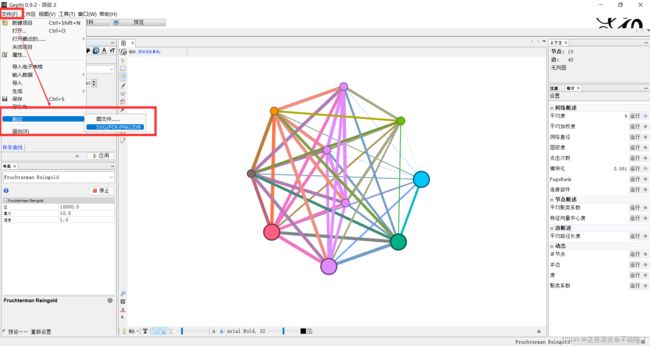
导出文件