机器学习 入门详细解析(三)线性回归、岭回归、逻辑回归、聚类
文章目录
-
- 回归算法-线性回归分析
-
- 线性模型
- 线性回归
- 损失函数(误差大小)
- 最小二乘法之正规方程(不做要求)
- 最小二乘法之梯度下降(理解过程)
- 正规方程与梯度下降的对比
- sklearn 线性回归正规方程、梯度下降API
- 线性回归算法实例——波士顿房价数据集分析
-
- 代码实例
- 梯度下降与正规方程的区别
- LinearRegression与SGDRegressor评估
- 过拟合与欠拟合
- L2正则化
- 带有正则化的线性回归-Ridge
- 分类算法-逻辑回归
-
- 逻辑回归公式
- 逻辑回归的损失函数、优化(了解)
- sklearn 逻辑回归API
- LogisticRegression回归案例
-
- pandas使用
- 良/恶性乳腺癌肿分类流程
- LogisticRegression总结
- 非监督学习(unsupervised learning)
-
- k-means API
- k-means对Instacart Market用户聚类
- Kmeans性能评估指标
- Kmeans性能评估指标API
- Kmeans总结
机器学习 入门详细解析(一)开发流程\sklearn\k近邻算法\朴素贝叶斯算法\交叉验证
机器学习 入门详细解析(二)决策树、随机森林
回归算法-线性回归分析
线性模型
-
试图学得一个通过属性的线性组合来进行预测的函数:
-
f ( x ) = w 1 x 1 + w 2 x 2 + … + w d x d + b ; f(x)=w_1x_1+w_2x_2+…+w_dx_d+b; f(x)=w1x1+w2x2+…+wdxd+b;
注:w为权重,b称为偏置项,可以理解为:w_0×1
线性回归
-
定义:线性回归通过一个或者多个自变量与因变量之间之间进行建模的回归分析。其中特点为一个或多个称为回归系数的模型参数的线性组合
-
一元线性回归:涉及到的变量只有一个
-
多元线性回归:涉及到的变量两个或两个以上
-
通用公式:
h ( w ) = w 0 + w 1 x 1 + w 2 x 2 + … = w T x ℎ(w)= w_0+w_1x_1+w_2x_2+…= w^Tx h(w)=w0+w1x1+w2x2+…=wTx
损失函数(误差大小)
如何去求模型当中的W,使得损失最小?(目的是找到最小损失对应的W值)
最小二乘法之正规方程(不做要求)
求解:
w = ( X T X ) − 1 X T y w=(X^TX)^−1X^Ty w=(XTX)−1XTy
X为特征值矩阵,y为目标值矩阵
缺点:当特征过于复杂,求解速度太慢。 对于复杂的算法,不能使用正规方程求解(逻辑回归等)
最小二乘法之梯度下降(理解过程)
我们以单变量中的w0,w1为例子:
正规方程与梯度下降的对比
sklearn 线性回归正规方程、梯度下降API
sklearn.linear_model.LinearRegression #正规方程
sklearn.linear_model.SGDRegressor #梯度下降
sklearn.linear_model.LinearRegression()
# 普通最小二乘线性回归
# coef_:回归系数
sklearn.linear_model.SGDRegressor( )
# 通过使用SGD最小化线性模型
# coef_:回归系数
线性回归算法实例——波士顿房价数据集分析
波士顿房价数据案例分析流程
- 波士顿地区房价数据获取
- 波士顿地区房价数据分割
- 训练与测试数据标准化处理
- 使用最简单的线性回归模型LinearRegression和梯度下降估计SGDRegressor对房价进行预测
代码实例
from sklearn.datasets import load_boston
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import LinearRegression,SGDRegressor,Ridge
import numpy as np
np.random.seed(1)
def mylinear():
# 获取数据
lb = load_boston()
# 分割数据
x_train, x_test, y_train, y_test = train_test_split(lb.data, lb.target, test_size=0.25)
print(y_train, y_test)
# 特征值标准化处理
std_x = StandardScaler()
x_train = std_x.fit_transform(x_train)
x_test = std_x.transform(x_test)
# 目标值标准化处理
std_y = StandardScaler()
y_train = std_y.fit_transform(y_train.reshape(-1, 1))
y_test = std_y.transform(y_test.reshape(-1, 1))
# 预测房价结果
model = joblib.load("./tmp/test.pkl")
y_predict = std_y.inverse_transform(model.predict(x_test))
print("保存的模型预测的结果:", y_predict)
if __name__ == "__main__":
mylinear()
sklearn 回归评估 API
sklearn.metrics.mean_squared_error
mean_squared_error(y_true, y_pred)
# 均方误差回归损失
# y_true:真实值
# y_pred:预测值
# return:浮点数结果
# 注:真实值,预测值为标准化之前的额值
梯度下降与正规方程的区别
LinearRegression与SGDRegressor评估
- 特点:线性回归器是最为简单、易用的回归模型。从某种程度上限制了使用,尽管如此,在不知道特征之间关系的前提下,我们仍然使用线性回归器作为大多数系统的首要选择。
- 小规模数据:LinearRegression (不能解决拟合问题)以及其他
- 大规模数据:SGDRegressor
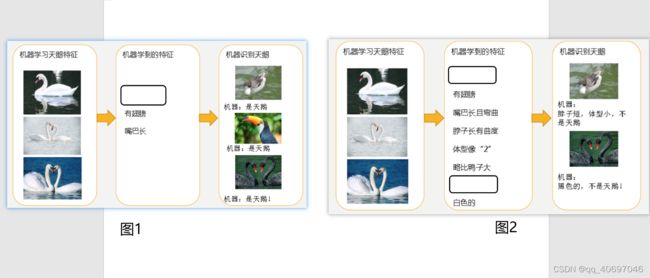
过拟合与欠拟合
- 分析上图1
- 经过训练后,知道了天鹅是有翅膀的,天鹅的嘴巴是长长的。简单的认为有这些特征的都是天鹅。因为机器学习到的天鹅特征太少了,导致区分标准太粗糙,不能准确识别出天鹅。
- 分析上图2
- 机器通过这些图片来学习天鹅的特征,经过训练后,知道了天鹅是有翅膀的,天鹅的嘴巴是长长的弯曲的,天鹅的脖子是长长的有点曲度,天鹅的整个体型像一个"2"且略大于鸭子。这时候机器已经基本能区别天鹅和其他动物了。然后,很不巧已有的天鹅图片全是白天鹅的,于是机器经过学习后,会认为天鹅的羽毛都是白的,以后看到羽毛是黑的天鹅就会认为那不是天鹅。
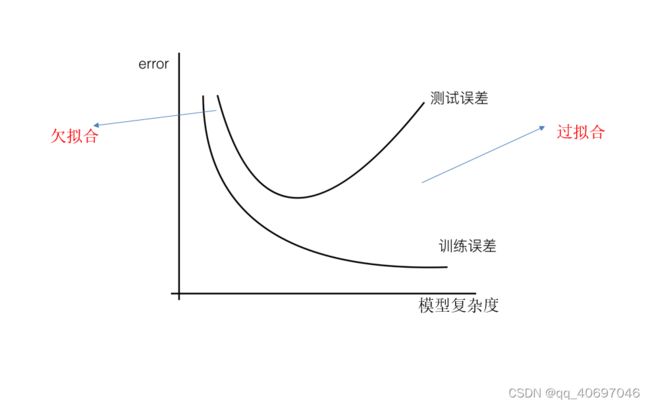
**过拟合:**一个假设在训练数据上能够获得比其他假设更好的拟合, 但是在训练数据外的数据集上却不能很好地拟合数据,此时认为这个假设出现了过拟合的现象。(模型过于复杂)
**欠拟合:**一个假设在训练数据上不能获得更好的拟合, 但是在训练数据外的数据集上也不能很好地拟合数据,此时认为这个假设出现了欠拟合的现象。(模型过于简单)
解决方法
- 过拟合原因以及解决办法
- 原因:原始特征过多,存在一些嘈杂特征, 模型过于复杂是因为模型尝试去兼顾各个测试数据点
- 解决办法:进行特征选择,1.消除关联性大的特征(很难做)。2.交叉验证(让所有数据都有过训练)。3.正则化(了解)
- 欠拟合原因以及解决办法
- 原因:学习到的数据特征过少
- 解决办法:增加数据的特征数量
L2正则化
作用:可以使得W的每个元素都很小,都接近于0
优点:越小的参数说明模型越简单,越简单的模型则越不容易产生过拟合现象
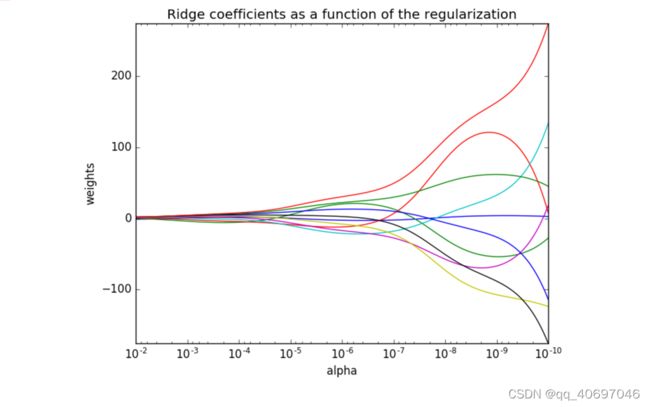
带有正则化的线性回归-Ridge
sklearn.linear_model.Ridge(alpha=1.0)
# 具有l2正则化的线性最小二乘法
# alpha:正则化力度
# coef_:回归系数
观察正则程度的变化,对结果的影响
线性回归 LinearRegression 与 Ridge 对比
- 岭回归:回归得到的回归系数更符合实际,更可靠。另外,能让估计参数的波动范围变小,变的更稳定。在存在病态数据偏多的研究中有较大的实用价值
# estimator预测
# 正规方程求解方式预测结果
lr = LinearRegression()
lr.fit(x_train, y_train)
print("系数:", lr.coef_)
# 保存好训练好的模型
joblib.dump(lr, "./tmp/test2.pkl")
# 预测测试集的房价
y_lr_predict = std_y.inverse_transform(lr.predict(x_test))
print("正规方程测试集里面每个房子的预测价格:", y_lr_predict)
print("正规方程的均方误差:", mean_squared_error(std_y.inverse_transform(y_test),y_lr_predict))
# 梯度下降求解方式预测结果
sgd = SGDRegressor()
sgd.fit(x_train, y_train)
print("梯度下降系数:", sgd.coef_)
# 梯度下降测试集的预测结果
y_sgd_predict = std_y.inverse_transform(sgd.predict(x_test).reshape(-1, 1))
print("梯度下降测试集里面每个房子的预测价格:", y_sgd_predict)
print("梯度下降的均方误差:", mean_squared_error(std_y.inverse_transform(y_test), y_sgd_predict))
# 岭回归求解方式预测结果
rd = Ridge(alpha=1.0)
rd.fit(x_train, y_train)
print("岭回归系数:", rd.coef_)
# 梯度下降测试集的预测结果
y_rd_predict = std_y.inverse_transform(rd.predict(x_test).reshape(-1, 1))
print("岭回归测试集里面每个房子的预测价格:", y_rd_predict)
print("岭回归的均方误差:", mean_squared_error(std_y.inverse_transform(y_test), y_rd_predict))
分类算法-逻辑回归
逻辑回归是解决二分类问题的利器
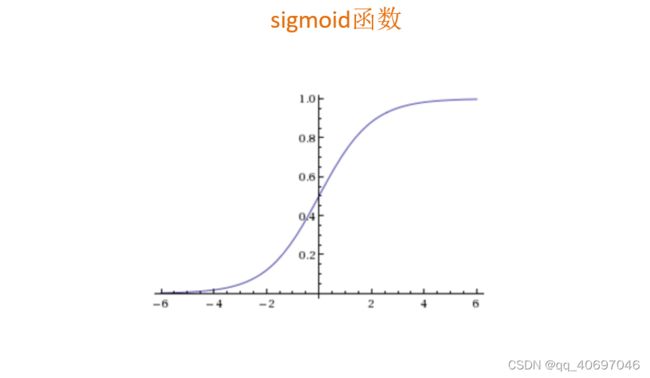
逻辑回归公式
sigmoid函数为:
逻辑回归的损失函数、优化(了解)
sklearn 逻辑回归API
sklearn.linear_model.LogisticRegression(penalty=‘l2’, C = 1.0)
# Logistic回归分类器
# coef_:回归系数
LogisticRegression回归案例
原始数据的下载地址:
https://archive.ics.uci.edu/ml/machine-learning-databases/
数据描述
(1)699条样本,共11列数据,第一列用语检索的id,后9列分别是与肿瘤
相关的医学特征,最后一列表示肿瘤类型的数值。
(2)包含16个缺失值,用”?”标出。
pandas使用
pd.read_csv(’’,names=column_names)
column_names:指定类别名字,['Sample code number','Clump Thickness', 'Uniformity of Cell Size','Uniformity of Cell Shape','Marginal Adhesion','Single Epithelial Cell Size','Bare Nuclei','Bland Chromatin','Normal Nucleoli','Mitoses','Class']
return:数据
replace(to_replace=’’,value=):返回数据
dropna():返回数据
良/恶性乳腺癌肿分类流程
- 网上获取数据(工具pandas)
- 数据缺失值处理、标准化
- LogisticRegression估计器流程
代码实例
from sklearn.linear_model import LinearRegression, SGDRegressor, Ridge, LogisticRegression
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.metrics import mean_squared_error, classification_report
import pandas as pd
import numpy as np
def logistic():
"""
逻辑回归做二分类进行癌症预测(根据细胞的属性特征)
:return: NOne
"""
# 构造列标签名字 名字自定,但是数目要一致
column = ['Sample code number','Clump Thickness', 'Uniformity of Cell Size','Uniformity of Cell Shape','Marginal Adhesion', 'Single Epithelial Cell Size','Bare Nuclei','Bland Chromatin','Normal Nucleoli','Mitoses','Class']
# 读取数据
data = pd.read_csv("https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data", names=column)
print(data)
# 缺失值进行处理
data = data.replace(to_replace='?', value=np.nan)
data = data.dropna()
# 进行数据的分割
x_train, x_test, y_train, y_test = train_test_split(data[column[1:10]], data[column[10]], test_size=0.25)
# 进行标准化处理
std = StandardScaler()
x_train = std.fit_transform(x_train)
x_test = std.transform(x_test)
# 逻辑回归预测
lg = LogisticRegression(C=1.0)
lg.fit(x_train, y_train)
print(lg.coef_)
y_predict = lg.predict(x_test)
print("准确率:", lg.score(x_test, y_test))
print("召回率:", classification_report(y_test, y_predict, labels=[2, 4], target_names=["良性", "恶性"]))
return None
if __name__ == "__main__":
logistic()
结果为:
[[1.20978993 0.2487854 0.63901048 0.73015212 0.39714288 1.40510768
0.97045854 0.40176488 0.80020768]]
准确率: 0.9707602339181286
召回率: precision recall f1-score support
良性 0.97 0.99 0.98 114
恶性 0.98 0.93 0.95 57
accuracy 0.97 171
macro avg 0.97 0.96 0.97 171
weighted avg 0.97 0.97 0.97 171
LogisticRegression总结
- 应用:广告点击率预测、电商购物搭配推荐
- 优点:适合需要得到一个分类概率的场景
- 缺点:当特征空间很大时,逻辑回归的性能不是很好(看硬件能力)
非监督学习(unsupervised learning)
k-means API
sklearn.cluster.KMeans(n_clusters=8,init=‘k-means++’)
# k-means聚类
# n_clusters:开始的聚类中心数量
# init:初始化方法,默认为'k-means ++’
# labels_:默认标记的类型,可以和真实值比较(不是值比较)
k-means对Instacart Market用户聚类
- 降维之后的数据
- k-means聚类
- 聚类结果显示
Kmeans性能评估指标
轮廓系数:
$$
计算公式:_= (_− _)/(max(_ ,_))
$$
注:对于每个点i 为已聚类数据中的样本 ,b_i 为i 到其它族群的所有样本的平均距离,a_i 为i 到本身簇的距离平均值最终计算出所有的样本点的轮廓系数平均值
如果〖〗_ 小于0,说明_ 的平均距离大于最近的其他簇。
聚类效果不好
如果〖〗_ 越大,说明_ 的平均距离小于最近的其他簇。
聚类效果好
轮廓系数的值是介于 [-1,1] ,越趋近于1代表内聚度和分离度都相对较优
Kmeans性能评估指标API
sklearn.metrics.silhouette_score(X, labels)
计算所有样本的平均轮廓系数
X:特征值
labels:被聚类标记的目标值
Kmeans总结
-
特点分析: 采用迭代式算法,直观易懂并且非常实用
-
缺点:容易收敛到局部最优解(多次聚类),需要预先设定簇的数量(k-means++解决)