【机器学习】吴恩达作业7.1,python实现PCA主成分分析
7.1主成分分析
主成分分析(PCA)来实现降维。
首先用一个二维的样本集来实验,对PCA如何运行的有一个直观的感受,然后再在一个更大的由5000个人脸图像组成的数据集上实现PCA。
降噪 :
降维就是一种对高维度特征数据预处理方法。降维是将高维度的数据保留下最重要的一些特征,去除噪声和不重要的特征,从而实现提升数据处理速度的目的。在实际的生产和应用中,降维在一定的信息损失范围内,可以为我们节省大量的时间和成本。降维也成为应用非常广泛的数据预处理方法。
降维具有如下一些优点:
- 使得数据集更易使用。
- 降低算法的计算开销。
- 去除噪声。
- 使得结果容易理解。
PCA :
PCA(Principal Component Analysis),即主成分分析方法,是一种使用最广泛的数据降维算法。PCA的主要思想是将n维特征映射到k维上,这k维是全新的正交特征也被称为主成分,是在原有n维特征的基础上重新构造出来的k维特征。
差最小)
1 压缩数据(减少内存和硬盘的需求/加速算法)
2 可视化数据
线性回归和神经网络算法,都可以先使用PCA对特征进行降维
步骤:
先将数据进行均值归一化(mean normalization),然后找到一个低维平面,将数据投影到上面,使得投影误差(projection error)最小(即找到一组基向量,然后将数据投影到这个向量空间里,使得投影误差最小
选择主成分K的数量:
使误差降到最小,使绝大部分方差得以保留
不要用PCA去防止过拟合,可能会丢失一些有价值的信息!
除非原始数据直接建模不好(内存太大或者运行太慢),才使用PCA进行降维处理之后再进行建模,一般建议直接用原始数据进行建模
代码 :
1 导入数据及可视化
import numpy as np
import matplotlib.pyplot as plt
import scipy.io as sio
data = sio.loadmat('ex7data1')
data
X = data['X']
X.shape#(50, 2)
data.keys()#dict_keys(['__header__', '__version__', '__globals__', 'X'])
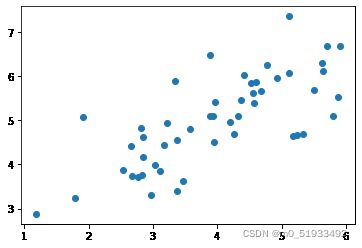
plt.scatter(X[:,0],X[:,1])
plt.show
2 对数据标准化,可视化
#对X标准化
def demean(X):
# X_demean = X - np.mean(X,axis = 0)#对纵轴求均值
mu = X.mean(axis=0) #求每列的
sigma = X.std(axis=0, ddof=1) #无偏的标准差,自由度为n-1
X = (X-mu)/sigma
return X, mu, sigma
X_demean, mu, sigma = demean(X)
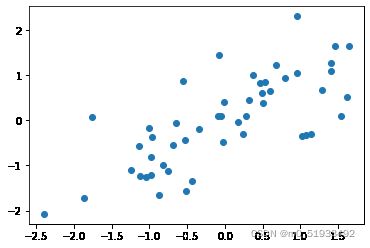
plt.scatter(X_demean[:,0],X_demean[:,1])
plt.show3 计算协方差及特征向量
比较两个不同的特征向量(第1列和第2列)哪个投影误差更小,画出以数据均值为中心的特征向量,取U矩阵中第1列得到矩阵([-0.70710678, -0.70710678])
#得到协方差和特征向量U
def pca(x):
m = len(X)
C = (x.T @ x)/m #计算协方差矩阵 主对角线上是X的方差,副对角线是X的协方差
U, S, V = np.linalg.svd(C) #SVD奇异值分解计算特征向量U
return U, S
U, S = pca(X_demean)
#U1 = U[:,0]#取特征向量中第一列矩阵
#U1.shape #(2,)
#画出特征向量
plt.figure(figsize=(6,6))
plt.scatter(X[:,0], X[:,1], marker='o', facecolors='none', edgecolors='b')
plt.plot([mu[0], mu[0]+1.5*S[0]*U[0,0]], [mu[1], mu[1]+1.5*S[0]*U[1,0]], 'k') #两个点的连线
plt.plot([mu[0], mu[0]+1.5*S[1]*U[0,1]], [mu[1], mu[1]+1.5*S[1]*U[1,1]], 'k') #这里的1.5和S表示特征向量的长度
plt.title('Computed eigenvectors of the dataset')4 将均值化后的数据投影到特征向量上,实现降维
#降维,将均值化后的数据投影到特征向量上
def X_reduction(x, U, K):
U_reduce = U[:, 0:K]#选择特征向量矩阵的前K列
return X @ U_reduce
#降维(50, 1)
z = X_reduction(X_demean, U, 1)#K取1
z[0] #1.481273915 重建数据,得到数据的近似值
#重建降维后的数据
def recoverData(z, U, K):
U_reduce = U[:, 0:K]
return z @ U_reduce.T #U是特征向量矩阵,由标准正交基组成,U.T@U为单位矩阵
#重建后的数据
X_restore = recoverData(z, U, 1)
X_restore[0,:] #[-1.04741883, -1.04741883]6 可视化投影,画出标准化数据以及投影(降维)后又重建的数据,对应点用虚线连接
#可视化投影
plt.scatter(X[:,0],X[:,1],marker='o', facecolors='none', edgecolors='b')
plt.scatter(X_restore[:,0],X_restore[:,1],marker='o', facecolors='none', edgecolors='r')
for i in range(len(X)):
plt.plot([X[i,0], X_restore[i,0]], [X[i,1], X_restore[i,1]], 'k--')
plt.title('The normalized and projected data after PCA')
plt.show()7 查看降维后的数据保留了原始数据多少差异性
# 查看保留差异
def retained_variance(S, K):
rv = np.sum(S[:K])/np.sum(S)
return print('{:.2f}%'.format(rv*100))
retained_variance(S, 1) #86.78%案例:使用pca进行图像的降维
mat = sio.loadmat('ex7faces.mat')
mat.keys()
A = mat['X']
A.shape#(5000, 1024)
def plot_100_images(A):
fig,axs = plt.subplots(ncols = 10,nrows = 10,figsize = (10,10))#定义n*n的子画布(n = 10)
#在每个子画布中画出一个图像
for c in range(10):
for r in range(10):
axs[c,r].imshow(A[10*c + r].reshape(32,32).T,cmap = 'Greys_r')#显示单通道的灰图
axs[c,r].set_xticks([])
axs[c,r].set_yticks([])
plot_100_images(A) A_demean = A - np.mean(A,axis = 0)#A去均值化
C = A_demean.T@A_demean/len(A)#计算协方差
U,S,V = np.linalg.svd(C)#特征值 特征向量
U1 = U[:,:36]
U.shape,U1.shape#((1024, 1024), (1024, 36))
A_reduction = A_demean @ U1#对A进行降维
A_reduction.shape#(5000, 36)
A_recover = A_reduction @ U1.T + np.mean(A,axis = 0)
plot_100_images(A_recover),plot_100_images(A)#原始数据与还原后数据做对比