Medical Image Synthesis for Data Augmentation and Anonymization
Medical Image Synthesis for Data Augmentation and Anonymization Using Generative Adversarial Networks
摘要
在训练深度学习模型时,数据多样性是成功的关键。医学影像数据集通常是不平衡的,因为病理发现通常是稀少的,这在训练深度学习模型时带来了重大挑战。在这项工作中,我们提出了一种方法,通过使用两个公开可用的大脑MRI数据集训练生成对抗网络,来生成带有脑瘤的合成异常MRI图像。我们展示了合成图像提供的两个独特好处。首先,我们说明利用合成图像作为数据增强的一种形式来改善肿瘤分割的性能。其次,我们展示了生成模型作为匿名化工具的价值,在合成数据和真实受试者数据上进行训练时,可以获得可比的肿瘤分割结果。总之,这些结果为机器学习在医学成像领域面临的两个最大挑战提供了一个潜在的解决方案,即病理发现的小发生率和患者数据共享方面的限制。
关键词:生成模型 GAN 图像合成 深度学习 脑肿瘤 磁共振成像 MRI分割
1 介绍
众所周知,足够的数据量是训练一个成功的机器学习算法1用于医学图像分析的必要条件。分类不平衡性高或2可变性不足的数据导致分类性能差。在异常并不常见医学影像领域中,这通常证明是有问题的。而且,在图像分割任务中,手动注释体数据所需的时间只会加剧这种差异;人工分割三维异常数据需要15分钟以上的时间,这在繁忙的放射医学实践中是不现实的。结果是带注释的数据非常少,并且在尝试训练一个精确的算法时面临相当大的挑战。虽然传统的数据增强技术(如裁剪、平移、旋转)可以缓解其中一些问题,但它们从根本上产生了高度相关的图像训练数据。
在本文中,我们通过使用生成对抗网络(GAN)3生成合成图像来演示这一问题的一种潜在解决方案,该网络提供了一种额外的数据增强形式,也作为数据匿名化的一种有效方法。利用脑解剖和肿瘤的分割mask生成异常脑(含肿瘤)的多参数磁共振图像。这提供了一种可自动化、低成本的多种数据来源,可用于补充训练集。例如,我们可以改变肿瘤的大小,改变它的位置,或将肿瘤放置在健康的大脑中,从而系统地获得图像和相应的注释。此外,GAN利用医院的数据进行训练,生成合成图像,可以在医院外共享数据,作为匿名化工具使用。
医学图像的仿真与合成研究已经有一段时间了,在医学图像领域4中越来越受到关注。一部分原因是数据可用性的指数级增长,另一部分原因是更好的机器学习模型和支持系统的可用性。已经有12篇医学影像合成与仿真的最新研究成果在医学影像的仿真与合成专刊4上被发表了。
本研究属于合成的分类,相关研究最多的是Chartsias5和Costa等人的6。我们使用公开的数据集(ADNI和BRATS)来演示多参数MRI图像合成,Chartsias等人5使用BRATS和ISLES(缺血性中风病变分割(ISLES) 2015挑战)数据集。尽管如此,虽然合成图像的评价标准在MSE、SSIM和PSNR上得到了证明,但在诊断质量上没有直接的评价标准。Costa等人的6使用GAN生成带有标签的合成视网膜图像,但与本研究相比,代表更多不同病理特征的能力有限。另外,之前的两个作品都是在2D图像或3D图像切片/视图上展示的,而在这个作品中,我们直接处理3D输入/输出。当输入/输为多参数(T1/T2/T1c/Flair)时,输入/输出维数为4D。我们相信,在本质上以3D/4D的方式处理数据,能够更好地反映数据的真实性及其相关问题。
去年,GANs在医学成像中的使用大幅增加,这反映了机器学习社区的总体趋势。GANs已被用于从单个术前MRI7生成运动模型,对低分辨率眼底图像8采样,从脑MRI9生成合成头部CT,并从T1加权10合成T2加权MRI(反之亦然)。使用GANs进行分割在1112中得到了证明。
最后,Frid-Adar等人在肝损伤分类13的背景下利用GAN进行数据增强。据我们所知,目前还没有关于生成合成医学图像作为匿名化和数据增强的形式用于肿瘤分割任务的文献。
2 数据
2.1 数据集
我们使用了两个公开的脑部MRI数据集:
Alzheimer’s Disease Neuroimaging Initiative (ADNI) Data Set
ADNI成立于2003年,是一项公私合作项目,由首席研究员Michael W. Weiner博士领导,ADNI的主要目标是测试序列磁共振成像(MRI)、正电子发射断层扫描(PET)、其他生物标志物以及临床和神经心理学评估是否可以结合起来测量轻度认知障碍(MCI)和早期阿尔茨海默病(AD)的进展。有关ADNI研究的最新信息,请参见www.adni-info.org。我们采用14]的方法,该方法被证明对ADNI数据的脑图谱分割有效。采用SPM1215分割算法和ANTs SyN16非线性配准算法,得到ADNI T1加权图像的白质、灰质和脑脊液图谱。总共有3416对T1加权MRI及其对应的分段组织类图像。
Multimodal Brain Tumor Image Segmentation Benchmark (BRATS) Data Set
BRATS利用多机构术前核磁共振成像技术,专注于本质上异质性(外观、形状和组织学)的脑肿瘤的分割,即脑胶质瘤17。每个患者的MRI图像集包括各种序列,包括T1加权、T2加权、对比增强T1和FLAIR,以及水肿、增强肿瘤和非增强肿瘤的体素级基底真实注释(Ground Truth)。有关BRATS数据集的更多细节,请参见braintumorsegmentation.org。BRATS挑战赛每年举办一次,我们使用的是BRATS 2015训练数据集,该数据集是公开的。
Dataset Split and Pre-processing
作为预处理步骤,我们对ADNI数据集执行头骨剥离18操作,因为BRATS数据集中没有头骨。BRATS 2015训练集提供264项研究数据,我们使用前80%作为训练集,剩余20%作为测试集,以评估最终算法性能。在训练集内进行超参数优化,对评估的每种算法和设置只评估测试集一次。我们的GAN是在3D环境下工作的,由于内存和计算的限制,训练图像轴向裁剪,训练图像被轴向裁剪,为了包含中心的108片,剔除中心区域上下的部分,然后重新采样到128×128×54进行模型训练和推理。为了公平评价BRATS挑战的分割性能,我们使用了分辨率为256×256×108的原始图像进行评价和比较。然而,也有可能非常小的肿瘤在下采样中丢失,从而影响最终的分割性能。
3 方法
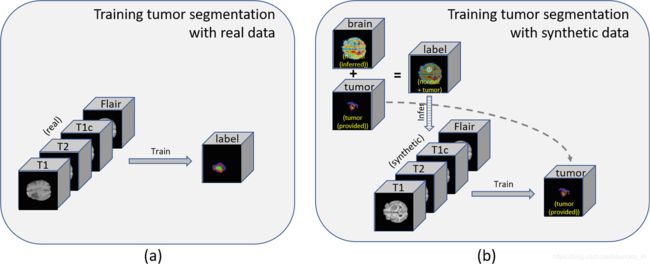
采用19中引入的图像到图像平移条件GAN (pix2pix)模型,实现了标签到MRI(合成图像生成)和MRI到标签(图像分割)的转换。对于大脑分割,生成器G将ADNI的T1加权图像作为输入,训练生成含有白质、灰质和CSF(脑脊液)的mask。另一方面,判别器D被训练来区分“真实”标签和合成的“假”标签。在这个过程中(如图1(a)所示),生成器G从T1加权MRI输入中学习分割大脑标签。由于我们在BRATS数据集中没有合适的现成的大脑解剖分割方法,而且ADNI数据集不包含肿瘤信息,我们首先训练pix2pix模型从ADNI数据集的T1加权图像中实现正常的大脑分割。然后我们使用这个模型对BRATS数据集的T1系列进行推演。神经结构的分割,结合BRATS数据集提供的肿瘤分割,提供了一个完整的大脑肿瘤分割。合成图像生成是通过将输入反转到生成器并训练鉴别器来执行反向任务来训练的(例如,“这些成像数据是从扫描仪获取的还是合成生成的?”)而不是“这种分割是基础事实的注解还是综合生成的?”——图1 (b))我们从标签生成合成的异常大脑MRI,并通过调整这些标签(例如,改变肿瘤的大小,移动肿瘤的位置,或将肿瘤放置在没有肿瘤的大脑标签上)来引入可变性。然后再次使用GAN分割模块,从BRATS数据集分割肿瘤(输入:多参数MRI;输出:肿瘤标签)。我们比较了分割性能(1)有和没有额外的合成数据,(2)只使用合成数据并在10%的真实数据上微调模型;并将他们的GAN性能与BRATS 2017挑战赛中最出色的算法20进行比较。
图1 训练GAN的过程(a) MRI-to-brain分割;(b) label-to-MRI合成;© MRI-to-tumor分割
图2 获取具有变化的合成图像的流程。在BRATS数据集上,将MRI-to-label图像翻译GAN应用于T1加权图像,得到脑图谱。然后,将它与BRATS数据集中给出的肿瘤标签合并,可能还会发生改变(转移肿瘤位置;扩大;收缩)。然后,将合并的标签(可能有改变)作为label-to-MRI GAN的输入,生成脑肿瘤的合成多参数MRI。
3.1 合成图像的数据增强
训练后的GAN从标签生成合成图像,允许生成任意的多系列异常大脑核磁共振成像。既然我们分别有大脑解剖标签和肿瘤标签,我们可以改变肿瘤标签或大脑标签来得到我们想要的合成图像。例如,我们可以改变肿瘤的特征,如大小、现有的大脑和肿瘤标签的位置,或者将肿瘤标签放在一个没有肿瘤的大脑标签上。示例如图3所示。
图3 生成图像的示例。第一行是合成肿瘤所基于的原始(“真实”)图像。未调整分割标签的生成图像显示在第二行。对肿瘤分割标签进行各种调整的生成图像的示例显示在第三至第五行。最后一行描述合成图像的示例,其中肿瘤标签放置在来自ADNI数据集的无肿瘤脑标签上。
本研究尚未对大脑分割算法性能的影响进行评估。由于GAN首先在来自ADNI数据集的3416对T1加权(T1)图像上进行训练,因此生成的T1图像质量高,并且在定性上难以与原始图像区分。使用BRATS数据训练生成非T1加权图像序列。对比增强的T1加权图像使用与T1加权图像相同的图像采集方案。因此,合成的对比度增强T1图像看起来相当真实,尽管在一些生成的图像中可以观察到沿肿瘤边界的更高的对比度。T2加权(T2)和FLAIR图像采集与T1加权图像有着本质上的不同,这使得合成图像与扫描仪采集的图像之间的区别不那么具有挑战性。然而,如果在所有这些模态上有足够大的训练集,早期证据表明在所有模态上生成真实的合成图像是可能的。
除了提高图像分辨率和获取更多的数据,特别是对于T1加权图像以外的序列,还有一些重要的途径可以探索以提高整体图像质量。例如,在放置合成肿瘤时,可能需要更多地注意肿瘤边界,使其看起来不重叠和离散。此外,大脑分割算法的性能及其跨不同数据集的泛化能力需要检查,以获得来自不同的患者群体的更高质量的合成图像数集。
除了常用的数据增强方法,如随机裁剪、旋转、平移或弹性变形21之外,还可以使用合成图像的增强。此外,使用基于GAN的合成图像生成方法,我们对增强图像有更多的控制,与通常的数据增强技术相比,我们有更多的输入选项(即标签)来扰动给定图像。通常的数据增强方法主要依赖于随机过程,并在整个图像水平上运行,而不是特定的位置,如肿瘤。此外,由于我们从相应的标签生成图像,我们得到更多的图像来进行训练,而不需要经过劳动密集型的手工注释过程。图4显示了用真实图像和合成图像以及标签对训练GAN的过程。
图4 训练GAN用于肿瘤分割(a) 真实数据;和(b)合成图像标签对。合成数据生成可以增加具有所需特征(如肿瘤大小、位置等)的训练数据集,而不需要人工密集标注。
3.2 生成具有可变性的匿名合成图像
个人健康信息(PHI)的保护是处理患者数据的一个关键方面。通常情况下,对患者数据传播的关注限制了研究社区的数据可用性,阻碍了该领域的发展。在删除所有DICOM元数据和头骨剥离数据的同时通常会消除几乎所有可识别的信息,向医院的数据共享委员会证明这一点几乎是不可能的。仅仅对数据进行反标识是不够的。此外,当模型来自敏感的患者数据时,它们本身也需要谨慎。22已经证明,私人数据可以从训练过的模型中提取出来。开发一种能生成合成但真实的数据的GAN或许可以解决这个问题。图3的前两行说明了,即使使用相同的分割mask,在生成的数据和原始数据之间也可以观察到显著的变化。这表明GAN产生的图像并不反映潜在的患者个体,而是从总体人群中提取个体。它产生新的数据,不能归因于一个单一的病人,而是一个实例的训练数据集条件下提供的分割。
4 实验和结果
4.1 使用合成数据进行数据增强
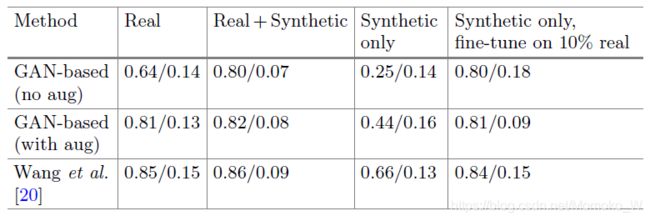
对基于GAN的模型和Wang等人20(在真实和合成数据集上)的模型所产生的整个肿瘤分割的Dice得分进行评价,结果如表1所示。分割模型仅在BRATS的15个训练数据中的80%上进行训练,训练数据辅以合成数据。Dice得分是根据BRATS的15个训练数据中的20% 保留数据集进行评估的。所有模型都经过了在NVIDIA DGX系统上运行200次的训练。
表1。基于GAN的分割算法的Dice得分评估(均值/标准差)和17年BRATS最佳算法20,仅在“真实”数据上训练;真实+合成数据;只对合成数据进行训练,并根据10%的真实数据对模型进行微调。基于GAN的模型在训练过程中,对于包含通常的数据增强技术(裁剪、旋转、平移和弹性变形),在增强和非增强方面都进行了训练。所有模型都经过了200次迭代的训练才收敛。
在没有常规数据增强(裁剪,旋转,弹性变形,基于GAN的(无增强))的情况下,添加了合成数据的性能得到了很大的改善。然而,当添加常规的数据增强时(基于GAN的(无增强)),可以观察到性能的小幅提高,它也适用于Wang等人的20模型,该模型包含了通常的数据增强技术。
Wang等人的模型在全分辨率(256×256)下运行,结合了每个轴向/冠状/矢状视图的三个2D模型,而由于GPU内存限制,我们的模型和生成器只能运行一半的分辨率(128×128×54)。为了与BRATS挑战进行公平比较,我们对GAN生成的图像进行了两倍于生成分辨率的上采样,然而,在下/上采样过程中,非常小的肿瘤可能会丢失。如果使用基于GAN的模型,使用具有更多内存的可用GPU,可以观察到更好的性能。此外,我们认为生成的合成图像具有一半的分辨率,加上缺乏除了T1加权的图像序列以外的用于训练的图像序列,可能导致与使用通常的数据增强技术相比,分割性能的提高相对较小。我们仔细假设有更多可用的T2/Flair图像,这些序列的图像质量将会更好,因此更多模型和肿瘤类型的表现也会更好。
4.2 对匿名合成数据进行训练
我们还评估了基于GAN的仅在合成数据上的分割性能,其数量大于或等于真实数据的数量,但不包括任何原始数据。Dice得分计算如表1所示。当合成数据量等于原始训练集的80%时,我们基于GAN的模型和Wang等人的20模型的性能都达到了次优。然而,如果使用5倍以上的合成数据(仅)来训练这两个模型,并随机选择10%的的“真实”训练数据进行微调,就可以获得比在真实数据上进行训练更高的性能。在这种情况下,合成数据提供了一种形式的预训练,允许使用较少的“真实”数据来实现可比较的性能水平。
5 结论
在本文中,我们提出了一种生成算法,利用图像到图像转换GAN,从相应的分割mask生成合成的异常脑肿瘤多参数MRI图像。当通过改变输入标签映射来生成这样的合成图像时,可以引入更高水平的差异。这导致了在多个算法的分割性能的改进。此外,这些相同的算法可以在完全匿名的数据集上进行训练,允许共享训练数据。当与较小的、特定于机构的数据集相结合时,中等规模的组织就有机会训练成功的深度学习模型。
参考文献
Domingos, P.: A few useful things to know about machine learning. Commun. ACM 55(10), 78–87 (2012) ↩︎
Shin, H.-C., et al.: Deep convolutional neural networks for computer-aided detection: CNN architectures, dataset characteristics and transfer learning. IEEE Trans. Med. Imaging 35(5), 1285–1298 (2016) ↩︎
Goodfellow, I., et al.: Generative adversarial nets. In: Advances in Neural Information Processing Systems, pp. 2672–2680 (2014) ↩︎
Frangi, A.F., Tsaftaris, S.A., Prince, J.L.: Simulation and synthesis in medical imaging. IEEE Trans. Med. Imaging 37(3), 673–679 (2018) ↩︎ ↩︎
Chartsias, A., Joyce, T., Giuffrida, M.V., Tsaftaris, S.A.: Multimodal MR synthesis via modality-invariant latent representation. IEEE Trans. Med. Imaging 37(3), 803–814 (2018) ↩︎ ↩︎
Costa, P.: End-to-end adversarial retinal image synthesis. IEEE Trans. Med. Imaging 37(3), 781–791 (2018) ↩︎ ↩︎
Hu, Y., et al.: Intraoperative organ motion models with an ensemble of conditional generative adversarial networks. In: Descoteaux, M., et al. (eds.) MICCAI 2017 Part II. LNCS, vol. 10434, pp. 368–376. Springer, Cham (2017). https://doi.org/10.1007/978-3-319-66185-8_42 ↩︎
Mahapatra, D., Bozorgtabar, B., Hewavitharanage, S., Garnavi, R.: Image super resolution using generative adversarial networks and local saliency maps for retinal image analysis. In: Descoteaux, M., Maier-Hein, L., Franz, A., Jannin, P., Collins, D.L., Duchesne, S. (eds.) MICCAI 2017 Part III. LNCS, vol. 10435, pp. 382–390.Springer, Cham (2017). https://doi.org/10.1007/978-3-319-66179-7_44 ↩︎
Nie, D., et al.: Medical image synthesis with context-aware generative adversarial networks. In: Descoteaux, M., Maier-Hein, L., Franz, A., Jannin, P., Collins, D.L., Duchesne, S. (eds.) MICCAI 2017 Part III. LNCS, vol. 10435, pp. 417–425. Springer, Cham (2017). https://doi.org/10.1007/978-3-319-66179-7_48 ↩︎
Dar, S.U.H., Yurt, M., Karacan, L., Erdem, A., Erdem, E., C¸ ukur, T.: Image synthesis in multi-contrast MRI with conditional generative adversarial networks. arXiv preprint arXiv:1802.01221 (2018) ↩︎
Yang, D., et al.: Automatic liver segmentation using an adversarial image-toimage network. In: Descoteaux, M., Maier-Hein, L., Franz, A., Jannin, P., Collins, D.L., Duchesne, S. (eds.) MICCAI 2017 Part III. LNCS, vol. 10435, pp. 507–515.
Springer, Cham (2017). https://doi.org/10.1007/978-3-319-66179-7_58
s.) MICCAI 2017 Part III. LNCS, vol. 10435, pp. 507–515.
Springer, Cham (2017). https://doi.org/10.1007/978-3-319-66179-7_58 ↩︎Zhang, Y., Yang, L., Chen, J., Fredericksen, M., Hughes, D.P., Chen, D.Z.: Deep adversarial networks for biomedical image segmentation utilizing unannotated images. In: Descoteaux, M., Maier-Hein, L., Franz, A., Jannin, P., Collins, D.L., Duchesne, S. (eds.) MICCAI 2017 Part III. LNCS, vol. 10435, pp. 408–416. Springer, Cham (2017). https://doi.org/10.1007/978-3-319-66179-7_47 ↩︎
Frid-Adar, M., Klang, E., Amitai, M., Goldberger, J., Greenspan, H.: Synthetic data augmentation using GAN for improved liver lesion classification. In: IEEE International Symposium on Biomedical Imaging (ISBI) (2018) ↩︎
Christopher, G.S., et al.: A large-scale comparison of cortical thickness and volume methods for measuring Alzheimer’s disease severity. NeuroImage: Clin. 11, 802–812(2016) ↩︎
Ashburner, J., Friston, K.J.: Unified segmentation. Neuroimage 26(3), 839–851(2005) ↩︎
Tustison, N.J., et al.: Large-scale evaluation of ANTS and FreeSurfer cortical thickness measurements. Neuroimage 99, 166–179 (2014) ↩︎
Menze, B.H., et al.: The multimodal brain tumor image segmentation benchmark (BRATS). IEEE Trans. Med. Imaging 34(10), 1993–2024 (2015) ↩︎
Iglesias, J.E., Liu, C.-Y., Thompson, P.M., Tu, Z.: Robust brain extraction across datasets and comparison with publicly available methods. IEEE Trans. Med. Imaging 30(9), 1617–1634 (2011) ↩︎
Isola, P., Zhu, J.-Y., Zhou, T., Efros, A.A.: Image-to-image translation with conditional adversarial networks. In: The IEEE Conference on Computer Vision and Pattern Recognition (CVPR), July 2017 ↩︎
Wang, G., Li, W., Ourselin, S., Vercauteren, T.: Automatic brain tumor segmentation using cascaded anisotropic convolutional neural networks. arXiv preprint arXiv:1709.00382 (2017) ↩︎ ↩︎ ↩︎ ↩︎ ↩︎
Milletari, F., Navab, N., Ahmadi, S.-A.: V-net: fully convolutional neural networks for volumetric medical image segmentation. In: 2016 Fourth International Conference on 3D Vision (3DV), pp. 565–571. IEEE (2016) ↩︎
Carlini, N., Liu, C., Kos, J., Erlingsson, ´U., Song, D.: The secret sharer: measuring unintended neural network memorization & extracting secrets. arXiv preprint arXiv:1802.08232 (2018) ↩︎