医学图像处理入门知识 | 格式DICOM,MHD+RAW | 坐标系 | ITK-SNAP | 重采样
医学图像处理入门知识
-
- 引言
- 常见的医学图像格式
-
- DICOM
- mhd+raw
- 坐标系
-
- 世界坐标体系
- 解剖学坐标体系
- 图像坐标体系
- 这三个坐标体系有什么关系呢?怎样转换呢?
- ITK-SNAP工具使用
-
- 基本功能
- 调整对比度
- 重采样
- 项目思路
- 总结
引言
最近做了一个医学图像的项目,将这块好好的整理一下,给入门的同学提供点帮助,让大家能够将时间花在核心算法上,缩短准备工作的时间。
文章内容较多,本人力求将每个地方讲明白并没有错误,不过文笔有限,可能还是有错误或者表述上不够严谨。如果有错误或者不妥的地方,还请指正,衷心感谢。
本文参考博客内容较多较杂,以至于不知道参考了哪些文章了,如果有侵权的地方,麻烦及时和我联系,第一时间解决。
文章主要介绍医学图像的特点,基本的医学图像文件格式。具体每个知识点,可能本人讲的不会特别深,目的知识希望读者通过学习该内容,可以快速上手项目,具体详细内容可在网上搜索。如DICOM文件TAG就有很多,网上都可以搜到的。
常见的医学图像格式
常见的医学图像格式有六种,DICOM(医疗中的数字图像和通信),NIFTI(神经影像学信息技术计划),PAR/REC(菲利普MIR扫描格式),ANALYZE(Mayo医疗成像)),NRRD(近乎原始光栅数据),MINC格式。
Analyze格式:Analyze格式储存的每组数据组包含2个文件,一个为头文件,扩展名为.hdr,包含图像的元数据;一个为数据文件,其扩展名为.img,包含二进制的图像资料。
NIFTI格式:标准NIFTI图像的扩展名是.nii,也包含了头文件及图像资料。由于NIFTI格式和Analyze格式的关系,因此NIFTI格式也可使用独立的图像文件(.img)和头文件(.hdr)。
以下主要介绍常见的dicom、raw+mhd格式。
DICOM
DICOM(Digital Imaging and Communications in Medicine)即医学数字成像和通信,是医学图像和相关信息的国际标准。其是目前应用最为广泛的医学影像格式,常见的CT,核磁共振,心血管成像等大多采用的是DICOM格式的存储。
简单的说,一个DICOM数据中包含了两部分:
- 图像信息:检查部位的影像信息,如脑部、胸部等。(我们通常要处理的像素信息)
- 患者信息:带着一系列编号的TAG,如姓名、性别、年龄等。
DICOM数据是怎么产生的呢?
CT或核磁共振等探测器围绕人体的某一部位作一个接一个的断面扫描,得到多层图像,再把一层层的图像在z轴上堆叠起来就可以形成三维图像,如下图所示。因此通常DICOM把每一层图像都作为一个独立的文件,这些文件用数字命名从而反映相对应的图像层数(在不同的系统有一定差异)。
通常使用Python中的pydicom和SimpleITK库来读取DICOM图像,这块代码网上有很多,这里不在展示。
注
由于DICOM把每层图像都存储为独立文件,这会导致产生大量较小的数字文件,从而堵塞文件系统,降低分析速度。而且DICOM数据也比较庞大,所以数据分析前往往要把DICOM格式转化为其他分析格式。
mhd+raw
mhd+raw格式是常见的一种医学图像格式,每一个病人的数据包含一个mhd文件和一个同名的raw文件(一一一对应),使用Python的SimpleITK库即可读取。
- mhd(数据头部信息):存放数据的非图像信息,如图像大小、切片大小、像素大小等。
- raw(未加工的数据):存储病人的图像信息,往往是三维体数据。(可以理解为将该病人不同的dicom切片图像都叠到一起,形成了一个三维图像,也就是我们通常处理的数据)。
熟悉完基本格式,我们打开一个mhd文件看一下。可以看到mhd和zraw(压缩的raw,本质上和raw相同)是同名且放在同一目录下。

那怎么查看mhd的头部信息呢?
将mhd文件用记事本打开即可。mhd中的各项参数十分重要,有一些同学用很多套数据训练网络,却发现无论怎么调参,网络学习效果都不好。可能的原因就是数据之间的一些基本信息不相同,如spacing不同,那么就要先重采样,然后再训练,后文也会提到该内容。
参数解释
- NDims :图片的维度
- BinaryData:是否用二值存储
- TransformMatrix:x, y,z方向(结合向量理解,如 1 0 0表示的就是 x 轴方向向量)
- Offset:原点坐标
- ElementSpacing:一个体素(图像像素)在xyz(真实世界CT采样)方向上的大小(也是像素间隔大小),其中一般xy对应的spacing是相同的
- DimSize:每张slice的二维大小与张数(可理解为图像分辨率,并不是真实大小)
- ElementType:MET_SHORT类型,数值有正负
- ElementDataFile:头文件对应的原始数据
只看到上面参数,是不是懵懵的,不慌,下面会讲到的~
坐标系
顺着思路思考,既然已经知道了图像是怎么存储的,我们不禁想到,这些像素按照什么坐标系来存储的呢?
世界坐标体系
如上图左所示,
世界坐标体系是典型的笛卡尔坐标体系,整个模型只有一个世界坐标来定义位置与方向,在这个坐标系中,每个模型都(如核磁扫描器,病人)有它自身的坐标体系,但是只存在一个世界坐标体系来定义模型的位置与方向。也就是说,每个模型都可以被世界坐标系表示,可以理解为绝对坐标。
解剖学坐标体系
如上图中所示,
这个解剖学坐标体系是一个连续的三维空间,在这个空间中,图像被采样。如上图中所示,这个坐标系包括3个位面(解剖轴):
- 横断面:与地面平行,上下分离人体(绿色)
- 矢状面:垂直地面,左右分离人体(红色)
- 冠状面:垂直地面,前后分离人体(紫色)
本文以 LPS 为坐标轴。
不同的医学应用软件运用不同的3D基本定义,下面这些基本上是最常用的:
- LPS (Left,Posterior,Superior):Dicom与ITK工具包上用这个
- from right to left
- from anterior to posterior
- from inferior to superior
- RAS (Right,Anterior,Superior):3D Slicer上用这个
- from left to right
- from posterior to anterior
- from inferior to superior
从根本上来说,这两种坐标轴是等价使用的,有着相同的逻辑。
图像坐标体系
如上图右所示,
医学扫描仪器创建了规则的点和网格的矩形数组,它的原点在左上角,向右为 i 轴正方向,向下为 j 轴正方向,向后为 k 轴正方向。除每个立体像素voxel (i,j, k)的强度值外,这个解剖学坐标的原点以及间距也被保存下来。(体素:立体像素,本质上还是像素,换个名字而已)
这个原点代表着第一个voxel (0,0,0) 体素在解剖学坐标体系中的位置如(100mm,50mm,-25mm)。这个间距指定立体像素在各个坐标轴的实际距离间隔例如(1.5mm,0.5mm,0.5mm)。在下面的2D例子中显示了原点与间距的意义,3D的类推即可。

也就是说,通过解剖学坐标系我们获得了一个3D图像,这个3D图像中的坐标原点(原点是对应图像坐标系说的)在解剖学坐标系并不是原点,而是对应着一个坐标,在解剖学坐标系中,单位基本都是mm。
利用原点和间距,每个立体像素在解剖学坐标体系中的对应位置都能够被计算出来
那有人就问了,为什么不直接在解剖学坐标系中获取呢?因为解剖学坐标系中的单位都是1 mm,你写程序的时候怎么去获取这个1mm呢,而用图像坐标系中的坐标,不用考虑单位,直接根据索引获取就可以。
是不是突然悟了?可以这样理解,图像坐标体系是虚拟的,为了方便处理才有的。
这三个坐标体系有什么关系呢?怎样转换呢?
图像坐标体系与解剖学坐标体系的关系
DICOM的Orientation和MHD的TransformMatrix 表示图像坐标与解剖学坐标体系对应坐标的夹角余弦值。
图像坐标体系与世界坐标体系的关系
原点(origin)和间距(spacing)这两个参数将世界坐标转换为相应的图像坐标,即求出结节中心在图像中的位置。
origin表示图像坐标系原点在世界坐标系中的位置单位mm,
spacing表示每个像素的实际距离间隔,单位也是mm
所以结节中心坐标减去origin再除以spacing,就是对应像素在图像坐标系中的位置。
// 世界坐标转换到图像中的坐标
def worldToVoxelCoord(worldCoord, origin, spacing):
stretchedVoxelCoord = np.absolute(worldCoord - origin)
voxelCoord = stretchedVoxelCoord / spacing
return voxelCoord
不过ITK-SNAP软件中,图像坐标(1,1,1)对应世界坐标(0,0,0),也就是说多了一个坐标,这个注意一下就行了,其实影响不大。

ITK-SNAP工具使用
工欲善其事,必先利其器
目前我比较常用的医学图像处理软件,就是ITK-SNP,可以处理常见的DICOM、MHD等等。该软件在网上也很容易下载到。
基本功能
下面就简单介绍一下 ITK-SNP的常用功能,
软件打开界面如下,不过我一般常用的是,直接将文件拖到软件上打开。
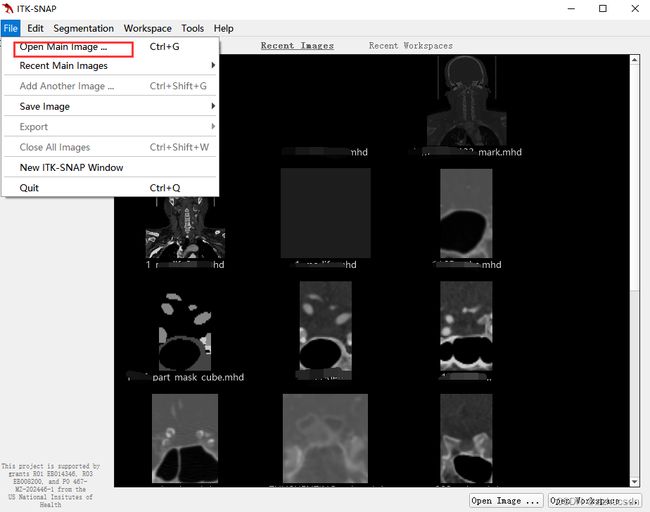
这里我打开一套数据,为展示一下基本的功能。打开后软件,不断地尝试一下,就知道的差不多了。
那怎么显示标签图呢?也很简单,直接将“标签图文件”拖到“已经显示的原图”上,会出现一个弹窗,选择"Load as Segmentation"即可。ITK-SNAP不仅可以显示标签图,可以修改标签图,添加标签图等等。
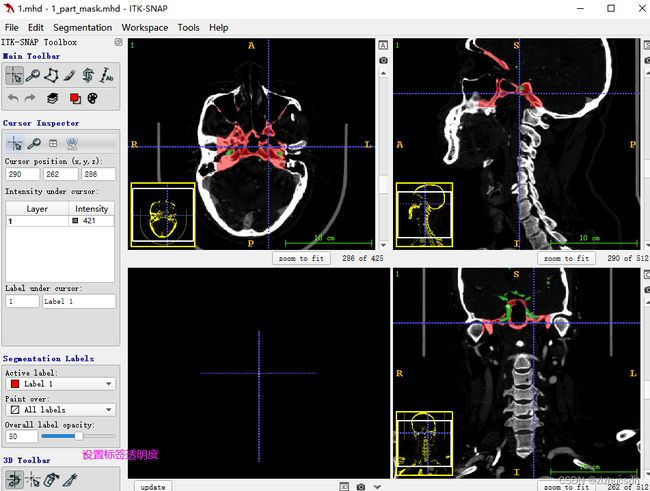
打开Tool -> Layer Inspector,这里也有一些常用的功能,如调整对比度,查看基本信息等。
调整对比度
为什么要调整对比度呢?
因为当我们打开一套数据时,整幅CT图像中所包含的信息可能过多,当需要观察某种部位时,可以调节对比度来显示局部细节。
对比度的调整,实际上是窗宽(Window Width)和窗位(Window Level)的调整。通过调整窗宽窗位将要显示的数据,控制在某一像素值区间范围内。如我将窗宽设置为1000,窗位设置为500,其显示的是[0, 1000]的CT值,这样骨头就被很好地显示出来了,如下图所示。

窗宽窗位是怎么计算显示的CT值区间[Minimum, Maximun]的呢?
- Minimum: 窗宽 - (窗位 / 2)
- Maximum:窗宽 + (窗位 / 2)
通过调整窗宽窗位,不仅可以让我们人眼看的更清楚。在我们训练网络时,当研究对象是某一特定组织时,那么调整到一个合适的窗宽窗位,让该组织显示地更加明显、边界分明,对网络训练也有很大的帮助。
不过一些常见的器官组织,如骨头、血管、肺部等,网上都可以搜到窗宽窗位对应的设置值。
重采样
概念
在医疗图像中,重采样是指将医疗图像中大小不同的体素归一化到相同的大小。体素是体积元素(Volume Pixel)的简称,一张3D医疗图像可以看成是由若干个体素构成的。说的通俗点,就是将每套图像的spacing调整到一致大小。
在上文我们也简单提到过体素的概念。不妨试着这样理解,在图像坐标体系中,每个像素中间都隔着spcing大小的间距,那如果将每个spacing / 2 分到其两侧的像素上,这样每个像素在其x,y,z三个方向上,均会分到 spacing / 2大小的空间,从而像素不在是一个点,而是一个边长为{spacing[x], spcing[y], spcing[z]}的小长方体,这就是体素。(spacing本身是含有三个元素的,分别在x,y,z上)
顺着这个思路继续走,那是不是知道了spacing,我们就可以求出图像的物理大小了(实际大小),很显然,实际大小 = 图像分辨率(DimSize) 乘 Spacing。
如上文展示的一个mhd文件的信息,
ElementSpacing = 0.58593799999999996 0.58593799999999996 0.80000000000000004
DimSize = 512 512 425
那么该图像的实际大小为DimSize * ElementSpacing,(对应位置相乘即可)。
ok,让我们再回到重采样这里,不禁要问一声,为什么要重采样呢?
我不重采样,不是也可以训练吗,还是一样可以用图像坐标系的坐标训练。这样当然是不行的,因为spacing大小不一致,而网络的CNN又无法理解体素间距,就会导致学习效果很差。举个例子,假如我们有100套数据,每一套数据的spacing都不相同,甚至差别很大,但是呢在网络眼里都是统一的图像坐标系,即两个像素点之间距离是相等,试问如果不重采样的话,怎么能学习好呢?
重采样,即将spacing调整到一致大小。
图像的实际大小是不会变化,当spcing改变时,图像分辨率(size)必然也会发生改变。那么,自然会想到,当spacing变大时,图像分辨率会变小;当spacing减小时,图像分辨率会变大。当分辨率变大时,又会出现另一个问题了,这多出来的部分,怎么添加上去呢?对,插值。
插值就会涉及到一些插入算法的选择,如近邻插值、线性插值等,不过这些在Python的SimpleITK库中已经写好了,直接调用即可。
好啦,这块内容差不多到这里了,下面给出.mhd文件重采样的代码。
def img_resmaple(ori_img_file, target_img_file, new_spacing, resamplemethod=sitk.sitkNearestNeighbor):
"""
@param ori_img_file: 原始的itk图像路径,一般为.mhd
@param target_img_file: 保存路径
@param new_spacing: 目标重采样的spacing,如[0.585938, 0.585938, 0.4]
@param resamplemethod: itk插值⽅法: sitk.sitkLinear-线性、sitk.sitkNearestNeighbor-最近邻、sitk.sitkBSpline等,SimpleITK源码中会有各种插值的方法,直接复制调用即可
"""
data = sitk.ReadImage(ori_img_file) # 根据路径读取mhd文件
original_spacing = data.GetSpacing() # 获取图像重采样前的spacing
original_size = data.GetSize() # 获取图像重采样前的分辨率
# 有原始图像size和spacing得到真实图像大小,用其除以新的spacing,得到变化后新的size
new_shape = [
int(np.round(original_spacing[0] * original_size[0] / new_spacing[0])),
int(np.round(original_spacing[1] * original_size[1] / new_spacing[1])),
int(np.round(original_spacing[2] * original_size[2] / new_spacing[2])),
]
print("处理后新的分辨率:{}".format(new_shape))
# 重采样构造器
resample = sitk.ResampleImageFilter()
resample.SetOutputSpacing(new_spacing) # 设置新的spacing
resample.SetOutputOrigin(data.GetOrigin()) # 原点坐标没有变,所以还用之前的就可以了
resample.SetOutputDirection(data.GetDirection()) # 方向也未变
resample.SetSize(new_shape) # 分辨率发生改变
resample.SetInterpolator(resamplemethod) # 插值算法
data = resample.Execute(data) # 执行操作
sitk.WriteImage(data, target_img_file) # 将处理后的数据,保存到一个新的mhdw文件中
项目思路
医学图像处理的项目也有很多类型,如插值、分割、生成等等。每种项目处理的步骤可能也有些不同,所以下面只是列一些注意点,算作一个小提醒吧。
- 熟悉研究部位的结构,观察数据的标签是否准确
- 观察数据的原点、间距、方向等是否一致,是否需要重采样
- 针对研究部位,调整到合适的窗宽窗位
- 由于图像本身CT值都很大,训练网络前需归一化
- 训练网络、优化等
总结
行笔于此,文已将尽。
本人也是刚学不久,文中存在的问题,还希望各位同仁多多指教。