【实战】霸榜各大医学分割挑战赛的Transformer架构--nnFormer
文章目录
-
- 安装
- 下载以及预处理实验数据
-
- 预处理数据
- 修改源代码错误
- train
- test
简介:我们介绍了nnFormer(not-another transFormer),一种用于3D医学图像分割的 transformer。

nnFormer 不仅使用了卷积和自注意力的结合,还引入了基于局部和全局体积的自注意机制来学习体积表示。
此外,nnFormer 建议使用跳跃注意来取代U-Net类体系结构中跳跃连接中的传统操作。
实验表明,在三个公共数据集上,nnFormer 性能显著。与 nnUNet 相比,nnFormer 产生的HD95显著降低,DSC结果也具有可比性。此外,nnFormer 和 nnUNet 在模型融合中是高度互补的。nnFormer的代码也是基于nnUNet改的。
因此,只要用过 nnUNet, 这部分代码相对顺畅
不用写代码神器!教你用4行命令轻松使用nnUNet训练自己的医学图像分割模型
本教程难度:
没使用过nnUNet: ⭐️⭐️⭐️⭐️
使用过nnUNet: ⭐️⭐️
难点在于安装环境,下载数据,预处理数据,训练和测试都是一句命令就搞定。前期工作要做好。
nnFormer 论文下载
nnFormer github
安装
1.官方系统版本
Ubuntu 18.01、Python 3.6、PyTorch 1.8.1 和 CUDA 10.1 。有关软件包和版本号的完整列表,请参阅 Conda 环境文件 environment.yml。
- 安装步骤
建议使用 conda 包管理器安装所需的包
git clone https://github.com/282857341/nnFormer.git
(默认下载位置不同,下载下来后找不到百度一下)
cd nnFormer (将整个文件剪切到你日常的项目所在文件夹,方便使用)
conda env create -f environment.yml (这一步会创建叫nnFormer的conda环境)
source activate nnFormer
pip install -e .
这一步安装,如果网络不好,多半是会出错的。是在不行的话,建议手动创建一个环境conda create -n nnFormer python=3.6, 并手动安装 environment.yml 文件里面需要的包。
下载以及预处理实验数据
官方使用了三个数据集,每个数据集都有各自的model,train, inference.
所以实验的时候一定要指定使用的数据集。
- ACDC数据集
- Synapse 多器官 CT 数据集
- Brain_tumor数据集
本教程使用的是Brain_tumor数据集,下载的时候是task01_Braintomor,重命名为 Task03_tumor(该论文中task03才是brain tumor, 要对应。)

下载不下来的同学可以去我网盘下载:
链接: https://pan.baidu.com/s/1TChc4yXZjPlv9ApqS-OHkQ 提取码: c0mj
受网盘上传限制,一共3个压缩包,把它解压后放在Task03_tumor文件夹下。包含以下内容

预处理数据
我们要像nnUNet那样,数据有严格的格式。
首先创建以下文件夹

其中DATASET随便你放置在哪里,为了方便,我放在了nnFormer里面, 图片上标注了文件夹级别,不要搞错了哦。本实验第四级别只需要Task03_tumor,把刚才下好的文件夹放进来。
注意:下载的数据有一个dataset.json,这个里面的训练集和测试集同nnFormer不一样。你可以按照现在的划分进行下一步,但是这个测试集里的数据没有ground truth, 在做测试的时候就无法求dice. 如果你想要知道测试集的性能,就按照nnFormer的dataset.json重新划分imagesTr, imagesTs, labelsTr, labelsTs. nnFormer是把训练集再次划分为训练集和测试集,因此他的测试集才有ground truth。(啰嗦这么多,不知道讲清楚么)
初始数据有了,下面进行预处理
- 打开终端
cd nnFormerconda activate nnFormernnFormer_convert_decathlon_task -i ../DATASET/nnFormer_raw/nnFormer_raw_data/Task03_tumor
这一步会在数据目录下新建一个 Task003_tumor 的文件夹,并且把多模态数据转成了4个单模态数据,同nnUNet要的数据格式一样nnFormer_plan_and_preprocess -t 3
以上预处理数据就完了
下面进入正式实战阶段
修改源代码错误
下载的代码有几处错误需要修改
nnFormer/nnformer/run/default_configuration.py文件
有一个else位置不对nnFormer/nnformer/run/run_train.py文件
import numpy as no 改成 npnnFormer/nnformer/training/network_training/nnFormerTrainerV2_nnformer_tumor.py文件
self.load_pretrain_weight设置成False
train
训练一共有2种方法
不管使用哪种方法,先 切换到下面路径
cd nnFormer
bash train_inference.sh -c 0 -n nnformer_tumor -t 3
使用该命令会执行训练和测试

ps: 确保之前已经设置好了变量
export nnFormer_raw_data_base='/xxxxxxxx/nnFormer/DATASET/nnFormer_raw'
export nnFormer_preprocessed='/xxxxxx/nnFormer/DATASET/nnFormer_preprocessed'
export RESULTS_FOLDER='/xxxxxxx/nnFormer/DATASET/nnFormer_trained_models'
环境变量的设置见之前的文章nnunet
训练好的模型默认保存在
xxxx/nnFormer/DATASET/nnFormer_trained_models/nnFormer/3d_fullres/Task003_tumor/nnFormerTrainerV2_nnformer_tumor__nnFormerPlansv2.1
- 2 使用
nnFormer_train
打开 train_inference.sh 文件, 可以看到在predict部分,实际调用的就是 nnFormer_train 功能, 因此我们可以直接调用这个功能做训练.
eg. CUDA_VISIBLE_DEVICES=1 nnFormer_train 3d_fullres nnFormerTrainerV2_nnformer_tumor 3 0
- 第一个参数: network
- 第二个参数: network_trainer
- 第三个参数: task, can be task name or task id
- 第四个参数: fold,五折交叉,fold可以是具体的第x折(0-4),如果是5折都要做,fold=5,or all
test
测试一共有2种方法
不管使用哪种方法,先 切换到下面路径
cd nnFormer
- 1 使用
bash train_inference.sh
该文件在下载的 nnFormer主目录下面,运行前需要打开文件,把文件夹地址改成自己的地址,
[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-quJrtIVU-1655445035770)(imgs/20220614-150345.png)]
我们这里不需要训练, 只推理
bash train_inference.sh -c 0 -n nnformer_tumor -t 3
注意: 如果不训练,使用此命令需要手动把 train 部分的代码注释掉.我不懂代码里的 getopts设置的参数怎么使用,在命令行怎么设置都关不掉训练.所以采用这种笨方法.
-
c: stands for the index of your cuda device
-
n: denotes the suffix of the trainer located at nnFormer/nnformer/training/network_training/
-
t: denotes the task index
-
2 使用
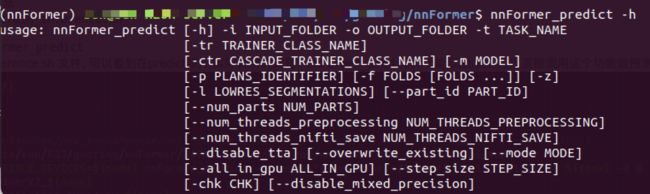
nnFormer_predict
打开 train_inference.sh 文件, 可以看到在predict部分,实际调用的就是 nnFormer_predict功能, 因此我们可以直接调用这个功能做预测.

eg: nnFormer_predict -i xxx/nnFormer/DATASET/nnFormer_raw/nnFormer_raw_data/Task003_tumor/imagesTs -o xxx/nnFormer/DATASET/nnFormer_raw/nnFormer_raw_data/Task003_tumor/inferTS/nnformer_tumor -t 3 -m 3d_fullres -f 0 -chk model_best -tr nnFormerTrainerV2_nnformer_tumor
正确运行后,会出现如下内容
然后就可以在OUTPUT_FOLDER文件夹下查看分割结果了.
由于测试数据没有ground truth, 因此只能手动查看分割性能.
ps: 官方的数据是有测试集标签的,需要的可以找一下. 如果有标签, 可以使用下面命令求dice
python nnformer/inference_tumor.py nnformer_tumor
文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连
![]()