机器学习(6):聚类算法:K-MEANS算法、DBSCAN算法
目录
一、聚类算法原理
二、K-MEANS算法
2.1 K-MEANS算法基本介绍
2.2 K-MEANS算法过程
三、DBSCAN算法定义
聚类评估:轮廓系数(Silhouette Coefficient )
四、sklearn中的聚类
4.1 K-MEANS
参数:
属性:
方法:
4.2 DBSCAN算法
一、聚类算法原理
对于"监督学习"(supervised learning),其训练样本是带有标记信息的,并且监督学习的目的是:对带有标记的数据集进行模型学习,从而便于对新的样本进行分类。而在“无监督学习”(unsupervised learning)中,训练样本的标记信息是未知的,目标是通过对无标记训练样本的学习来揭示数据的内在性质及规律,为进一步的数据分析提供基础。对于无监督学习,应用最广的便是"聚类"(clustering)。
二、K-MEANS算法
2.1 K-MEANS算法基本介绍
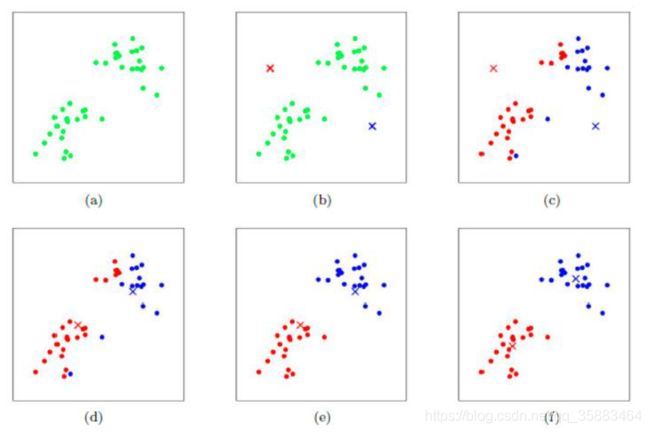
kmeans算法又名k均值算法。其算法思想大致为:先从样本集中随机选取 kk 个样本作为簇中心,并计算所有样本与这 kk 个“簇中心”的距离,对于每一个样本,将其划分到与其距离最近的“簇中心”所在的簇中,对于新的簇计算各个簇的新的“簇中心”。
根据以上描述,我们大致可以猜测到实现kmeans算法的主要三点:
(1)簇个数 kk 的选择
(2)各个样本点到“簇中心”的距离
(3)根据新划分的簇,更新“簇中心”
基本概念:
要得到簇的个数,需要指定K值
距离的度量:常用欧几里得距离和余弦相似度(先标准化)
优化目标:
2.2 K-MEANS算法过程
工作流程:
优势:
简单,快速,适合常规数据集
劣势:
复杂度与样本呈线性关系
很难发现任意形状的簇
例如:
三、DBSCAN算法定义
DBSCAN(Density-Based Spatial Clustering of Applications with Noise,具有噪声的基于密度的聚类方法)是一种很典型的密度聚类算法,和K-Means,BIRCH这些一般只适用于凸样本集的聚类相比,DBSCAN既可以适用于凸样本集,也可以适用于非凸样本集。下面我们就对DBSCAN算法的原理做一个总结。
DBSCAN是基于一组邻域来描述样本集的紧密程度的,参数(ϵ, MinPts)用来描述邻域的样本分布紧密程度。其中,ϵ描述了某一样本的邻域距离阈值,MinPts描述了某一样本的距离为ϵ的邻域中样本个数的阈值。
几个核心概念:
参数选择:
MinPts:k-距离中k的值,一般取的小一些,多次尝试。
半径ϵ,可以根据K距离来设定:找突变点。
K距离:给定数据集P={p(i); i=0,1,…n},计算点P(i)到集合D的子集S中所有点之间的距离,距离按照从小到大的顺序排序,d(k)就被称为k-距离。
可视化:https://www.naftaliharris.com/blog/visualizing-dbscan-clustering/
优势:
不需要指定簇个数
擅长找到离群点(检测任务)
可以发现任意形状的簇
两个参数就够了
劣势:
高维数据有些困难(可以做降维)
Sklearn中效率很慢(数据削减策略)
参数难以选择(参数对结果的影响非常大)
和传统的K-Means算法相比,DBSCAN最大的不同就是不需要输入类别数k,当然它最大的优势是可以发现任意形状的聚类簇,而不是像K-Means,一般仅仅使用于凸的样本集聚类。同时它在聚类的同时还可以找出异常点,这点和BIRCH算法类似。
那么我们什么时候需要用DBSCAN来聚类呢?一般来说,如果数据集是稠密的,并且数据集不是凸的,那么用DBSCAN会比K-Means聚类效果好很多。如果数据集不是稠密的,则不推荐用DBSCAN来聚类。
下面对DBSCAN算法的优缺点做一个总结。
DBSCAN的主要优点有:
1) 可以对任意形状的稠密数据集进行聚类,相对的,K-Means之类的聚类算法一般只适用于凸数据集。
2) 可以在聚类的同时发现异常点,对数据集中的异常点不敏感。
3) 聚类结果没有偏倚,相对的,K-Means之类的聚类算法初始值对聚类结果有很大影响。
DBSCAN的主要缺点有:
1)如果样本集的密度不均匀、聚类间距差相差很大时,聚类质量较差,这时用DBSCAN聚类一般不适合。
2) 如果样本集较大时,聚类收敛时间较长,此时可以对搜索最近邻时建立的KD树或者球树进行规模限制来改进。
3) 调参相对于传统的K-Means之类的聚类算法稍复杂,主要需要对距离阈值ϵϵ,邻域样本数阈值MinPts联合调参,不同的参数组合对最后的聚类效果有较大影响。
聚类评估:轮廓系数(Silhouette Coefficient )
- 计算样本i到同簇其他样本的平均距离ai。ai 越小,说明样本i越应该被聚类到该簇。将ai 称为样本i的簇内不相似度。
- 计算样本i到其他某簇Cj 的所有样本的平均距离bij,称为样本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:bi =min{bi1, bi2, ..., bik}
- si接近1,则说明样本i聚类合理
- si接近-1,则说明样本i更应该分类到另外的簇
- 若si 近似为0,则说明样本i在两个簇的边界上
四、sklearn中的聚类
4.1 K-MEANS
from sklearn.cluster import KMeans
km = KMeans(n_clusters=3).fit(X) 3个簇,训练模型参数:
(1)n_clusters:要分成的簇数也是要生成的质心数
类型:整数型(int)
默认值:8
(2)init:初始化质心,
类型:{‘k-means++’, ‘random’ or an ndarray}
默认值:采用k-means++(一种生成初始质心的算法)
(3)n_init: 设置选择质心种子次数,默认为10次。返回质心最好的一次结果(好是指计算时长短)
类型:整数型(int)
默认值:10
目的:每一次算法运行时开始的centroid seeds是随机生成的, 这样得到的结果也可能有好有坏. 所以要运行算法n_init次, 取其中最好的。
(4)max_iter:每次迭代的最大次数
类型:整型(int)
默认值:300
(5)tol: 容忍的最小误差,当误差小于tol就会退出迭代(算法中会依赖数据本身)
类型:浮点型(float)
默认值:le-4(0.0001)
(6)precompute_distances: 这个参数会在空间和时间之间做权衡,如果是True 会把整个距离矩阵都放到内存中,auto 会默认在数据样本大于featurs*samples 的数量大于12e6 的时候False,False时核心实现的方法是利用Cpython 来实现的
类型:布尔型(auto,True,False)
默认值:“auto”
(7)verbose:是否输出详细信息
类型:布尔型(True,False)
默认值:False
(8)random_state: 随机生成器的种子 ,和初始化中心有关
类型:整型或numpy(RandomState, optional)
默认值:None
(9)copy_x:bool 在scikit-learn 很多接口中都会有这个参数的,就是是否对输入数据继续copy 操作,以便不修改用户的输入数据。这个要理解Python 的内存机制才会比较清楚。
类型:布尔型(boolean, optional)
默认值:True
(10)n_jobs: 使用进程的数量,与电脑的CPU有关
类型:整数型(int)
默认值:1
属性:
cluster_centers_:向量,[n_clusters, n_features] (聚类中心的坐标)
Labels_: 每个点的分类
inertia_:float,每个点到其簇的质心的距离之和。
n_iter_ : int,运行的迭代次数。
方法:
fit(X[,y]):
计算k-means聚类。
fit_predictt(X[,y]):
计算簇质心并给每个样本预测类别。
get_params([deep]):
取得估计器的参数。
predict(X):predict(X)
给每个样本估计最接近的簇。
score(X[,y]): 计算得分。
set_params(**params): 为这个估计器手动设定参数。
transform(X[,y]): 将X转换为群集距离空间。
4.2 DBSCAN算法
from sklearn.cluster import DBSCAN
db = DBSCAN(eps=10, min_samples=2).fit(X) 参数
1)eps: DBSCAN算法参数,即我们的ϵϵ-邻域的距离阈值,和样本距离超过ϵϵ的样本点不在ϵϵ-邻域内。默认值是0.5.一般需要通过在多组值里面选择一个合适的阈值。eps过大,则更多的点会落在核心对象的ϵϵ-邻域,此时我们的类别数可能会减少, 本来不应该是一类的样本也会被划为一类。反之则类别数可能会增大,本来是一类的样本却被划分开。
2)min_samples: DBSCAN算法参数,即样本点要成为核心对象所需要的ϵϵ-邻域的样本数阈值。默认值是5. 一般需要通过在多组值里面选择一个合适的阈值。通常和eps一起调参。在eps一定的情况下,min_samples过大,则核心对象会过少,此时簇内部分本来是一类的样本可能会被标为噪音点,类别数也会变多。反之min_samples过小的话,则会产生大量的核心对象,可能会导致类别数过少。
3)metric:最近邻距离度量参数。可以使用的距离度量较多,一般来说DBSCAN使用默认的欧式距离就可以满足我们的需求。
以上就是DBSCAN类的主要参数介绍,其实需要调参的就是两个参数eps和min_samples,这两个值的组合对最终的聚类效果有很大的影响。