机器学习之降维算法:主成分分析(PCA)
文章目录
- 1. PCA理论
-
- 1.1 高维样本均值和方差表示
- 1.2 最大投影方差&最小重构距离
- 2. PCA实操
-
- 2.1 重要参数n_components
-
- 2.1.1 高维数据的可视化特征分布
- 2.1.2 其他方式选取n_components
- 2.2 参数svd_solver&random_state
- 2.3 重要属性components_
- 2.4 重要接口inverse_transform
- 2.5 用PCA做噪音过滤
- 2.6 实战:手写数据集降维
1. PCA理论
解决过拟合问题常见思路:增加训练处理数据、加入正则化项、对特征进行降维。
降维方法:直接进行特征选择、线性降维(PCA,MDS)、非线性降维(流形)。
1.1 高维样本均值和方差表示
给定数据Data矩阵, X = ( x 1 , x 2 , . . . , x N ) N ∗ p T X=(x_1,x_2,...,x_N)^{T}_{N*p} X=(x1,x2,...,xN)N∗pT。
样本特征均值: X ‾ p ∗ 1 = 1 N ∑ i = 1 N x i \overline{X}_{p*1}= \frac{1}{N}\sum_{i=1}^Nx_i Xp∗1=N1i=1∑Nxi
样本特征方差: S p ∗ p = 1 N ∑ i = 1 N ( x i − X ‾ ) ( x i − X ‾ ) T S_{p*p}=\frac{1}{N}\sum_{i=1}^N(x_i-\overline{X})(x_i-\overline{X})^{T} Sp∗p=N1i=1∑N(xi−X)(xi−X)T
则有高维样本均值和方差的表示:
X ‾ p ∗ 1 = 1 N ∑ i = 1 N x i \overline{X}_{p*1}= \frac{1}{N}\sum_{i=1}^Nx_i Xp∗1=N1∑i=1Nxi
= 1 N ( x 1 , x 2 , . . . , x N ) ( 1 . . . 1 ) N ∗ 1 = \frac{1}{N}(x_1,x_2,...,x_N)\begin{pmatrix} 1 \\ ...\\ 1 \\ \end{pmatrix}_{N*1} =N1(x1,x2,...,xN)⎝⎛1...1⎠⎞N∗1
= 1 N X T 1 N ∗ 1 = \frac{1}{N}X^{T}1_{N*1} =N1XT1N∗1
S p ∗ p = 1 N ∑ i = 1 N ( x i − X ‾ ) ( x i − X ‾ ) T S_{p*p}=\frac{1}{N}\sum_{i=1}^N(x_i-\overline{X})(x_i-\overline{X})^{T} Sp∗p=N1∑i=1N(xi−X)(xi−X)T
= 1 N ( x 1 − X ‾ , . . . , x N − X ‾ ) ( ( x 1 − X ‾ ) T . . . ( x N − X ‾ ) T ) N ∗ 1 =\frac{1}{N}(x_1-\overline{X},...,x_N-\overline{X})\begin{pmatrix} (x_1-\overline{X})^{T} \\ ...\\ (x_N-\overline{X})^{T}\\ \end{pmatrix}_{N*1} =N1(x1−X,...,xN−X)⎝⎛(x1−X)T...(xN−X)T⎠⎞N∗1
= 1 N X T ( I N − 1 N 1 N 1 N T ) N X T ( I N − 1 N 1 N 1 N T ) T X =\frac{1}{N}X^T(I_N-\frac{1}{N}1_N1_N^T){N}X^T(I_N-\frac{1}{N}1_N1_N^T)^TX =N1XT(IN−N11N1NT)NXT(IN−N11N1NT)TX
将括号里的表达式看成H,则有:
= 1 N X T H H T X =\frac{1}{N}X^THH^TX =N1XTHHTX
可以尝试计算会发现:
H T = H H^T=H HT=H
H n = H H^n=H Hn=H
则有:
S p ∗ p = 1 N X T H X S_{p*p}=\frac{1}{N}X^THX Sp∗p=N1XTHX
1.2 最大投影方差&最小重构距离
classical PCA可以看成:
一个中心:原始特征空间的重构(使得特征从相关 → \to →无关);
两个基本点:1. 最大投影方差 2. 最小重构距离
基本思想是将原始特征在一个方向上进行投影,思想和LDA线性判别分析有点像但是有区别,若有相关问题需要温故LDA就再写一篇LDA的笔记。使得在样本特征在投影方向上得到的新样本方差最大,且使得新样本数据能够区分,重构回去的可能性尽可能大。如图所示:
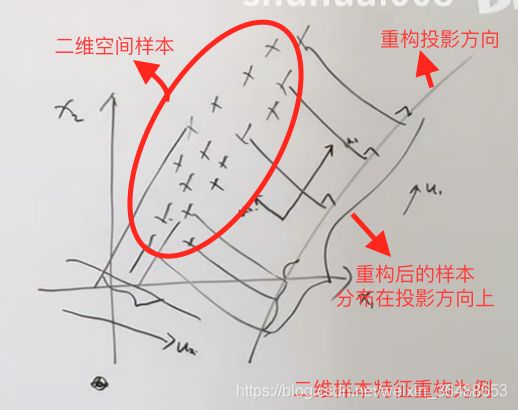
中心化: x i − X ‾ x_i-\overline{X} xi−X
投影方向为: u 1 ⃗ \vec{u_1} u1
根据如图所示的投影定理,则有:
J = ( ∑ i = 1 N ( x i − X ‾ ) T u 1 ⃗ ) 2 J=(\sum_{i=1}^N(x_i-\overline{X})^T\vec{u_1})^2 J=(∑i=1N(xi−X)Tu1)2
= J = ∑ i = 1 N u 1 ⃗ T ( x i − X ‾ ) ( x i − X ‾ ) T u 1 ⃗ =J=\sum_{i=1}^N\vec{u_1}^T(x_i-\overline{X})(x_i-\overline{X})^T\vec{u_1} =J=∑i=1Nu1T(xi−X)(xi−X)Tu1
= u 1 ⃗ T ( ∑ i = 1 N 1 N ( x i − X ‾ ) ( x i − X ‾ ) T ) u 1 ⃗ =\vec{u_1}^T(\sum_{i=1}^N\frac{1}{N}(x_i-\overline{X})(x_i-\overline{X})^T)\vec{u_1} =u1T(∑i=1NN1(xi−X)(xi−X)T)u1
= u 1 ⃗ T S u 1 ⃗ =\vec{u_1}^TS\vec{u_1} =u1TSu1
则PCA的求解可以看作带约束的优化问题:
{ u 1 ′ = a r g m a x u 1 T S u 1 s . t . u 1 T u 1 = 1 \begin{cases} u_1'=argmaxu_1^TSu_1\\ s.t.u_1^Tu_1=1\\ \end{cases} {u1′=argmaxu1TSu1s.t.u1Tu1=1
根据拉格朗日乘子法:
f ( u , λ ) = u 1 T S u 1 + λ ( u 1 T u 1 − 1 ) f(u,\lambda)=u_1^TSu_1+\lambda(u_1^Tu_1-1) f(u,λ)=u1TSu1+λ(u1Tu1−1)
求偏导:
∂ f ∂ u 1 = 2 S u 1 − λ 2 u 1 = 0 \frac{\partial f}{\partial u_1}=2Su_1-\lambda2u_1=0 ∂u1∂f=2Su1−λ2u1=0
则有:
S u 1 = λ u 1 Su_1=\lambda u_1 Su1=λu1
可以看出PCA其实是对特征空间的重构。每选定一个方向,就存在一个对应的特征值。新的重构坐标体系为 u 1 , u 2 , . . . , u p u_1,u_2,...,u_p u1,u2,...,up,则每个中心化后的的重构样本可以表示为:
x i = ∑ k = 1 p ( x i T u k ) u k x_i=\sum_{k=1}^p(x_i^Tu_k)u_k xi=k=1∑p(xiTuk)uk
而降维就是针对重构的特征空间样本根据需要保留空间维度,从所有的 p p p个空间中选择 q q q个维度作为保留,则降维后的样本表示为:
x i ′ = ∑ k = 1 q ( x i T u k ) u k x_i'=\sum_{k=1}^q(x_i^Tu_k)u_k xi′=k=1∑q(xiTuk)uk
则最小重构代价可以表示为:
J = 1 N ∑ i = 1 N ∣ ∣ x i − x i ′ ∣ ∣ 2 = 1 N ∑ k = q + 1 p ∣ ∣ x i − x i ′ ∣ ∣ 2 J=\frac{1}{N}\sum_{i=1}^{N}||x_i-x_i'||^2=\frac{1}{N}\sum_{k=q+1}^{p}||x_i-x_i'||^2 J=N1i=1∑N∣∣xi−xi′∣∣2=N1k=q+1∑p∣∣xi−xi′∣∣2
= 1 N ∑ i = 1 N ∑ k = q + 1 p ( x i T u k ) 2 =\frac{1}{N}\sum_{i=1}^{N}\sum_{k=q+1}^{p}(x_i^Tu_k)^2 =N1i=1∑Nk=q+1∑p(xiTuk)2
考虑中心化:
= 1 N ∑ i = 1 N ∑ k = q + 1 p ( ( x i − X ‾ ) T u k ) 2 =\frac{1}{N}\sum_{i=1}^{N}\sum_{k=q+1}^{p}((x_i-\overline{X})^Tu_k)^2 =N1i=1∑Nk=q+1∑p((xi−X)Tuk)2
= ∑ k = q + 1 p ∑ i = 1 N 1 N ( ( x i − X ‾ ) T u k ) 2 = ∑ k = q + 1 p u k T S u k =\sum_{k=q+1}^{p}\sum_{i=1}^{N}\frac{1}{N}((x_i-\overline{X})^Tu_k)^2=\sum_{k=q+1}^{p}u_k^TSu_k =k=q+1∑pi=1∑NN1((xi−X)Tuk)2=k=q+1∑pukTSuk
所以从两个角度看PCA会得到相同的结果。
2. PCA实操
sklearn中降维算法都包含在decomposition模块中。
2.1 重要参数n_components
代表降维后需要的维度,即降维后需要保留的特征数量。
2.1.1 高维数据的可视化特征分布
若要对数据进行可视化,则需要将特征降维到二维(三维也可)。
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
from sklearn.decomposition import PCA
iris = load_iris()
y = iris.target
X = iris.data
import pandas as pd
print(pd.DataFrame(X))
pca = PCA(n_components=2)
pca = pca.fit(X) # 拟合模型
X_dr = pca.transform(X) # 获取新矩阵
# 一步到位:X_dr = PCA(2).fit_transform(X)
# 取出分类为0的样本数据的第一个特征
X_dr[y == 0, 0]
# 取出分类为0的样本数据的第二个特征
X_dr[y == 0, 2]
# 画图,也可以循环画
plt.figure()
plt.scatter(X_dr[y==0, 0], X_dr[y==0, 1], c="red", label=iris.target_names[0])
plt.scatter(X_dr[y==1, 0], X_dr[y==1, 1], c="black", label=iris.target_names[1])
plt.scatter(X_dr[y==2, 0], X_dr[y==2, 1], c="orange", label=iris.target_names[2])
plt.legend()
plt.title("PCA of IRIS dataset")
plt.show()
# 查看新特征的可解释性方差大小
print(pca.explained_variance_)
# 查看新特征的可解释性方差贡献率(每个新特征携带原占始信息量比例)
print(pca.explained_variance_ratio_)
# 查看降维后信息占原始信息比例总和
print(pca.explained_variance_ratio_.sum())
import numpy as np
# 对列表进行累加
np.consum(pca.explained_variance_ratio_)
2.1.2 其他方式选取n_components
# 可以使用最大似然估计来取n_components的值
pca_mle = PCA(n_components="mle")
pca_mle = pca_mle.fit(X)
X_mle = pca_mle.transform(X)
# 按照信息量占比选取n_components
pca_f = PCA(n_componets=0.97, svd_solver="full")
pca_f = pca_f.fit(X)
X_f = pca_f.transform(X)
2.2 参数svd_solver&random_state
svd_solver有四种:auto(数据量小用full,大的话用randomized)、full(完整地分解出奇异值矩阵)、arpack(可以加速运算,适合特征矩阵较大且为稀疏矩阵)、randomized(适合特征矩阵巨大,计算量庞大)。
2.3 重要属性components_
from sklearn.datasets import fetch_lfw_people
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
import numpy as np
faces = fetch_lfw_people(min_faces_per_people=60)
X = faces.data
# 现实绘制数据集中的图像
# 4行5列 4*5=20张图
# figsize是画布大小
fig, axes = plt.subplot(4, 5, figsize=(8, 4), subplot_kw={"xticks":[], "yticks":[]})# 不显示坐标轴
for i, ax in enumerate(axes.flat):
ax.imshow(faces.images[i,:,:], cmap="gray")
pca = PCA(150).fit(X)
V = pca.components_
# 可视化components_核心信息
fig, axes = plt.subplot(3, 8, figsize=(8, 4), subplot_kw={"xticks":[], "yticks":[]})# 不显示坐标轴
for i, ax in enumerate(axes.flat):
ax.imshow(V[i,:].reshape(62,47), cmap="gray")
2.4 重要接口inverse_transform
降维是不可逆的,即使采用该接口也无法恢复。
X_inverse = pca.inverse_transform(X_dr)
fig, axes = plt.subplot(2, 10, figsize=(10, 2.5), subplot_kw={"xticks":[], "yticks":[]})# 不显示坐标轴
for i in range(10):
ax[0,i].imshow(faces.images[i,:,:], cmap="binary_r")
ax[1,i].imshow(X_inverse[i].reshape(62,47), cmap="binary_r")
2.5 用PCA做噪音过滤
from sklearn.datasets import load_digits
digits = load_digits()
# 查看标签
print(set(digits.target.tolist()))
# 绘图
def plot(data):
fig, axes = plt.subplot(4, 10, figsize=(10, 4), subplot_kw={"xticks":[], "yticks":[]})# 不显示坐标轴
for i, ax in enumerate(axes.flat):
ax.imshow(data[i].reshape(8,8), cmap="binary")
# 为数据加上噪音
np.random.RandomState(42)
noisy = np.random.normal(digits.data, 2)
plot(noisy)
pca = PCA(0.5, svd_solver="full").fit(noisy)
X_dr = pca.transform(noisy)
witout_noise = pca.inverse_transform(X_dr)
plot(without_noise)
2.6 实战:手写数据集降维
已经导过的包就不导了。
from sklearn.esemble import RandomForestClassifier as RFC
from sklearn.model_selection import cross_val_score
data = pd.read_csv("数据集CSV路径")
X = data.iloc[:, 1:]
y = data.iloc[:,0]
# 画累计方差贡献率曲线,找最佳降维后维度的范围
pca_line = PCA().fit(X)
plt.figure(figsize=[20,5])
plt.plot(np.consum(pca.line.explained_variance_ratio_))
plt.xlabel("number of components after dimension reduction")
plt.ylabel("cumulative explained variance")
plt.show()
# 降维后的学习曲线,继续缩小最佳维度的范围
score = []
# 缩小范围(10,25)再次绘制学习曲线
for i in range(1, 101, 10):
X_dr = PCA(i).fit_transform(X)
once = cross_val_score(RFC(n_estimator=10, random_state=0), X_dr, y, cv=5).mean()
score.append(once)
plt.figure(figsize=[20,5])
plt.plot(range(1, 101, 10), score)
plt.show()
# 找到最佳的参数后确定将维维度
X_dr = PCA(23).fit_transform(X)
cross_val_score(RFC(n_estimator=100, random_state=0), X_dr, y, cv=5).mean()
# 使用KNN来训练
from sklearn.neighbors import KNeighborsClassifier as KNN
cross_val_score(KNN(), X_dr, y, cv=5).mean()
# KNN的k值学习曲线
score = []
# 确定k值为3
for i in range(10):
X_dr = PCA(23).fit_transform(X)
once = cross_val_score(KNN(i+1), X_dr, y, cv=5).mean()
score.append(once)
plt.figure(figsize=[20,5])
plt.plot(range(10), score)
plt.show()

