Image Translation for Medical Image Generation Ischemic Stroke Lesion Segmentation——论文翻译
摘要
基于深度学习的疾病检测和分割算法有望改善许多临床过程。然而,此类算法需要大量带注释的训练数据,而由于数据隐私、法律障碍和非统一的数据获取协议,这些数据通常在医疗环境中不可用。带有注释病理的合成数据库可以提供所需数量的训练数据。我们以缺血性中风为例证明,基于深度学习的增强技术在病灶分割方面的改进是可行的。为此,我们训练不同的图像到图像的翻译模型,从语义分割图合成有和没有脑卒中病灶的脑体素磁共振图像。此外,我们训练生成对抗网络,以生成合成病变mask。随后,我们结合这两个组件来构建一个大的合成卒中图像数据库。使用U-Net评估各种模型的性能,该U-Net在临床测试集上用于训练分割中风病灶。我们报告了性能最好的模型的Dice得分为72.8%[70.8±1.0%],优于单独在临床图像上训练的模型67.3%[63.2±1.9%],接近人们已知的Dice得分76.9%。此外,我们还发现,对于只有10或50个临床病例的小型数据库,与不使用人工数据的情况相比,人工数据增强的效果显著。据我们所知,这是第一次比较分析基于图像到图像翻译的合成数据增强,并首次应用于缺血性中风。
关键词:生成对抗模型,图像到图像的翻译,脑卒中病灶分割,图像合成
1 介绍
缺血性中风(IS)是全球第二大死亡原因1,而且,即使不是致命的,也经常会导致不可逆的脑组织病灶和残疾。IS是由血管阻塞引起的,导致大脑某些区域的血液供应受到限制或抑制。因此,治疗的成功与症状的出现和成功的血管重建治疗之间的时间密切相关23。因此,简化和优化诊断过程至关重要。虽然许多临床中心使用计算机断层扫描(CT)作为中风的第一成像方式,因为它更广泛的可用性、速度和相对没有禁忌症,但识别和定量IS病变的金标准是扩散加权磁共振成像(DWI),与正常组织相比,梗死区诱发高强度,对应于相对扩散受限区。这些区域的手工分割相对简单,但对于训练有素的专家来说,这是一项乏味的任务,而使用标准方法进行自动分割则是一项困难的任务,例如,由于病变的几何形状复杂,磁共振信号强度的非微小变异性,不同的位置,以及大量的图像伪影。机器学习,特别是深度学习(DL),具有鲁棒性地自动化这些任务的潜力456。然而,DL模型的训练需要大量的标记数据,而这些数据通常由于隐私问题、不兼容的数据格式或疾病罕见而不可用。此外,手工分割医学图像是一项只有有经验的放射医师才能完成的任务,昂贵,容易出错,而且耗时。因此,大型医学图像注释数据库是稀缺的:例如,缺血性中风病灶分割挑战(ISLES)数据集包含64 IS卷7。相比之下,ImageNet数据库8是非医疗领域对象识别的一个流行数据集,包含1400万张图像。
使用深度生成模型9的合成图像生成可以通过增加可用的数据集或完全替换它们,绕过前面提到的与数据安全和可用性相关的担忧,从而将DL成功连接到医疗应用。然而,这些模型也需要大量的训练数据集,以获得良好的结果。此外,除了逼真的图像外,模型还必须提供病变标记,这在目前深度生成模型中是不能直接实现的。
我们尝试用图像到图像转换模型(ITMs)来合成脑卒中病灶的DWI,即从一个训练域转换到另一个训练域的DL模型:包含脑结构解剖标记和脑卒中病灶标记的语义分割图被翻译为脑DWIs。此外,新的病变标记由三维生成对抗网络生成。我们设想,解剖标签将引导网络产生与真实DWIs相当的组织对比清晰的图像。同时,训练集的病变标记将提供足够的信息来学习病理特异性的对比修饰。ITM应该学会在病变标记内部产生高强度,同时忽略任何(统计上未被充分代表的)错误分类,例如病变标记外部的高强度或被人为误分类为病变的正常组织。这有一个可喜的副作用:网络将产生与高置信度标记的输入区域相匹配的DWI病变,从而提供准确的病变标记。使用这种方法获得的合成图像可以用来增强临床数据库,或者,就像这里,直接用于监督学习,从正常脑组织中分割中风病灶。
相关工作 建议的方法在本质上类似,但比参考文献10更通用。其中,通过线性增加轮廓内的体素强度,将真实的IS病变轮廓融合到健康脑DWI中,从而模拟病变,最后生成合成的IS体素。这确保了解剖学边界条件正确,并生成了现实的强度模式。我们建议的一部分是将这个任务外包给生成DL模型,该模型概括了神经网络本质上是非线性的。通过他们的方法,作者实现了Dice相似系数(DSC)从65%的增加,当包含2,000张合成图像时,仅使用临床数据的比例达到70%,而使用40,000张合成DWI的比例甚至高达72%。这种方法的优势在于,它可以生成连贯的3D数据;然而,它的组合是有限的,因为只有有限数量真正的病变标记可用。此外,参考文献1112成功使用生成对抗网络(generative adversarial networks, GANs)生成合成磁共振图像(synthetic magnetic resonance images, mri),提高了脑肿瘤分割精度。13采用了一种不同的方:作者使用ITM从CT图像中获得DWI,这使他们能够提高CT上使用合成DWI增强的缺血性中风核心组织的分割质量。同样,Ref.14使用ct - mr图像翻译来改善肺肿瘤的分割。最近在文献15中提出了使用风格转换的进一步方法,其中使用图像到图像转换将健康的磁共振图像转换为病变的磁共振图像以进行脑肿瘤分割,在文献16中将磁共振图像转换为CT图像以进行一般治疗计划。最后,参考文献17介绍了一种用于各种医疗任务的ITM方法,包括图像去噪、运动校正和域自适应。
贡献 我们提供了一种从语义分割图中合成IS病灶标记和DWI的新方法。前一个任务由ITMs实现,病变标记由3D GAN生成。这使我们能够建立空间一致的脑体积,包括病灶的标签。此外,这使我们的管道具有可扩展性,因为GAN可以用来生产无限数量的合成的、但不同的病变标签。因此,我们证明了基于DL的数据增强易于利用有限的医疗数据集中包含的信息,并优于传统的数据增强技术,18提出了这一想法,并进一步发展了19在非医疗背景下的细分。通过比较人工标记数据训练的U-Net20和人工合成数据训练的U-Net20的DSC,我们量化了这一说法,每一个都在一组临床IS数据上进行评估。我们强调了我们的流程的几个应用领域,并强调了方法的实用性,因为训练有素的IT管理人员可以自由分发,而不存在任何数据隐私问题。目前已有许多研究探讨了下游临床任务的潜在改进,如解剖分割和肿瘤分割;然而,许多只讨论一个特定的模型设置。据我们所知,这是第一次使用ITMs对IS病灶分割进行数据增强的对比研究。
本文的结构如下。在第二章中,我们描述了在图像合成流程中使用的数据和模型,并详细说明了如何定量评估结果。分析的结果在第III节中提出和讨论,然后在第IV节中得出结论。附录中包含补充信息。
2 数据和方法
我们提出了一种方法,通过两个连续生成模型来生成显示IS病灶的大脑注释DWIs,一个用于生成真实的中风病灶标签,另一个用于将大脑分割掩模转换为DWIs。健康大脑的解剖分割mask是现成的,不需要医学培训,因为有几种自动分割算法存在。另一方面,假设GAN合成中风病灶标记引入一般的变换,以及可用病灶掩模的潜在空间表示的现实插值。最后,使用条件生成模型来生成合成图像,与无条件、潜在空间模型相比,显著改善了生成器的收敛行为。因此,我们构建了一个最小工具箱,可以很容易地通过利用GAN的插值能力来合成更多的病灶掩模,来生成更多的带注释的DWIs。该工作流程如图1所示。下面我们将详细描述所涉及的数据和方法。

图1. 训练 GAN 以从 365 个手动分割的中风患者的 DWI 的数据库中生成病变标签。使用相同的 365 个病理体积和另外 2027 个健康大脑 MR 扫描,训练各种 ITM 合成来自解剖分割掩码的 DWI。然后可以使用经过训练的 ITM 将假病灶标签与健康大脑分割掩码(真实或虚假)以生成具有高质量分割标签的合成中风 DWI。
2.1 数据
巴塞尔大学医院获得了具有IS症状的804个DWI患者的数据库,机构审查委员会批准了该数据库。该数据库(今后的卒中DB)包含449例DWI阳性病例,即诊断为IS病变(患者平均年龄72±14岁;左侧ISs 200例,右侧ISs 193例,双侧ISs 56例;女194名,男255名)。DWI阳性病例随机分为训练(365例)和测试(74例)。此外,测试集中纳入了85个DWI阴性样本,共159个测试样本。2027例健康DWI扫描的单独数据库(平均年龄38±24岁;1088名女性,939名男性)用于扩大流程(正常DB)。卒中DB中的大多数图像是在1.5T扫描仪上获得的(67% @1.5T;23% @ 3t),而大多数正常DB扫描使用3t扫描仪(68% @1.5T;平均而言,卒中DB扫描的回波和恢复时间相似(TE= 90±16 ms, TR= 7400±1300 ms[正常DB] vs.TE= 100±2 ms, TR= 7000±1500 ms[卒中DB])。
图像重新配准到蒙特利尔神经学研究所的标准图谱21,使用FSL的“大脑提取工具”[^23]进行头骨剥离,并使用ANTs22重新采样到标准分辨率128×128×40的体素。为了在训练过程中获得更好的稳定性,顶部和底部的四个切片被裁剪。体素强度被裁剪为99.5%,背景则被裁剪为绝对体素强度为35。最后,将信号强度重新调整到范围[−1,1]。使用3D U-Net训练通过FreeSurfer23获得的参考图像得到大脑的解剖分割,如参考文献24所述。这将整个数据库的处理时间减少到几个小时。
2.2 图像翻译
虽然无条件的GAN已经被训练来产生关于脑DWIs25的最先进的结果,但它们不能自动为解剖结构或病理提供基本的真实标签。另一方面,ITM是生成模型,它根据输入的分割图生成样本。因此,Ground Truth标签可以通过构造来获得。此外,这种方法通过提供分割标签的不同实例之间的边界,有助于提高生成的图像的质量。在这一节中,我们介绍了本文研究的ITM,进一步的细节可以在附录中找到。
This cycle consistency lends its name to the
2.2.1 Pix2Pix
Pix2Pix是一个经过充分研究的ITM,它首先由26提出,并由27进一步开发。Pix2Pix被广泛接受为成对图像翻译的选择方法,即当两个域的图像成对出现时,就像我们的数据库一样。它先前已成功地应用于医学数据,如参考文献12132829。最初的Pix2Pix是基于U-Net架构的;然而,在模型的高分辨率导数中使用残差块2730。我们已经训练了两种架构,但发现在质量上差别不大,因此使用了基于U-Net的版本,收敛更快。该网络的损失函数是对抗性损失和L1范数重建损失的加权和:
L P i x 2 P i x = E y [ l o g D ( y ) ] + E x [ 1 − l o g D ( G ( x ) ) ] + λ ∣ ∣ y − G ( x ) ∣ ∣ L 1 L_{Pix2Pix}=E_y[logD(y)] + E_x[1-logD(G(x))] + \lambda||y-G(x)||_{L1} LPix2Pix=Ey[logD(y)]+Ex[1−logD(G(x))]+λ∣∣y−G(x)∣∣L1
在这个公式中, E x / y E_{x/y} Ex/y表示从两个域中任意一个域获得的期望值,D表示运行在目标域上的鉴别器网络,G表示生成器网络。我们考虑重构损失权重λ的不同值,因为我们发现λ= 100(Pix2Pix100)比λ= 10(Pix2Pix10)的推荐值在质量上更有吸引力。对于鉴别器架构,我们依赖于PatchGAN2631 32。与普通鉴别器不同,PatchGAN不输出单一的数字来表征图像的真伪,而是输出图像的小块,因此可以在较小的尺度上为生成器提供更精确的反馈。L1 loss26能够很好地捕捉大尺度或低频特征。
2.2.2 CycleGAN
利用cycleGAN33给出了一种非成对图像平移的方法。两个生成器-鉴别器对,每个图像域一个,允许模型通过重构损失进行训练,使用第一个生成器从域A转换到B,然后使用另一个生成器从B转换到A,计算结果和原始图像之间的像素(L1或L2 norm)差异。这种循环一致性为模型提供了名称,并解决了未配对数据的问题。然而,需要训练的参数数量是相当庞大的,在我们的设置中达到了4200万。为了处理大量的自由参数,该模型强制一致性损失,这将惩罚生成器与一致性映射的偏差。注意cycleGAN已经成功应用于医学图像生成中的成对数据3435。
2.2.3 SPADE
SPADE是本研究中所考虑的最新的ITM。虽然它还没有广泛应用于医学领域,但环境图像的结果是有希望的36。此外,与标准GAN基本相同的SPADE架构非常经济,与其他ITM相比减少了需要训练的参数数量。与GANs9类似,该模型绘制了一个随机潜在向量,随后在生成过程中引入差异元素。分割掩码被注入到SPADE规范化层中,这些规范化层取代了通常的批处理或实例化规范化。这将引导模型更有效地学习和更快速地收敛到一个可见的吸引人的结果36。
2.2.4 Training
我们已经在tensorflow37中实现了所有ITM作为2D CNN,并在一个单一的GPU (Nvidia Titan RTX 24GB)上对它们进行了100个迭代的临床正常和中风合并数据库的训练。所有模型都使用批大小为8,学习速率为2·10−4,否则按照上一节中提到的参考文献进行设置。所有模型都在同样基础上进行训练,使用可用的分割体积获得一个条件生成模型,该模型仅在模型架构上不同于其竞争者,但在训练过程中相同。最后,我们在训练集中的三维病灶掩膜上训练一个GAN作为病灶标签生成器,对其进行500次迭代的训练。在接下来的病灶注入中,所有ITM使用相同的病灶生成器。
2.3 病灶注入和DWI生成
为了综合IS数据来补充现有的临床数据,我们使用2027个健康大脑DWIs的解剖分割图。原则上,我们也可以引入合成分割Ground Truth,这也有其自身的复杂性(主要与3D生成建模有关,见第III节E)。由于这不是我们分析的主要方面,我们诉诸于健康患者的可用分割。这也保证了增强体有足够的解剖学差异。这些健康的分割图随后通过注入假病灶进行修改,假病灶由3D Wasserstein GAN38生成,该GAN38在脑卒中DB的病灶mask上受训,其softmax输出通过阈值为0.5转换为硬分割编码。注入是通过调整DWI分割图的标签来实现的;然而,要求最小的病灶体积为20体素。如果不满足此要求,则生成一个新的病灶掩膜,直到生成至少20个体素的病灶。这种方法确保生成的脑容量的解剖特征是真实的,我们不会产生大量小的、分散的病变。Wasserstein损失在训练中被证明是最稳定的。此外,假设GAN产生的病变将推广以前通过合成数据进行数据增强的尝试,例如参考文献10。潜在的假设是生成模型在潜在空间中产生有意义的内插,同时对可用的临床病变mask进行简单(几何)转换的推广。
生成的分割图随后被分解成二维切片,并送入ITM以生成假的DWI。通过这种方式,我们获得了每个ITM包含2027个假DWIs的数据库。图2显示了这种病灶注入过程的一个例子,它也被绘制在图形摘要中。左列显示健康的输入DWI体积,其对应的语义分割图显示在其旁边。第三列根据GAN的输出显示植入病灶标签后的分割图。随后,我们使用Pix2Pix、SPADE和cycleGAN三个itm,从这些3D分割地图的轴向切片生成了真实的DWI,如图最右边的一列所示。
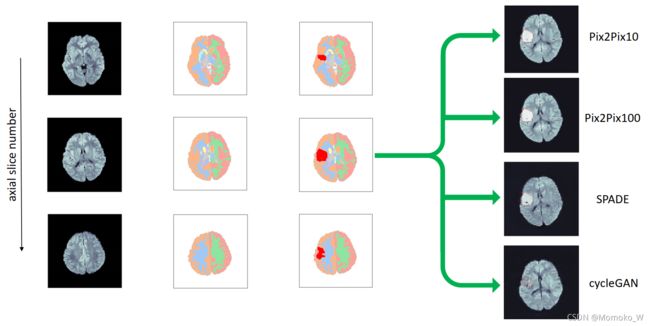
图2 植入假病变并产生 DWI。左:原始健康 DWI 的 3 片。中左:原始体素的分割。中右:带有由 GAN 生成的植入病灶标签的分割图。右图:使用不同的 ITM 从分割图生成的 IS 体素,如图所示。
2.4 评估病灶分割
为了评估各种ITM定量的性能,我们训练一个3D U-Net20分割网络在真实、真实和合成和仅合成数据库(每个上面讨论ITM),随后评估mask从训练数据中分离出来的测试集上的分割结果,包括74名中风患者和85名健康病人的DWI。图3是该过程的可视化表示。
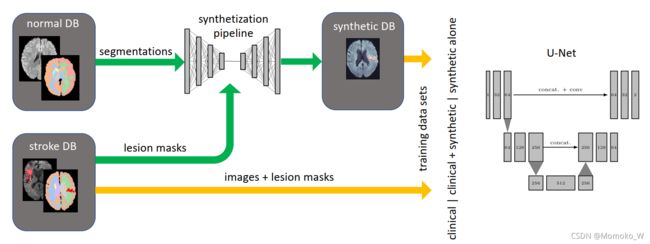
图3 评价方法。一旦所有 ITM 都经过训练,它们中的每一个都用于生成一组合成 DWI,这些 DWI用作 U-Net 分割网络的训练集的一部分。最终,U-Net 在临床测试集上进行评估病例。合成流程显示在图形摘要中。
U-Net在生物医学图像分割中是一个完善的标准,已经被用于各种任务,包括卒中1039,并且仍然产生与国家的最先进的40相匹配的结果。如果在100个迭代内验证损失没有增加,训练将停止。为此,15%的训练数据用于验证。为了定量地比较模型的性能,我们考虑在训练历史结束时选择100个迭代的范围,以评估测试集中的每个模型。
U-Net的架构在附录中有详细的介绍。该网络的输出是与输入相同维数的特征图;然而,它包含与分割标签一样多的图像通道(在我们的例子中是两个:背景和病灶),通过在通道轴上应用argmax可以获得硬分割。
通过交叉熵损失和DSC的组合对模型进行训练,并利用U-Net P的softmax输出和Ground Truth G进行计算
L U − n e t ( P , G ) = − D S C ( P , G ) + ∑ i G i l o g P i L_{U-net}(P,G)=-DSC(P,G) + \sum_iG_ilogP_i LU−net(P,G)=−DSC(P,G)+i∑GilogPi
D S C ( P , G ) = 2 ∣ P ⋂ G ∣ ∣ P ∣ + ∣ G ∣ = 2 ∑ i P i G i ∑ i P i + ∑ j G j DSC(P,G)=\frac{2|P\bigcap G|}{|P| + |G|}=\frac{2\sum_iP_iG_i}{\sum_iP_i+\sum_jG_j} DSC(P,G)=∣P∣+∣G∣2∣P⋂G∣=∑iPi+∑jGj2∑iPiGi
其中|·|表示集合的基数,(3)中的第二个等式适用于基于像素/体素的二进制分割映射,并且总和覆盖所有条目。为了更好的可比性,我们还报告了两个人为标注(每个人都有2年的经验)的分割之间的DSC。
此外,我们还通过一些额外的指标来评估分割的质量。首先,我们评估相对体积差,因为病灶体积是对治疗患者进行分诊的一个重要标准。此外,我们评估了Hausdorff距离(HD)和平均对称表面距离(ASSD),这两个量经常被提到,用来量化预测的病变形状与Ground Truth相比的质量。曲面距离 d A ( b ) d_A(b) dA(b)定义为点b∈B与曲面A的最小欧氏距离,即b与a∈A的任意点的带间的最小距离。由此,ASSD被构造为对称和,
A S S D ( A , B ) = 1 ∣ A ∣ + ∣ B ∣ ( ∑ a ∈ A d B ( a ) + ∑ b ∈ B d A ( b ) ) ASSD(A,B) = \frac{1}{|A| + |B|} \left(\sum_{a\in A}d_B(a) + \sum_{b \in B}d_A(b)\right) ASSD(A,B)=∣A∣+∣B∣1(a∈A∑dB(a)+b∈B∑dA(b))
而(对称)HD是最大的对称表面距离,
H D ( A , B ) = 1 2 ( m a x a ∈ A d B ( a ) + m a x b ∈ B d A ( b ) ) HD(A,B)=\frac{1}{2}\left( max_{a \in A}d_B(a) + max_{b \in B}d_A(b) \right) HD(A,B)=21(maxa∈AdB(a)+maxb∈BdA(b))
最后,对模型的召回率和进精度进行了评价。所有的计算都依赖于python包MedPy[^45]。
U-Net在500个epoch(单独的临床数据)或300个epoch(所有其他)的临床、合成和组合数据集上进行训练,使用批量大小为5和学习速率为10−4。为了确保最佳的基准,我们对临床数据41使用标准的仿射数据增强,包括翻转、旋转、透视和平移。在某些情况下,我们进一步研究是否可以通过只使用少数临床病例微调,从而在合成病例上训练模型来提高性能。
为了评估训练集大小的影响,我们进行了许多后续实验,在这些实验中,我们评估了上述指标,并计算了根据临床数据和合成数据的分数训练的模型在测试集上的均值和标准差。我们考虑了两种情况:一种情况下,我们使用正常DB的分割掩码来合成假数据,另一种情况下,我们使用428个DWI阴性中风DB案例来代替。最后,我们将这428个病例的分割四次注入不同的随机种子,生成一个与正常DB大小相当的合成数据库。此外,我们分析了不同的传统数据增强技术的影响,而不使用任何增强。
3 结果
3.1 合成数据生成和定性评价
图4显示了一些生成图像的例子。ITMs生成的合成DWI显示的解剖结构在位置、形状和大小上是一致的,包括病变。左边的前两张图像是使用Pix2Pix生成的。面板(a)是使用重建损失权重λ=10生成的,而面板(b)是使用λ=100创建的。结果质量好,主观真实,并如预期的那样,伴有高保真病变标签。只有对一些体积进行更仔细的检查,才能发现更大的重建重量产生的图像稍微更清晰,病灶内部和脑组织的细节也更详细。因此,我们在下一节中对这两个模型进行了定量测试。
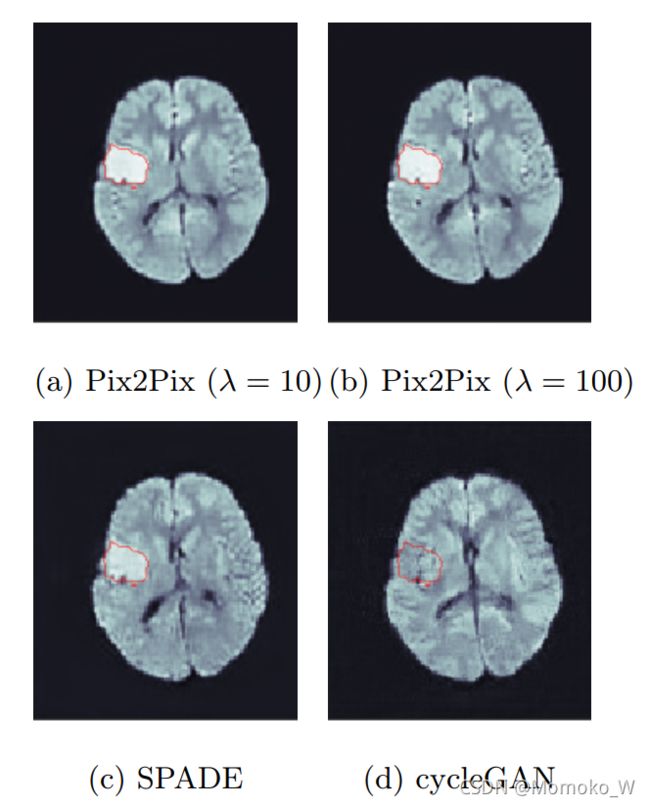
图4 从图2中的分割mask生成的卒中DWI
SPADE[图4©]生成的病灶强度明显低于Pix2Pix生成的图像,但病灶外的对比度也低得多:在合成脑的后部,几乎没有任何灰质/白质对比度。虽然乍一看这可能是一个缺点,但它实际上可能有利于训练U-Net来可靠地检测信号增加较低的病变。
接下来,注意cycleGAN不能识别和合成任何高强度的病变,而产生非常高的组织间对比[图4(d)]。我们已经尝试了在IS数据上微调模型,单独训练IS数据,并修改网络架构,然而,这一结果似乎是不可避免的。我们推测这与cycleGAN的循环一致性要求密切相关,即两个生成器尽可能接近地将图像映射回自身,以及病变体素与非病变体素之间存在较大的类不平衡。类似地,Ref.33确定了“失败的案例”,表明cycleGAN的泛化能力很差。由于这一失败,我们已经将cycleGAN排除在定量分析之外,并将使用cycleGAN进行数据增强的专门研究留给未来的工作。我们强调,这并不排除任何cycleGAN应用于这项任务;但是,它需要一种不同于这里选择的方法,因此没有给出ITM的可比性。
作为本节的总结,我们在图5中给出了经过预处理的合成数据集和真实数据集的强度直方图,并将其归一化到范围[0,1]。注意正常和临床(即中风)脑梗死在0.5以下区域的强度有显著差异。此外,合成图像的强度与正常体积强度密切相关。这并不令人惊讶,因为我们已经使用正常的分割mask来合成卒中图像。其中一个结论是,一些系统的差异,无论是解剖学的本质或根源于图像采集,是导致这种差异的原因。现在我们将转向图像生成质量的评估,并在第3.5节回到这个问题。
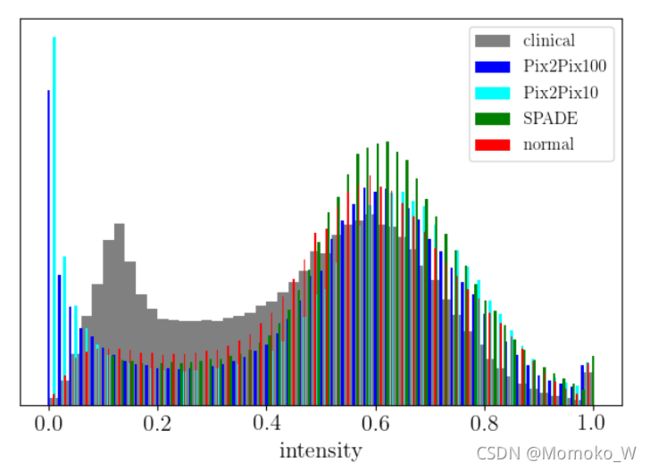
图5 生成和临床的卒中图像的强度分布
3.2 模型选择和测试集评估
结果如图6所示,汇总在表1中。在我们的设置中,对临床数据进行训练的U-Net达到平均测试集DSC为(63.2±1.9)%,最大DSC为67.3%。由Pix2Pix和临床数据结合合成数据训练的U-Net优于仅有临床数据的U-Net,我们发现平均DSCs为(70.3±1.1)%(Pix2Pix;λ= 100)和(70.8±1.0)%(Pix2Pix;λ= 10)。而SPADE表现稍差(62.9±1.2)。λ= 10时,Pix2Pix的最大DSC达到72.8%。其他模型的最大DSC达到66.5%(SPADE)和72.5%(Pix2Pix;λ= 100)。这是我们研究的一个关键结果,强调了合成数据并不是简单地复制训练数据中的可用信息,而是插值和有效地概括了训练数据。
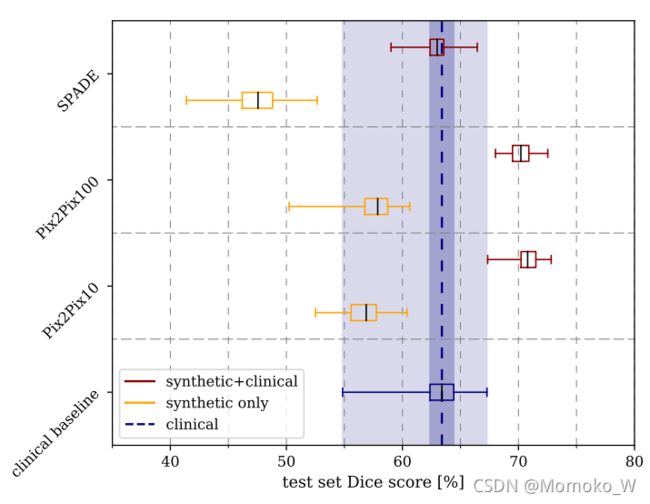
图6 在临床测试集上评估的各种 ITM 的盒线图。方框表示标准差和虚线最小值和最大值
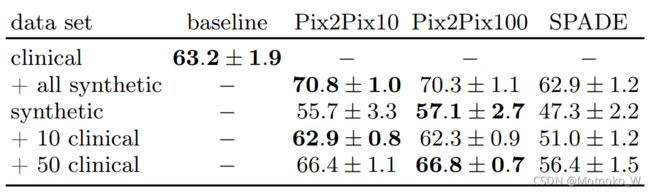
表1 用不同数量的合成数据训练的不同模型得到的 DSC 总结(以百分比表示)。最后两行对应于第3.3节中描述的微调步骤。
3.3 模型微调
这些结果表明,单纯的合成数据并不能取代临床数据集。这是否是因为生成图像的质量,还是用于图像合成和评估的数据库之间解剖特征的差异,将在下一节中进行调查。首先,我们要解决的问题是,仅通过合成数据训练的模型是否可以通过微调以产生更好的结果。
为此,我们对一个模型进行了微调,该模型之前仅使用10个或50个随机选择的临床训练样本进行了150个epoch的综合数据训练。在图7中,我们复制了之前的结果,其中模型单独在合成数据上训练(橙色)。随后,我们分别在10例(红色)和50例临床病例(暗红色)上显示训练结果。请注意Pix2Pix和SPADE这两个不同架构之间的区别。对于SPADE,将调优集大小从10增加到50只有少量改善:从10个样品的(51.0±1.2)%到50个样品的(56.4±1.5)%。在对50个样本进行调优后,SPADE的性能与临床基线模型的低频相匹配。Pix2Pix的情况非常不同,它的10个调优样本与临床基线的性能相匹配。对50个样本进行调优,可以获得足够的信息,使Pix2Pix优于临床模型,其平均DSC为(66.4±1.1)%(最大68.9%,λ= 10)和(66.8±0.7)%(最大68.3%,λ= 100)。在所有病例中,DSCs的方差都比临床基线低得多。如果我们只用10[50]个生成病例训练U-Net,结果会更糟:我们发现平均DSC为(2.9±1.6)%[(12.5±7.0)%]。

图7 微调模型的箱形图。仅根据合成数据训练的模型的性能可以得到改善达到匹配临床病例,甚至在某些情况下优于临床病例的效果。
因此,我们可以得出这样的结论:仅在合成数据上训练的模型与在临床数据集上训练的模型的表现不匹配。然而,这些模型只需要少量的临床训练案例就可以进行微调。这最终产生的结果与所有365例临床IS病例训练的临床基线模型相当;然而,方差要小得多。此外,在只有有限数量的临床病例可用的情况下,这种方法产生了显著的改善,正如我们在接下来的部分中强调的那样。
在图8中,我们展示了两个临床试验病例的分割示例,一个有大的病灶(顶部行),另一个有小的、不连通的病灶(底部行)。U-Nets已经在临床数据(最左边的面板)的训练,或组合数据(其他面板)上面进行了训练。在标记区域旁边,为Ground Truth(红色)和相应的DSC(整个体积)。

图 8 两个分割示例:一个大的病灶(顶部),它被所有模型很好地分割。和较小的、不连续的病变(底部)。后一种情况显示模型之间更大的DSC的差异。
3.4 更多实验
现在让我们关注在3.2节中获得最高DSC的模型,即Pix2Pix (λ= 10),并对其进行更详细的考虑。表2和图9是我们进行的四种不同类型实验的结果。第一行包含U-Net对10/25/50%的临床数据进行训练的结果,并在完整的测试集上进行评估。需要注意的是,在所有的情况下,误差都是相当大的,只有当更多的临床数据被纳入时,误差才会稍微减少,说明数据集是相当多样化的。随着训练集大小的增大,DSC迅速增大;然而,从50%到完整数据集只有一个小的改进。这种行为与相对体积损失形成对比,相对体积损失实际上对于完整的训练集来说是最大的。进一步观察发现,体积损失大的病例,其病灶体积较小(也见图8),因此,当研究不同大小的病灶时,体积损失似乎不是一个合适的度量指标。所有其他指标都证实使用完整的训练集可以获得最佳的性能。
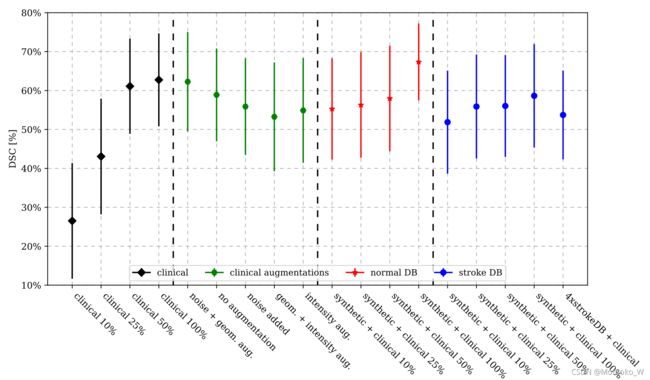
图 9 附加实验的结果,对应于表二的第一列。
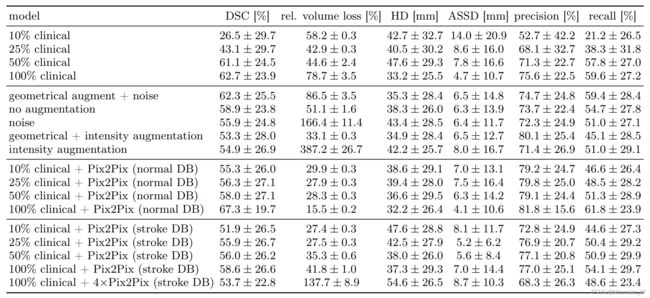
表2 使用不同的训练集大小和组成进行实验。结果显示了整个过程的平均值和标准差的测试集,第一部分将模型与部分临床数据进行比较,如图所示。第二部分比较了不同的数据
增加。在第三列和第四列中,显示了使用Pix2Pix合成数据的结果(λ=10)来自正常DB(第三列)和卒中DB(第四列)。在每个部分中,结果按DSC递增顺序排列,我们还包括了第3.2节中讨论的其他指标。
在第二类实验中,我们利用完整的训练集,并使用不同的数据增强技术,包括添加随机噪声和随机重映射图像强度,如参考文献42所建议的。然而,没有一个指标表明任何其他数据增强技术比单独的几何变换性能更好。
前一种情况下,对于小分数的训练数据集,可以得到显著的改进,例如当DWI阳性的临床病例少于40例(10%)时,可以得到大于50%的DSC。其他指标表明相同的行为。有趣的是,随着临床病例数量的增加,相对体积损失呈单调下降趋势,表明该模型对较小病变的预测更稳定。
有人可能会怀疑,使用来自卒中DB的分割mask(由于个体的年龄较高,平均而言脑室更大)将进一步提高性能;然而,事实并非如此。即使将分割mask回收四次(生成的数据集大小等于正常DB),并使用不同的损伤标签进行增强,我们也没有发现任何改进。因此,我们可以得出结论,在正常DB中增加可变性有利于U-Net的性能。此外,我们得出结论单独在合成数据上训练的模型的缺陷不是源于我们使用的分割mask的分布外性质,而是iITM本身或训练集大小的限制。
最后,我们对ISELS 2015 DWI图像7上的训练模型进行了评估。我们发现,所考虑的模型一致产生约50%的DSC,模型间差异不大。然而,这个测试集的差异相当大,标准差约为30%。对一些案例的进一步检查表明,ISLES数据的分割更为粗糙,因此DSCs实际上受到该测试集分割质量的限制,而不是模型的能力。定性地说,结果是高质量的,只有少数情况下,脑干的梗塞被我们的模型遗漏。图10是一个例子,其中模型提供了分割的非常详细的轮廓。另一方面,ground truth分割非常粗糙,甚至包括CSF以及不属于薄壁组织的结构。这有力地支持了我们的假设,即数据的质量抑制了模型的分数,而不是模型的性能本身。
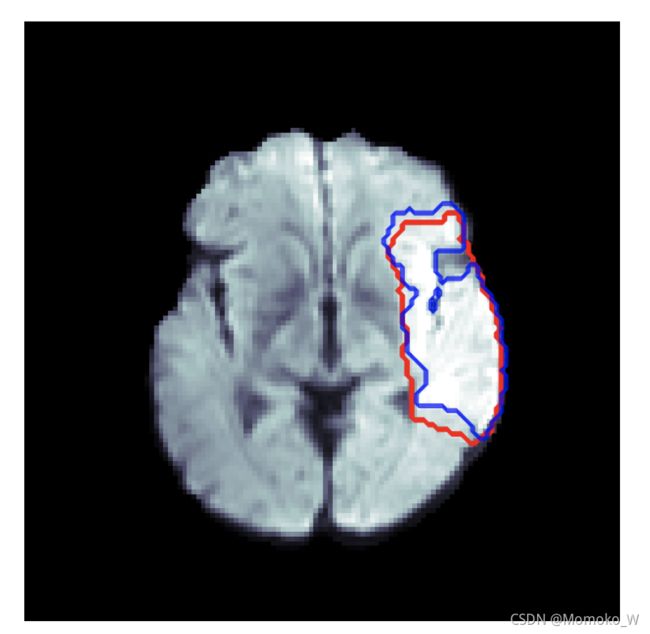
图10 对来自ISLES 2015 DWI训练数据的样本进行分割。红色:Ground Truth,蓝色:模型预测。注意到粗略的事实,脑脊液被标记为属于一个病变,而模型预测正确地遵循脑组织边界。
3.5 讨论
结果表明,基于DL的数据扩充可用于优化模型性能。根据可用的临床样本的数量,改进可能是巨大的,如表1和表2所示。
在生成数据增强的临床集上进行训练,Pix2Pix, λ=10的重建损失产生了最佳结果。我们发现,将重建损失权重增加到λ=100时,DSC最高。使用10个训练样本对该模型进行微调,其结果可以与临床数据集相媲美,而仅使用50个微调样本就可以产生更好的分割效果。这个结果与定性发现相匹配,即更大的重建损失产生比推荐的λ= 10更清晰的图像;然而,差异是相当微小的。SPADE框架是一个更经济的模型,其可调节参数更少,这有两个原因:第一,可训练权值越少,模型收敛时间越短,需要的训练数据也越少。其次,由于SPADE从潜在空间采样,它引入了随机性元素,而Pix2Pix则产生确定性输出。
如2.1节所述,每个数据库中的平均年龄差异显著[72±14岁(中风)与38±24岁(正常)]。因此,脑卒中DB表现出与老年患者有关的解剖特征,最明显的是脑室体积增大。例如,平均侧脑室容积为(9.1±6.6)ml(正常DB)与(20±12)ml(卒中DB)。我们已经验证了所有其他解剖特征在数据库之间是一致的。此外,大部分记录在1.5T扫描仪上,卒中DB图像比正常DB图像有更低的信噪比。为了调查这是否构成一个系统问题,我们从卒中DB的无病灶分割mask中生成了合成DWIs,但没有发现任何改善,见图9。因此,我们得出结论,如果综合数据引入额外的解剖变异,则是有益的。
最后,我们可以看到,所有模型在大的病变上都表现良好,如图8的上面板所示。相反,在底部面板中显示的病例,包括几个较小的病变,并不是所有模型都分割得同样好;尤其是只根据临床数据进行训练的U-Net会产生假阳性预测。这似乎是一个共同的特征,并且证明了20体素的低病灶阈值,这抑制了太多的错误预测。我们还训练了不限制病灶体积的模型;然而,只发现更差的表现和更多的假阳性标签。
我们的研究有几个局限性,需要在未来的工作中解决。首先,这是一项单中心研究,使用的数据仅来自一家培训机构。虽然我们确实尝试使用2015年的ISLES数据来评估我们的模型的泛化程度,但为了保证稳健性,还需要进行更多的定量分析。其次,DWI确实是病灶检测和量化的金标准,而CT是临床上更常见的成像方式,尽管病灶对比度低得多。在未来,一个重要的问题将是生成模型如何被用来产生不止一种对比。然而,在医学成像领域的适应性问题是正在进行的研究的主题,如参考文献4243。
将我们的结果与参考文献10进行比较,应该强调两个关键方面:在那里,作者使用从中风患者的真实DWI中提取的病灶,将它们植入健康患者的真实DWI中,从而创建一个类似的合成图像数据库来训练分割网络。虽然这创造了完全真实的3D图像(至少在病变外),但该方法在组合上是有限的,而我们的方法原则上可以从(连续的)潜在空间表示(SPADE和产生GAN的病变)生成连续的新的、不可见的图像,或分割mask的连续变形。虽然与我们的结果相似的是使用2000例合成病例的临床数据得到的,但作者没有单独讨论合成图像的使用。我们研究的一个关键结果是,一个U-Net只在ITMs生成的合成图像上训练,不能跟上模型在临床数据上训练的结果。然而,这些模型可以在少数临床病例中进行微调,其结果可以与纯临床环境中使用许多高质量的分割结果相媲美。
为了解决三维一致性的问题,我们也尝试对三维ITM进行培训。然而,训练3D生成模型是困难的,因为这些模型往往会迅速耗尽可用的内存容量,从而导致图像分辨率或训练批大小有限。此外,在三维数据上进行训练时,GAN通常不会收敛。因此,事实证明,提议的流程是最经济的设置,可靠地产生3D脑容量和可接受的3D一致性IS病变。这是由于我们使用了真正的解剖分割来填充ITM。
为了更好地看待这项研究的结果,我们报告了脑卒中病灶分割问题的一些近期方法的结果。前述的Ref.10报告了在与我们相同的最测集上获得的最大平均DSC为72.1%。这与我们的最大DSC 70.8%相比较,在一个标准差内是一致的。参考文献44中提出了另一种与我们的方法类似的方法,使用生成模型从CT输入图像中获得DWI。该方法产生了62.4%的DSC,而在没有生成模型的情况下,其DSC为57.2%。这突出了我们方法在跨模态分析中的可能应用,但也证实了我们的发现,即使用生成模型一般提高了分割的质量。请注意,最好的DSC与我们在临床DWI上获得的DSC相比,是在CT上获得的,CT上的IS相比DWI不那么明显。接下来,Ref.45选择利用Markov random fields对FLAIR图像进行脑卒中病灶的分割,得到58.2%的DSC。考虑到本文献中考虑的不同对比,这个数字不能直接与我们的结果进行比较。然而,本文提出的方法在系统上优于基于卷积神经网络的分割方法,从而为今后的研究提供了一个潜在的方向。类似地,Ref.46提出了一种所谓的放大缩小数据增强方法,该方法显著提高了基于神经网络在T1加权MR图像上分割中风病灶的质量。这表明形状变形可能是一种很有前途的方法来提高分割质量(也在合成图像上)。
4 总结&展望
我们研究了基于DL的图像到图像翻译模型在推广医学图像数据集数据增强方面的适用性。我们的结果强调了基于DL的算法比传统的数据增强技术产生了更好的性能。这与最近的多项研究一致,这些研究研究了不同的图像到图像翻译模型的性能。然而,迄今为止,只有少数文章研究了生成增强训练集对基于DL的分割和诊断系统的潜在改进1415。我们的工作表明,数据集大小的增加显著提高了分割网络的质量。此外,它还有许多有趣的结果,例如使用合成数据来训练疾病检测算法,例如在我们的案例中,用于IS病灶分割,避免了任何数据隐私障碍。因此,经过训练的ITM形式的数据可以提供给更广泛的受众,这有望加速机器学习在诊断医学应用方面的进展。此外,该技术可用于增加可用的数据集与合成数据。
根据我们的研究结果,我们可以想到几个方向。正如我们所看到的,仅仅对合成数据进行训练并不能提供有竞争力的结果。更仔细的数据准备可以提供与临床数据更一致的对比;然而,这是以将模型微调到单个数据集为代价的。或者,使用终身学习的思想4748,可以构建一个改进的网络架构,例如49的,它允许用户轻松地适应他们的数据,即使在完全不同的情况下获得的模型。在分割中整合多种对比是另一种有前途的方法。最后,建议的流程直接推广到其他可以通过DWI识别的病理,以及整个人体的其他成像方式。一个特别有趣的问题是,是否可以通过人工数据增加现有数据集来改善罕见疾病的诊断,以弥补临床训练数据的内在不平衡。
附录:模型结构
在本附录中,我们使用以下缩写:Ck为卷积,CTk为有k和filter的反卷积层,D是一个dropout层,(L) ReLU(leaky)修正线性单元激活层,M max-pooling层,U近邻上采样层,BN批标准化层,IN 实例归一化层。
U-net
3D U-Net20由一个下采样分支和一个上采样分支组成,由瓶颈层连接。在下采样分支中,我们包括4个块D16-D32-D64-D-128,上采样路径是U128-U64-U32-U16-Cn,其中n是分割标签的数量,在本例中n = 2(IS和non-IS),最后的卷积使用softmax激活。下采样块Dk是stride为1的卷积连续层Ck-ReLU-D-Ck-ReLU-D-M,上采样块Uk是带有stride为1的卷积和stride为2的反卷积 CTk-BN-ReLU-Ck-ReLU-D-Ck-ReLU-D。为了实现U-Net的特征跳跃连接,每个下采样块的输出与匹配图像分辨率的上采样分支的反卷积的输出相连接。在两个分支之间,所谓的瓶颈是C256-ReLU-D-C256-ReLU-D块。drop out为2%,所有卷积核为3×3×3。U-Net在128×128×32的图像分辨率下运行,共有 5.6 ∗ 1 0 6 5.6*10^6 5.6∗106个可训练参数。我们使用批处理大小为5的模型进行训练,这反映了内存的限制。
Pix2Pix
2D Pix2Pix生成器架构基于U-Net,包括下采样和上采样路径。生成器的细节在很大程度上与26提议的架构相同。但是,我们将批处理式的规范化替换为实例式的规范化50。下采样路径是D64-D128-D256-D512-D512-D512-D512-D512,其中下采样块Dk是Ck-IN-LReLU。每个卷积层包含4×4的kernel, stride为2,然后是实例归一化。在上采样路径中,我们使用上采样块U256-U128-U64,每个Uk由一个层序列U-Ck-LRelu-D-IN-LRelu和size为4×4的kernel,stride为1的卷积组成。下采样块的输出被连接到最后的ReLU层之前,leakiness为0.2。生成器模型共有 41.8 ∗ 1 0 6 41.8*10^6 41.8∗106个可训练参数,我们使用batch size为8进行训练。
SPADE
SPADE生成器从以原点为中心的128维随机正态分布中采样一个潜在矢量,各向同性方差为单位量级。然后将潜在向量传递到输出大小为4·4·128的dense层,并reshape为4*4*128的特征图。下面的五个residual块遵循36的原始设计,包括SPADE归一化层,它处理分割映射。每个块的设计是SPADE-LReLU-Ck-SPADE-LReLU-Ck,其中卷积的内核大小为3×3,单元stride与原始设置一样;然而,请注意,我们使用了较少的filter(128-64-32-16-8),Leaky ReLU层的斜率为0.2,每个SPADE块后面都有一个上采样层,直到达到所需的分辨率128×128×32。在末尾添加一个3×3的卷积、tanh激活层和一个filter来匹配数据维数。训练模型的批大小为8。
Discriminator
所有ITM都使用PatchGAN鉴频器263132,这是一个卷积网络,它的输出不是像普通鉴别器网络那样是单个数字。相反,PatchGAN输出一组数字,由于网络的卷积特性,这些数字只连接到输入图像的一个Patch。这些Patch的大小和数量取决于卷积层的数量。在最初的Pix2Pix设置中,考虑256×256图像,接收域是70×70。这里我们有三个卷积,C32-IN-LReLU-C64-IN-LReLU-C128-IN-LRelu,它减少了接收域从70×70到34×34 -这个选择反映了DWI切片考虑128×128的事实。最后一层后面是一个stride为1, size为1的filer,4×4的卷积,以映射到所需的输出形状。所有其他卷积的kernel大小为4×4, stride为2,以及所示的过滤器;Leaky ReLU的斜率为0.2。鉴别器模型共有888,898个参数。
Fake lesion generation
我们对几种结构和损失函数进行了实验,发现具有部分梯度损失51的Wasserstein GAN38产生了最好的结果。该模型可以稳定地训练到三维病灶mask上,为我们的合成流程提供了理想的构建块。该模型的输出激活是一个softmax函数,我们通过阈值化将其转化为病灶预测。GAN在IS数据库中提供的449个病灶mask上进行训练,批大小为8个,训练500个epoch。在每个卷积上采样步骤之后,我们使用实例归一化和Leaky ReLU(leakiness 0.2)激活。
参考文献
WHO, “The top ten causes of death,” https://www.who.int/en/news-room/fact-sheets/detail/the-top-10-causes-of-death, 2018, accessed: 08/10/2020. ↩︎
J. Tsai, M. Mlynash, S. Christensen, S. Kemp, S. Kim, N. Mishra, C. Federau, R. Nogueira, T. Jovin, T. Devlin, N. Akhtar, D. Yavagal, R. Bammer, M. Straka, G. Zaharchuk, M. Marks, G. Albers, and M. Lansberg, “Time from imaging to endovascular reperfusion predicts outcome in acute stroke,” Stroke, vol. 49, no. 4, pp. 952–957, Apr. 2018. ↩︎
J. Saver, M. Goyal, A. Lugt, B. Menon, C. Majoie, D. Dippel, B. Campbell, R. Nogueira, A. Demchuk, A. Tomasello, P. Cardona, T. Devlin, D. Frei, R. Rochemont, O. Berkhemer, T. Jovin, A. Siddiqui, W. Zwam, S. Davis, and M. Hill, “Time to treatment with endovascular thrombectomy and outcomes from ischemic stroke: A meta-analysis,” JAMA, vol. 316, pp. 1279–1288, 09 2016. ↩︎
N. Hainc, C. Federau, B. Stieltjes, M. Blatow, A. Bink, and C. Stippich, “The bright, artificial intelligenceaugmented future of neuroimaging reading,” Frontiers in Neurology, vol. 8, p. 489, 2017. ↩︎
J. Bernal, K. Kushibar, D. S. Asfaw, S. Valverde, A. Oliver, R. Martí, and X. Lladó, “Deep convolutional neural networks for brain image analysis on magnetic resonance imaging: a review,” Artificial Intelligence in Medicine, vol. 95, pp. 64 – 81, 2019. [Online]. Available: http://www.sciencedirect.com/science/article/pii/S0933365716305206 ↩︎
X. Yi, E. Walia, and P. Babyn, “Generative adversarial network in medical imaging: A review,” Medical Image Analysis, vol. 58, p. 101552, 2019, https://www.arxiv.org/abs/1809.07294. ↩︎
O. Maier et al., “ISLES 2015 - A public evaluation benchmark for ischemic stroke lesion segmentation from multispectral MRI Medical Image Analysis,” http://dx.doi.org/10.1016/j.media.2016.07.009, 2016, www. isles-challenge.org. ↩︎ ↩︎
Stanford Vision Lab, Stanford University, Princeton University, “ImageNet,” http://www.image-net.org, 2016, accessed: 08/10/2020. ↩︎
I. J. Goodfellow, J. Pouget-Abadie, M. Mirza, B. Xu, D. Warde-Farley, S. Ozair, A. Courville, and Y. Bengio, “Generative adversarial networks,” in Advances in neural information processing systems, 2014, pp. 2672–2680, https://www.arxiv.org/abs/1406.2661. ↩︎ ↩︎
C. Federau, S. Christensen, N. Scherrer, J. M. Ospel, V. Schulze-Zachau, N. Schmidt, H.-C. Breit, J. Maclaren, M. Lansberg, and S. Kozerke, “Improved segmentation and detection sensitivity of diffusionweighted stroke lesions with synthetically enhanced deep learning,” Radiology: Artificial Intelligence, vol. 2, no. 5, p. e190217, 2020. [Online]. Available: https://doi.org/10.1148/ryai.2020190217 ↩︎ ↩︎ ↩︎ ↩︎ ↩︎
H.-C. Shin, N. A. Tenenholtz, J. K. Rogers, C. G. Schwarz, M. L. Senjem, J. L. Gunter, K. P. Andriole, and M. Michalski, “Medical image synthesis for data augmentation and anonymization using generative adversarial networks,” in Simulation and Synthesis in Medical Imaging, A. Gooya, O. Goksel, I. Oguz, and N. Burgos, Eds. Cham: Springer International Publishing, 2018, pp. 1– 11. ↩︎
Q. Yang, N. Li, Z. Zhao, X. Fan, E. I.-C. Chang, and Y. Xu, “Mri cross-modality image-to-image translation,” Scientific Reports, vol. 10, no. 1, p. 3753, 2020. [Online]. Available: https://doi.org/10.1038/s41598-020-60520-6 ↩︎ ↩︎
J. Rubin and S. M. Abulnaga, “Ct-to-mr conditional generative adversarial networks for ischemic stroke lesion segmentation,” in 2019 IEEE International Conference on Healthcare Informatics (ICHI), 2019, pp. 1–7. ↩︎ ↩︎
J. Jiang, Y.-C. Hu, N. Tyagi, P. Zhang, A. Rimner, G. S. Mageras, J. Deasy, and H. Veeraraghavan, “Tumoraware, adversarial domain adaptation from ct to mri for lung cancer segmentation,” Med Image Comput Comput Assist Interv., vol. 11071, pp. 777–785, 2018. ↩︎ ↩︎
E. Vorontsov, P. Molchanov, W. Byeon, S. D. Mello, V. Jampani, M. Liu, S. Kadoury, and J. Kautz, “Boosting segmentation with weak supervision from image-to-image translation,” arXiv e-prints, vol. abs/1904.01636, 2019. [Online]. Available: http://arxiv.org/abs/1904.01636 ↩︎ ↩︎
P. Klages, I. Benslimane, S. Riyahi, J. Jiang, M. Hunt, J. O. Deasy, H. Veeraraghavan, and N. Tyagi, “Patch-based generative adversarial neural network models for head and neck mr-only planning,” IEEE Transactions on Medical Imaging, vol. XX, no. X, 2020. [Online]. Available: https://aapm.onlinelibrary.wiley.com/doi/abs/10.1002/mp.13927 ↩︎
K. Armanious, C. Jiang, M. Fischer, T. Küstner, T. Hepp, K. Nikolaou, S. Gatidis, and B. Yang, “Medgan: Medical image translation using gans,” Computerized Medical Imaging and Graphics, vol. 79, p. 101684, 2020. [Online]. Available: https://www.sciencedirect.com/science/article/pii/S0895611119300990 ↩︎
A. Antoniou, A. Storkey, and H. Edwards, “Data augmentation generative adversarial networks,” 2017. ↩︎
S. Pandey, P. R. Singh, and J. Tian, “An image augmentation approach using two-stage generative adversarial network for nuclei image segmentation,” Biomedical Signal Processing and Control, vol. 57, p. 101782, 2020. [Online]. Available: https://www.sciencedirect.com/science/article/pii/S1746809419303635 ↩︎
O. Ronneberger, P. Fischer, and T. Brox, “U-Net: Convolutional Networks for Biomedical Image Segmentation,” in Medical Image Computing and Computer-Assisted Intervention (MICCAI), 2015, pp. 234–241, https://www.arxiv.org/abs/1505.04597. ↩︎ ↩︎ ↩︎ ↩︎
McConnell Brain Imaging Centre, Montreal Neurological Institute, “MNI human atlas,” http://nist.mni.mcgill.ca/?page_id=714, 2009, accessed 08/10/2020.
[^ 23]: M. Jenkinson, C. F. Beckmann, T. E. Behrens, M. W. Woolrich, and S. M. Smith, “Fsl,” NeuroImage, vol. 62, no. 2, pp. 782–790, 2012, 20 YEARS OF fMRI. [Online]. Available: https://www.sciencedirect.com/science/article/pii/S1053811911010603 ↩︎University of Pennsylvania, Image Computing & Science Lab, “Advanced Normalization Tools (ANTs),” http://picsl.upenn.edu/software/ants, 2014, accessed 08/10/2020. ↩︎
B. Fischl, D. H. Salat, E. Busa, M. Albert, M. Dieterich, C. Haselgrove, A. van der Kouwe, R. Killiany, D. Kennedy, S. Klaveness, A. Montillo, N. Makris, B. Rosen, and A. M. Dale, “Whole brain segmentation: Automated labeling of neuroanatomical structures in the human brain,” Neuron, vol. 33, no. 3, pp. 341 – 355, 2002. [Online]. Available: http://www.sciencedirect.com/science/article/pii/S089662730200569X ↩︎
J. Zopes, M. Platscher, S. Paganucci, and C. Federau, “Multi-modal segmentation of 3d brain scans using neural networks,” 2020, https://www.arxiv.org/abs/2008.04594. ↩︎
A. U. Hirte, M. Platscher, T. Joyce, J. J. Heit, E. Tranvinh, and C. Federau, “Diffusion-weighted magnetic resonance brain images generation with generative adversarial networks and variational autoencoders: A comparison study,” arXiv e-prints, 2020, https://www.arxiv.org/abs/2006.13944. ↩︎
P. Isola, J.-Y. Zhu, T. Zhou, and A. A. Efros, “Image-toimage translation with conditional adversarial networks,” Conference on Computer Vision and Pattern Recognition (CVPR), 2017, https://www.arxiv.org/abs/1611.07004. ↩︎ ↩︎ ↩︎ ↩︎ ↩︎
T.-C. Wang, M.-Y. Liu, J.-Y. Zhu, A. Tao, J. Kautz, and B. Catanzaro, “High-resolution image synthesis and semantic manipulation with conditional gans,” in Proceedings of the IEEE conference on computer vision and pattern recognition, 2018, pp. 8798–8807, https://www.arxiv.org/abs/1711.11585. ↩︎ ↩︎
J. M. Wolterink, T. Leiner, M. A. Viergever, and I. Išgum, “Generative adversarial networks for noise reduction in low-dose ct,” IEEE Transactions on Medical Imaging, vol. 36, no. 12, pp. 2536–2545, 2017. ↩︎
H.-C. Shin, N. A. Tenenholtz, J. K. Rogers, C. G. Schwarz, M. L. Senjem, J. L. Gunter, K. P. Andriole, and M. Michalski, “Medical image synthesis for data augmentation and anonymization using generative adversarial networks,” in Simulation and Synthesis in Medical Imaging, A. Gooya, O. Goksel, I. Oguz, and N. Burgos, Eds. Cham: Springer International Publishing, 2018, pp. 1– 11. ↩︎
K. He, X. Zhang, S. Ren, and J. Sun, “Deep residual learning for image recognition,” in Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition (CVPR), June 2016, https://www.arxiv.org/abs/1512.03385. ↩︎
C. Li and M. Wand, “Precomputed real-time texture synthesis with markovian generative adversarial networks,” in Computer Vision – ECCV 2016, B. Leibe, J. Matas, N. Sebe, and M. Welling, Eds. Cham: Springer International Publishing, 2016, pp. 702–716, https://www.arxiv.org/abs/1604.04382. ↩︎ ↩︎
C. Ledig, L. Theis, F. Huszar, J. Caballero, A. P. Aitken, A. Tejani, J. Totz, Z. Wang, and W. Shi, “Photo-realistic single image super-resolution using a generative adversarial network,” 2017, pp. 4681–4690, https://www.arxiv.org/abs/1609.04802. ↩︎ ↩︎
J. Zhu, T. Park, P. Isola, and A. A. Efros, “Unpaired image-to-image translation using cycle-consistent adversarial networks,” in IEEE International Conference on Computer Vision (ICCV), 2017, pp. 2242–2251, https: //www.arxiv.org/abs/1703.10593. ↩︎ ↩︎
H. Yang, J. Sun, A. Carass, C. Zhao, J. Lee, Z. Xu, and J. Prince, “Unpaired brain mr-to-ct synthesis using a structure-constrained cyclegan,” in Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support, D. Stoyanov, Z. Taylor, G. Carneiro, T. Syeda-Mahmood, A. Martel, L. MaierHein, J. M. R. Tavares, A. Bradley, J. P. Papa, V. Belagiannis, J. C. Nascimento, Z. Lu, S. Conjeti, M. Moradi, H. Greenspan, and A. Madabhushi, Eds. Cham: Springer International Publishing, 2018, pp. 174–182. ↩︎
V. Sandfort, K. Yan, P. J. Pickhardt, and R. M. Summers, “Data augmentation using generative adversarial networks (CycleGAN) to improve generalizability in CT segmentation tasks,” Scientific reports, vol. 9, no. 1, p. 16884, November 2019, https://europepmc.org/articles/PMC6858365. ↩︎
T. Park, M.-Y. Liu, T.-C. Wang, and J.-Y. Zhu, “Semantic image synthesis with spatially-adaptive normalization,” Conference on Computer Vision and Pattern Recognition (CVPR), pp. 2332–2341, 2019, https://www.arxiv.org/abs/1903.07291. ↩︎ ↩︎ ↩︎
M. Abadi et al., “Tensorflow: A system for large-scale machine learning,” in 12th USENIX Symposium on Operating Systems Design and Implementation (OSDI 16), 2016, pp. 265–283. [Online]. Available: https://www.usenix.org/system/files/conference/osdi16/osdi16-abadi.pdf ↩︎
M. Arjovsky, S. Chintala, and L. Bottou, “Wasserstein GAN,” in Proceedings of the 34th International Conference on Machine Learning, Aug 2017, pp. 214–223, https://www.arxiv.org/abs/1701.07875. ↩︎ ↩︎ ↩︎
R. Zhang, L. Zhao, W. Lou, J. M. Abrigo, V. C. T. Mok, W. C. W. Chu, D. Wang, and L. Shi, “Automatic segmentation of acute ischemic stroke from dwi using 3-d fully convolutional densenets,” IEEE Transactions on Medical Imaging, vol. 37, no. 9, pp. 2149–2160, 2018. ↩︎
F. Isensee, P. F. Jaeger, S. Kohl, J. Petersen, and K. H. Maier-Hein, “nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation,” Nature methods, vol. 18, no. 2, p. 203–211, 2020.
[^ 45]: GitHub, “MedPy,” https://github.com/loli/medpy/. ↩︎A. Krizhevsky, I. Sutskever, and G. E. Hinton, “Imagenet classification with deep convolutional neural networks,” in Advances in Neural Information Processing Systems 25, F. Pereira, C. J. C. Weinberger,Burges, Eds. L. Bottou, and K. Q. Curran Associates, Inc., 2012, pp. 1097–1105. [Online]. Available: http://papers.nips.cc/paper/4824-imagenet-classification-with-deep-convolutional-neural-network.pdf ↩︎
L. S. Hesse, G. Kuling, M. Veta, and A. L. Martel, “Intensity augmentation for domain transfer of whole breast segmentation in mri,” 2019. ↩︎ ↩︎
H. Guan and M. Liu, “Domain adaptation for medical image analysis: A survey,” arXiv e-prints, p. arXiv:2102.09508, Feb. 2021. ↩︎
T. Song, “Generative model-based ischemic stroke lesion segmentation,” 2019. ↩︎
N. K. Subbanna, D. Rajashekar, B. Cheng, G. Thomalla, J. Fiehler, T. Arbel, and N. D. Forkert, “Stroke lesion segmentation in flair mri datasets using customized markov random fields,” Frontiers in Neurology, vol. 10, p. 541, 2019. [Online]. Available: https://www.frontiersin.org/article/10.3389/fneur.2019.00541 ↩︎
N. Tomita, S. Jiang, M. E. Maeder, and S. Hassanpour, “Automatic post-stroke lesion segmentation on mr images using 3d residual convolutional neural network,” NeuroImage: Clinical, vol. 27, p. 102276, 2020. [Online]. Available: https://www.sciencedirect.com/science/article/pii/S2213158220301133 ↩︎
S. Thrun, Lifelong Learning Algorithms. Boston, MA: Springer US, 1998, pp. 181–209. [Online]. Available: https://doi.org/10.1007/978-1-4615-5529-2_8 ↩︎
S. J. Pan and Q. Yang, “A survey on transfer learning,” IEEE Transactions on Knowledge and Data Engineering, vol. 22, no. 10, pp. 1345–1359, 2010. ↩︎
N. Karani, K. Chaitanya, C. Baumgartner, and E. Konukoglu, “A lifelong learning approach to brain mr segmentation across scanners and protocols,” in Medical Image Computing and Computer Assisted Intervention – MICCAI 2018, A. F. Frangi, J. A. Schnabel, C. Davatzikos, C. Alberola-López, and G. Fichtinger, Eds. Cham: Springer International Publishing, 2018, pp. 476– 484. ↩︎
D. Ulyanov, A. Vedaldi, and V. S. Lempitsky, “Instance normalization: The missing ingredi 2016,ent for fast stylization,” arXiv e-prints, http://arxiv.org/abs/1607.08022. ↩︎
I. Gulrajani, F. Ahmed, M. Arjovsky, V. Dumoulin, and A. C. Courville, “Improved training of wasserstein gans,” arXiv e-prints, 2017, http://arxiv.org/abs/1704.00028,. ↩︎