【机器学习sklearn】简单易懂核密度估计KernelDensity
前言
作业中遇到了需要使用KernelDensity的情况,但是网上的资料参差不齐,找了不短的时间却失望而归,最后还是靠着自己的理解才弄懂sklearn这个函数的使用,特此纪念。
【机器学习sklearn】简单易懂核密度估计KernelDensity
前言
官方文档
官方Sample解读
直方图
核密度
内核可视化
官方实例代码解析:
我的示例
所需文件获取:
题目:
参考答案:(jupyter notebook下环境)
0.导入包
1、数据预处理
2、得到最佳带宽作为真实值(我认为比较合理的方式去选取真实值)
3、开始使用KDE
4,计算估计密度与地面真实密度之间的MSE
官方文档
sklearn.neighbors.KernelDensity官方手册![]() https://scikit-learn.org/stable/modules/generated/sklearn.neighbors.KernelDensity.html#sklearn.neighbors.KernelDensity
https://scikit-learn.org/stable/modules/generated/sklearn.neighbors.KernelDensity.html#sklearn.neighbors.KernelDensity
官方Sample解读
这个例子使用KernelDensity类来演示一维核密度估计的原理。
直方图
下图显示了使用直方图可视化一维点密度的一个问题。直观上,直方图可以被认为是一种方案,其中一个单元“块”堆叠在规则网格上的每个点之上。然而,正如上面的两个面板所显示的那样,对这些块的网格选择可能会导致对密度分布的潜在形状产生截然不同的想法。
补充:可以看出直方图表达数据密度受带宽的取值影响非常之大,同一个数据不同的带宽完全是两种不同的图性,可见这种方式对于表达密度来讲并不理想。
核密度
如果我们将每个块放在它所代表的点的中心,我们就会得到面板所示的估计值。这是一个带有“大礼帽”核的核密度估计。这个想法可以推广到其他核形状:下图的右下面板显示了相同分布上的高斯核密度估计。
补充:以每个数据作为中心点设置带宽b,(x-b,x+b)区间范围内存在数据便统计在该柱形图上(这里和微积分的思想有一点类似,把一个不规则形状用无数个无线小的柱形描述他们的面积),这意味着带宽越小,那么数据划分的就越细致,但是也更加尖锐,容易欠拟合;带宽越大,那么数据划分的自然也就较粗糙,但是更加平滑,容易过拟合。
内核可视化
Scikit-learn使用球树或KD树结构,通过内核密度估计器实现高效的内核密度估计。可用的内核显示在本示例的下图中。
补充:不同的数据分布使用不同的内核,比如符合高斯分布的数据请使用gaussian内核。
官方实例代码解析:
# ----------------------------------------------------------------------
# Plot a 1D density example
# ---------------------------------------------------------------------------
'''
用随机种子生成100个数据,其中30个是符合高斯分布(0,1)的数据,70个是符合高斯分布(5,1)的数据,
(0,1)表示以x轴上的0为中心点,宽度为1的高斯分布。
(5,1)表示以x轴上5为中心店,宽度为1的高斯分布
'''
# ---------------------------------------------------------------------------
N = 100
np.random.seed(1)
X = np.concatenate(
(np.random.normal(0, 1, int(0.3 * N)), np.random.normal(5, 1, int(0.7 * N)))
)[:, np.newaxis]
# ---------------------------------------------------------------------------
# 创建一个[-5,10]范围内包含1000个数据的等差数列
X_plot = np.linspace(-5, 10, 1000)[:, np.newaxis]
# 使用简单的高斯模型norm得到两个高斯分布的概率密度作为真实值(我不觉得这是最佳的办法)
true_dens = 0.3 * norm(0, 1).pdf(X_plot[:, 0]) + 0.7 * norm(5, 1).pdf(X_plot[:, 0])
fig, ax = plt.subplots()
# 填充出用简单高斯模型得出的密度真实值
ax.fill(X_plot[:, 0], true_dens, fc="black", alpha=0.2, label="input distribution")
colors = ["navy", "cornflowerblue", "darkorange"]
# 使用不同的内核进行拟合,我也不推荐这样做,我们首先应该是观察数据的分布,然后选择模型,而不是
# 一个个尝试,应该做的是调整我们的带宽。
kernels = ["gaussian", "tophat", "epanechnikov"]
# 划线的粗细
lw = 2
for color, kernel in zip(colors, kernels):
# 用X数据进行训练模型
kde = KernelDensity(kernel=kernel, bandwidth=0.5).fit(X)
# 在X_plot数据上测试
log_dens = kde.score_samples(X_plot)
# 画图
ax.plot(
X_plot[:, 0],
np.exp(log_dens),
color=color,
lw=lw,
linestyle="-",
label="kernel = '{0}'".format(kernel),
)
ax.text(6, 0.38, "N={0} points".format(N))
ax.legend(loc="upper left")
# 用'+'代表真实的数据并且画出,用于观察数据分布集中情况
ax.plot(X[:, 0], -0.005 - 0.01 * np.random.random(X.shape[0]), "+k")
ax.set_xlim(-4, 9)
ax.set_ylim(-0.02, 0.4)
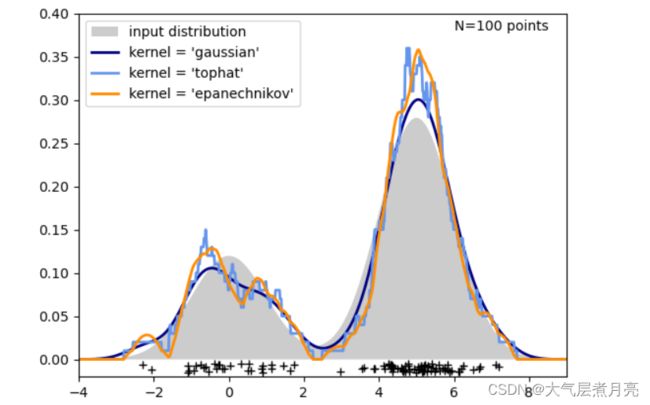
plt.show()上诉代码画出的示例图如下:该图比较了一维中100个样本分布的核密度估计。虽然本例使用1D分布,但核密度估计也可以轻松有效地扩展到更高的维度。补充:是有两个符合正态分布的数据叠加而成的。
在此,我更愿意提供一个更加合适的作业帮助大家理解KDE 。
我的示例:
所需文件获取:
百度网盘提取码:q4ef![]() https://pan.baidu.com/s/1eyyaxF51X4d9hZL_fQOVrA%C2%A0
https://pan.baidu.com/s/1eyyaxF51X4d9hZL_fQOVrA%C2%A0
题目:
a. Find and report the MSE between the estimated density and the ground truth density?
a.发现并报告估计密度与地面真实密度之间的MSE ?
b. What do you notice as you change the bandwidth parameter and why?
b.修改带宽参数时,您注意到什么?为什么?
参考答案:(jupyter notebook下环境)
0.导入包
# import package
import sklearn
from sklearn.neighbors import KernelDensity
from scipy.stats import norm
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
from collections import defaultdict1、数据预处理
# Step1、Data pretreatment
Q1_data = pd.read_csv('hw3/Question_1.csv')
X = np.array(Q1_data['X'].tolist())[:, np.newaxis]
N = len(X)
print('max_value_in_X:{}'.format(max(X)))
print('min_value_inX:{}'.format(min(X)))
X.shape2、得到最佳带宽作为真实值(我认为比较合理的方式去选取真实值)
# from sklearn.grid_search import GridSearchCV
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import LeaveOneOut
bandwidths = 10 ** np.linspace(-1, 1, 100)
grid = GridSearchCV(KernelDensity(kernel='gaussian'),{'bandwidth': bandwidths},cv=LeaveOneOut())
grid.fit(X)
# The best estimated bandwidth density is used as the truth value
best_KDEbandwidth = grid.best_params_['bandwidth']
kernel = "gaussian"
lw = 2
kde = KernelDensity(kernel=kernel, bandwidth=best_KDEbandwidth).fit(X)
truth_density = np.exp(kde.score_samples(X))
grid.best_params_3、开始使用KDE
# Step2、Kernel Density Estimation.
MSE_MAP = defaultdict(list)
fig, ax = plt.subplots()
ax.fill(X[:, 0], truth_density, fc="black", alpha=1, label="truth density")
bandwidths = [0.15, 0.5, 1]
colors = ["navy", "cornflowerblue", "darkorange"]
for bandwidth, color in zip(bandwidths, colors):
kde = KernelDensity(kernel=kernel, bandwidth=bandwidth).fit(X)
log_dens = kde.score_samples(X)
if bandwidth == best_KDEbandwidth:
bandwidth = 'ground truth'
ax.plot(
X[:, 0],
np.exp(log_dens),
color=color,
lw=lw,
linestyle="-",
label="bandwidth = '{0}'".format(bandwidth),
)
MSE_MAP[bandwidth] = log_dens
ax.text(6, 0.32, "N={0} points".format(N))
ax.legend(loc="upper right")
ax.plot(X[:, 0], -0.005 - 0.01 * np.random.random(X.shape[0]), "+k")
ax.set_xlim(-4, 9)
ax.set_ylim(-0.02, 0.50)
plt.show()(预测效果)
4,计算估计密度与地面真实密度之间的MSE
def cal_mse(a, b):
if len(a) == len(b):
n = len(a)
else:
return 'len(a) != len(b)'
res = 0
for i in range(n):
res += (a[i]-b[i])**2
return res/n
for bandwidth in MSE_MAP:
estimate_density = MSE_MAP[bandwidth]
MSE = cal_mse(estimate_density, truth_density)
print("When bandwidth is {:.2f} ----> MSE(estimate, truth): {:.3f}".format(bandwidth, MSE))When bandwidth is 0.15 ----> MSE(estimate, truth): 2.603
When bandwidth is 0.50 ----> MSE(estimate, truth): 2.803
When bandwidth is 1.00 ----> MSE(estimate, truth): 3.059可以看出MSE表示与3.中的图表示出的信息是一致的。
好啦,到此就结束啦!希望本文能帮到你。
如果觉得有用的话,欢迎大家三连~。祝你玩的开心。