python--kmeans聚类&DBSCAN聚类
以下内容笔记出自‘跟着迪哥学python数据分析与机器学习实战’,外加个人整理添加,仅供个人复习使用。
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
import warnings
warnings.filterwarnings('ignore')
import os
#os.chdir(r'')
1. 测试例子
构造数据:
from sklearn.datasets.samples_generator import make_moons
from sklearn.datasets.samples_generator import make_blobs
#构造非球形样本点
X1,y1=make_moons(n_samples=2000,noise=0.05,
random_state=1234)
#构造球形样本点
X2,y2= make_blobs(n_samples=1000,centers=[[3,3]],
cluster_std=0.5,random_state=1234)
#将y2中的0值替换为2(为避免与y1的值冲突,因为原始y1和y2中都有0这个值)
y2=np.where(y2==0,2,0)
#数据转换为数据框,用于绘图
plot_data=pd.DataFrame(np.row_stack([np.column_stack((X1,y1)),
np.column_stack((X2,y2))]),
columns=['x1','x2','y'])
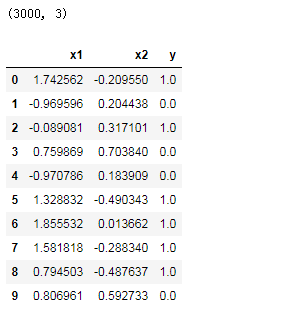
print(plot_data.shape)
plot_data.head(10)
plot_data.y.value_counts()
2.0 1000
0.0 1000
1.0 1000
Name: y, dtype: int64
作图查看数据分布:
sns.lmplot('x1','x2',data=plot_data,
hue='y',markers=['^','o','>'],
fit_reg=False,legend=False)
plt.show()
2. Kmeans聚类 & DBSCAN聚类
可先用拐点法、轮廓系数法等确定一下聚类个数(下篇)
- kmeans的初始质心是随机选定的,聚类结果受质心的影响
- dbscan需要设定半径和点个数,聚类结果受参数影响
2.1 建模
from sklearn.cluster import KMeans,DBSCAN
kmeans=KMeans(n_clusters=3,random_state=1234)
kmeans.fit(plot_data[['x1','x2']])
dbscan=DBSCAN(eps=0.3,min_samples=3)
dbscan.fit(plot_data[['x1','x2']])
#将聚类标签添加到数据集
plot_data['kmeans_label']=kmeans.labels_
plot_data['dbscan_label']=dbscan.labels_
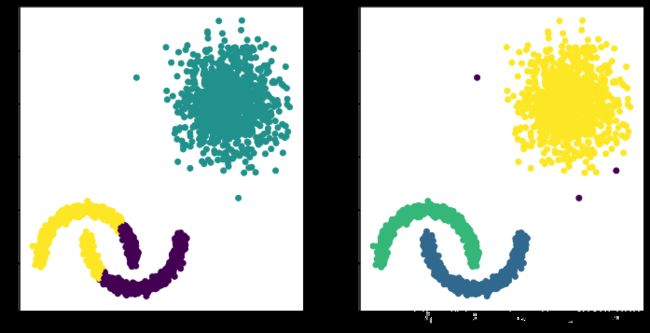
聚类结果绘图:
#绘图
fig,ax=plt.subplots(1,2,figsize=(12,6))
ax[0].scatter(plot_data.x1,plot_data.x2,
c=plot_data.kmeans_label)
ax[1].scatter(plot_data.x1,plot_data.x2,
c=plot_data.dbscan_label)
2.2 可以用轮廓系数作为评估指标
轮廓系数越接近1,表示聚类越合理。
from sklearn.metrics import silhouette_score
print('kmeans轮廓系数:')
print(silhouette_score(plot_data[['x1','x2']],kmeans.labels_))
print('dbscan轮廓系数:')
print(silhouette_score(plot_data[['x1','x2']],dbscan.labels_))
#额 好像不是很合理
kmeans轮廓系数:
0.5795065177613257
dbscan轮廓系数:
0.40195265087292487
3. 另一个建模例子(简)
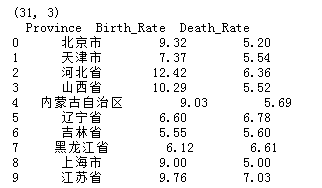
province=pd.read_excel(r'Province.xlsx')
print(province.shape)
print(province.head(10))
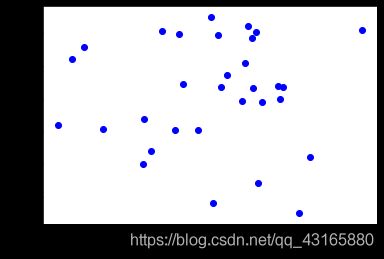
plt.rcParams['font.sans-serif']='SimHei'
plt.scatter('Birth_Rate','Death_Rate',c='b',data=province)
plt.xlabel('出生率')
plt.ylabel('死亡率')
3.1 数据预处理及建模
from sklearn import preprocessing
predictors=['Birth_Rate','Death_Rate']
#标准化
X=preprocessing.scale(province[predictors])
X=pd.DataFrame(X)
3.2 DBSCAN建模
由于dbscan建模的参数受半径和点个数的影响,这里可以通过for循环寻找合适参数。
np.arange(0.001,1,0.05)=[0.001,0.051,0.101 ,0.151 ,0.201 ,0.251 ,0.301 ,0.351 ,0.401, 0.451 0.501 ,0.551,0.601 ,0.651 ,0.701 ,0.751 ,0.801 ,0.851 ,0.901 ,0.951]
寻找合适参数:
# 构建空列表,保存不同参数组合下的结果
res=[]
#迭代不同的eps值
for eps in np.arange(0.001,1,0.05):
#迭代不同的min_samples值
for min_samples in range(2,10):
#拟合模型
#min_samples=3
dbscan=DBSCAN(eps=eps,min_samples=min_samples)
dbscan.fit(X)
#统计各参数组合下的聚类个数(-1表示异常点)
n_clusters=len([i for i in set(dbscan.labels_) if i !=-1])
#异常点个数
outliners=np.sum(np.where(dbscan.labels_==-1,1,0))
#统计每个簇的样本个数
stats=str(pd.Series([i for i in dbscan.labels_
if i !=-1]).value_counts().values)
#统计每次聚类的轮廓次数
if n_clusters>1:
com=silhouette_score(province[predictors],dbscan.labels_)
else:
com=0
#结果输出
res.append({
'eps':eps,'min_samples':min_samples,
'n_clusters':n_clusters,
'outliners':outliners,'stats':stats,
'轮廓系数':com})
#迭代结果存储到数据框
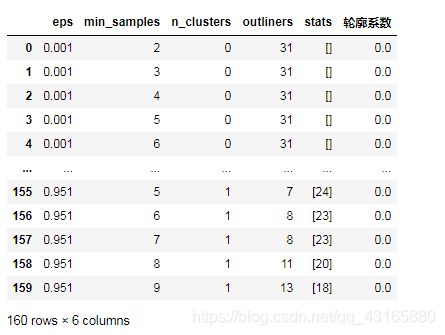
df=pd.DataFrame(res)
df
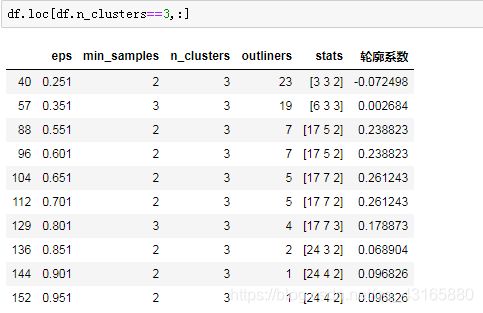
3.3 选择参数并聚类
#利用上述参数重新聚类
dbcscan=DBSCAN(eps=0.701,min_samples=2)
dbscan.fit(X)
province['dbscan_label']=dbscan.labels_
silhouette_score(province[predictors],dbscan.labels_)
0.28297765210486137
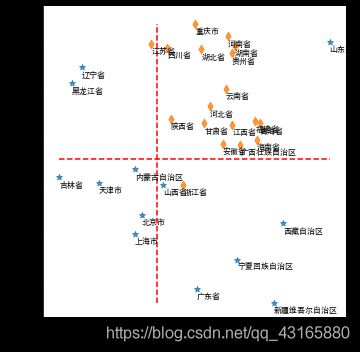
3.4 聚类效果作图
province.dbscan_label.value_counts()
0 18
-1 13
Name: dbscan_label, dtype: int64
#作图
plt.figure(figsize=(20,15))
sns.lmplot(x='Birth_Rate',y='Death_Rate',
hue='dbscan_label',
data=province,markers=['*','d'],
fit_reg=False,legend=False)
for x,y,text in zip(province.Birth_Rate,province.Death_Rate,
province.Province):
plt.text(x+0,y-0.1,text,size=8)
#添加参考线
plt.hlines(y=5.8,xmin=province.Birth_Rate.min(),
xmax=province.Birth_Rate.max(),
linestyles='--',colors='red')
plt.vlines(x=10,ymin=province.Death_Rate.min(),
ymax=province.Death_Rate.max(),
linestyles='--',colors='red')
plt.xlabel('出生率')
plt.ylabel('死亡率')
plt.show()