Kmeans&DBSCAN
文章目录
-
- Kmeans
-
- K-means定义
- K-Means算法流程
- K-Means优缺点
- K-means优化
-
- K-means初始化的优化方法K-Means++
- K-Means距离计算优化elkan K-Means
- 大样本优化Mini Batch K-Means
- K-Means与KNN
- KMeans参数
- K-means结果评估
- K-means练习sklearn
- DBSCAN
Kmeans
K-means定义
均值聚类算法(k-means clustering algorithm)是一种迭代求解的聚类分析算法,其步骤是,预将数据分为K组,则随机选取K个对象作为初始的聚类中心,然后计算每个对象与各个种子聚类中心之间的距离,把每个对象分配给距离它最近的聚类中心。聚类中心以及分配给它们的对象就代表一个聚类。每分配一个样本,聚类的聚类中心会根据聚类中现有的对象被重新计算。这个过程将不断重复直到满足某个终止条件。终止条件可以是没有(或最小数目)对象被重新分配给不同的聚类,没有(或最小数目)聚类中心再发生变化,误差平方和局部最小。
K-Means算法流程
输入是样本集 D = x 1 , x 2 , . . . , x m D={x_1,x_2,...,x_m} D=x1,x2,...,xm,聚类的簇树 k k k,最大迭代次数 N N N
输出是簇划分 C = C 1 , C 2 , . . . , C k C={C_1,C_2,...,C_k} C=C1,C2,...,Ck
- 从数据集 D D D中随机选择 k k k个样本作为初始的k个质心向量: μ 1 , μ 2 , . . . , μ k {\mu_1,\mu_2,...,\mu_k} μ1,μ2,...,μk
2)对于 n = 1 , 2 , . . . , N n=1,2,...,N n=1,2,...,N
\qquad a) 将簇划分 C C C初始化为 C t = ∅ , t = 1 , 2 , . . . , k C_t=\varnothing,\quad t=1,2,...,k Ct=∅,t=1,2,...,k
\qquad b) 对于 i = 1 , 2 , . . . , m i=1,2,...,m i=1,2,...,m,计算样本 x i x_i xi和各个质心向量 μ j ( j = 1 , 2 , . . . , k ) \mu_j(j=1,2,...,k) μj(j=1,2,...,k)的距离: d i j = ∣ ∣ x i − μ j ∣ ∣ 2 2 d_{ij}=||x_i−\mu_j||_2^2 dij=∣∣xi−μj∣∣22,将 x i x_i xi标记为最小的 d i j d_{ij} dij所对应的类别 C j C_j Cj。此时更新 C j = C j ⋃ { x i } C_j=C_j\bigcup\{x_i\} Cj=Cj⋃{xi}
\qquad c) 对于 j = 1 , 2 , . . . , k j=1,2,...,k j=1,2,...,k,对 C j C_j Cj中所有的样本点重新计算新的质心 μ j = 1 C j ∑ x ∈ C j x \mu_j=\dfrac{1}{C_j}\displaystyle\sum_{x\in C_j}x μj=Cj1x∈Cj∑x
\qquad d) 如果所有的 k k k个质心向量都没有发生变化,则转到步骤3)
3) 输出簇划分 C = C 1 , C 2 , . . . , C k C={C_1,C_2,...,C_k} C=C1,C2,...,Ck
K-Means优缺点
\quad 主要优点有:
-
原理比较简单,实现也是很容易,收敛速度快。
-
聚类效果较优。
-
算法的可解释度比较强。
-
主要需要调参的参数仅仅是簇数k。
\quad 主要缺点有:
-
K值的选取不好把握
-
对于不是凸的数据集比较难收敛
-
如果各隐含类别的数据不平衡,比如各隐含类别的数据量严重失衡,或者各隐含类别的方差不同,则聚类效果不佳。
-
采用迭代方法,得到的结果只是局部最优。
-
对噪音和异常点比较的敏感。
K-means优化
K-means初始化的优化方法K-Means++
k k k个初始化的质心的位置选择对最后的聚类结果和运行时间都有很大的影响,因此需要选择合适的 k k k个质心。如果仅仅是完全随机的选择,有可能导致算法收敛很慢。
K-Means++的对于初始化质心的优化策略如下:
\qquad a) 从输入的数据点集合中随机选择一个点作为第一个聚类中心 μ 1 \mu_1 μ1
\qquad b) 对于数据集中的每一个点 x i x_i xi,计算它与已选择的聚类中心中最近聚类中心的距离 D ( x i ) = a r g m i n ∣ ∣ x i − μ r ∣ ∣ 2 2 , r = 1 , 2 , . . . , k D(x_i)=argmin||x_i−\mu_r||_2^2,r=1,2,...,k D(xi)=argmin∣∣xi−μr∣∣22,r=1,2,...,k
\qquad c) 选择一个新的数据点作为新的聚类中心,选择的原则是: D ( x ) D(x) D(x)较大的点,被选取作为聚类中心的概率较大
\qquad d) 重复b和c直到选择出 k k k个聚类质心
\qquad e) 利用这 k k k个质心来作为初始化质心去运行标准的K-Means算法
K-Means距离计算优化elkan K-Means
在传统的K-Means算法中,每轮迭代时,要计算所有的样本点到所有的质心的距离,这样会比较的耗时。那么,对于距离的计算有没有能够简化的地方呢?elkan K-Means算法就是从这块入手加以改进。它的目标是减少不必要的距离的计算。那么哪些距离不需要计算呢?
elkan K-Means利用了两边之和大于等于第三边,以及两边之差小于第三边的三角形性质,来减少距离的计算。
-
第一种规律是对于一个样本点 x x x和两个质心 μ j 1 , μ j 2 \mu_{j1},\mu_{j2} μj1,μj2。如果我们预先计算出了这两个质心之间的距离 D ( j 1 , j 2 ) D(j1,j2) D(j1,j2),则如果计算发现 2 D ( x , j 1 ) ≤ D ( j 1 , j 2 ) 2D(x,j1)≤D(j1,j2) 2D(x,j1)≤D(j1,j2),我们立即就可以知道 D ( x , j 1 ) ≤ D ( x , j 2 ) D(x,j1)≤D(x,j2) D(x,j1)≤D(x,j2)。此时我们不需要再计算 D ( x , j 2 ) D(x,j2) D(x,j2),也就是说省了一步距离计算。
-
第二种规律是对于一个样本点 x x x和两个质心 μ j 1 , μ j 2 \mu_{j1},\mu_{j2} μj1,μj2。我们可以得到 D ( x , j 2 ) ≥ max { 0 , D ( x , j 1 ) − D ( j 1 , j 2 ) } D(x,j2)≥\max\{0,D(x,j1)−D(j1,j2)\} D(x,j2)≥max{0,D(x,j1)−D(j1,j2)}。这个从三角形的性质也很容易得到。
利用上边的两个规律,elkan K-Means比起传统的K-Means迭代速度有很大的提高。但如果样本特征稀疏,缺失值多的话,这个方法就不适用了,此时某些距离无法计算,则不能使用该算法。
大样本优化Mini Batch K-Means
在统的K-Means算法中,要计算所有的样本点到所有的质心的距离。如果样本量非常大,比如达到10万以上,特征有100以上,此时用传统的K-Means算法非常的耗时,就算加上elkan K-Means优化也依旧。在大数据时代,这样的场景越来越多。此时Mini Batch K-Means应运而生。
顾名思义,Mini Batch,也就是用样本集中的一部分的样本来做传统的K-Means,这样可以避免样本量太大时的计算难题,算法收敛速度大大加快。当然此时的代价就是我们的聚类的精确度也会有一些降低。一般来说这个降低的幅度在可以接受的范围之内。
在Mini Batch K-Means中,我们会选择一个合适的批样本大小batch size,我们仅仅用batch size个样本来做K-Means聚类。那么这batch size个样本怎么来的?一般是通过无放回的随机采样得到的。
为了增加算法的准确性,我们一般会多跑几次Mini Batch K-Means算法,用得到不同的随机采样集来得到聚类簇,选择其中最优的聚类簇。
K-Means与KNN
初学者很容易把K-Means和KNN搞混,两者其实差别还是很大的。
-
K-Means是无监督学习的聚类算法,没有样本输出
-
KNN是监督学习的分类算法,有对应的类别输出
-
KNN基本不需要训练,对测试集里面的点,只需要找到在训练集中最近的k个点,用这最近的k个点的类别来决定测试点的类别
-
K-Means则有明显的训练过程,找到k个类别的最佳质心,从而决定样本的簇类别
当然,两者也有一些相似点,两个算法都包含一个过程,即找出和某一个点最近的点。两者都利用了最近邻(nearest neighbors)的思想。
KMeans参数
-
n_clusters: k值,交叉验证选出最优的k值。
-
max_iter: 最大的迭代次数,一般如果是凸数据集的话可以不管这个值,如果数据集不是凸的,可能很难收敛,此时可以指定最大的迭代次数让算法可以及时退出循环。
-
n_init:用不同的初始化质心运行算法的次数。由于K-Means是结果受初始值影响的局部最优的迭代算法,因此需要多跑几次以选择一个较好的聚类效果,默认是10,一般不需要改。如果你的k值较大,则可以适当增大这个值。
-
init: 即初始值选择的方式,一般使用默认’k-means++’
'random':完全随机选择k个质心 'k-means++':'k-means++'方法选择初始化质心 自定义初始化k个质心 -
algorithm:有’auto’, ‘full’ or ‘elkan’三种选择。一般使用默认的’auto’
'full':传统的K-Means算法 'elkan:上面讲的elkan K-Means算法 'auto':根据数据值是否是稀疏的,来选择'full'和'elkan'。一般数据是稠密的,那么就是 'elkan',否则就是'full'
K-means结果评估
三种评价函数(无label_true)
-
轮廓系数(Silhouette Coefficient)
-
CH分数(Calinski Harabasz Score )
-
戴维森堡丁指数(DBI)——davies_bouldin_score
参考链接:
- 主要内容来自:K-Means聚类算法原理(刘建平)
- 百度百科
K-means练习sklearn
import numpy as np
import pandas as pd
from sklearn.cluster import KMeans,MiniBatchKMeans
import matplotlib.pyplot as plt
%matplotlib inline
# 构造测试数据
data1 = np.random.normal(-1,0.4,[300,2])
data2 = np.random.normal(0,0.2,[300,2])
data3 = np.random.normal(1,0.2,[300,2])
data4 = np.random.normal(2,0.2,[300,2])
train_data = np.vstack((data1,data2,data3,data4))
print (train_data.shape)
train_data[:5]
# from sklearn.datasets.samples_generator import make_blobs
# # X为样本特征,Y为样本簇类别, 共1000个样本,每个样本2个特征,共4个簇,簇中心在[-1,-1], [0,0],[1,1], [2,2], 簇方差分别为[0.4, 0.2, 0.2]
# X, y = make_blobs(n_samples=1000, n_features=2, centers=[[-1,-1], [0,0], [1,1], [2,2]], cluster_std=[0.4, 0.2, 0.2, 0.2],
# random_state =9)
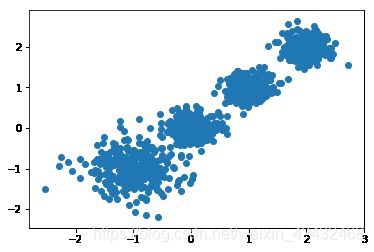
plt.scatter(train_data[:, 0], train_data[:, 1], marker='o')
plt.show();
(1200, 2)
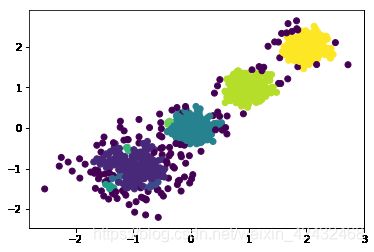
from sklearn import metrics
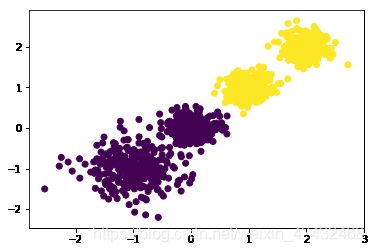
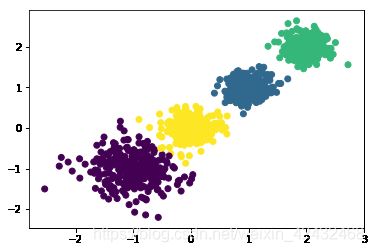
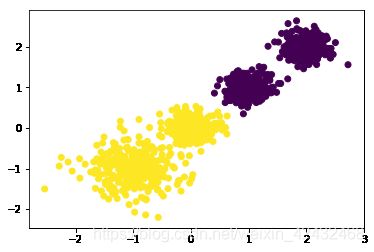
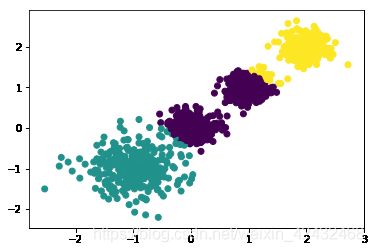
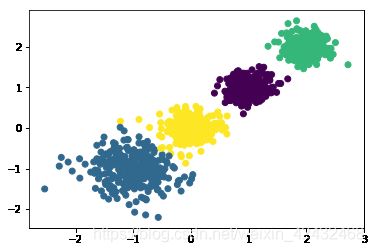
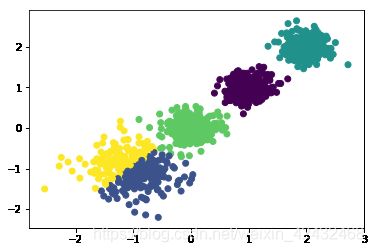
for k in range(2,6):
print ('n_clusters:',k)
y_pred = KMeans(n_clusters=k, random_state=9).fit_predict(train_data)
print (metrics.calinski_harabasz_score(train_data, y_pred)) # 评估聚类分数
plt.scatter(train_data[:, 0], train_data[:, 1], c=y_pred)
plt.show()
n_clusters: 2
3709.87110730457
n_clusters: 3
3637.335907012767
n_clusters: 4
7355.696086246327
n_clusters: 5
6869.280853723166
# MiniBatchKMeans
for k in range(2,6):
print ('n_clusters:',k)
y_pred = MiniBatchKMeans(n_clusters=k, batch_size = 200, random_state=9).fit_predict(train_data)
print (metrics.calinski_harabasz_score(train_data, y_pred)) # 评估聚类分数
plt.scatter(train_data[:, 0], train_data[:, 1], c=y_pred)
plt.show()
n_clusters: 2
3709.87110730457
n_clusters: 3
3635.773385211832
n_clusters: 4
7354.144468603429
n_clusters: 5
6864.799715313496
DBSCAN
- DBSCAN基本概念
\qquad 核心对象:若某个点的密度达到算法设定的阈值则其为核心点。(即邻域内点的个数不少于minPts)
ε \qquad \varepsilon ε邻域的距离阈值:设定的半径 ε \varepsilon ε。
\qquad 直接密度可达:若某点 p p p在 q q q的邻域内,且 q q q是核心点则 p − q p-q p−q直接密度可达。
\qquad 密度可达:若有一个带你的序列 q 0 , q 1 , … , q k q_0,q_1,…,q_k q0,q1,…,qk,对任意 q i − q i − 1 q_i-q_{i-1} qi−qi−1是直接密度可达的,责成从 q 0 q_0 q0到 q k q_k qk密度可达,这实际上是直接密度可达的“传播”。
\qquad 密度相连:若从某核心点出发,点 q q q和点 k k k都是密度可达的,则称点 q q q和点 k k k是密度相连的。
\qquad 边界点:属于某一个类的非核心点,不能发展下线了。
\qquad 噪声点:不属于任何一个类簇的点,从任何一个核心点出发都是密度不可达的。
\qquad 举例说明:A表示核心对象、B和C表示边界点以及N表示离群点。
-
DBSCAN算法流程
1)遍历所有样本,找出所有满足邻域距离e的核心对象的集合 2)任意选择一个核心对象,找出其所有密度可达的样本并生成聚类簇 3)从剩余的核心对象中移除2中找到的密度可达的样本 4)从更新后的核心对象集合重复执行2-3步直到核心对象都被遍历或移除 -
DBSCAN的3个问题
1)一些异常样本点或者说少量游离于簇外的样本点,这些点不在任何一个核心对象在周围,在DBSCAN中,我们一般将这些样本点标记为噪音点。 2)距离的度量问题,即如何计算某样本和核心对象样本的距离。在DBSCAN中,一般采用最近邻思想,采用某一种距离度量来衡量样本距离,比如欧式距离。这和KNN分类算法的最近邻思想完全相同。对应少量的样本,寻找最近邻可以直接去计算所有样本的距离,如果样本量较大,则一般采用KD树或者球树来快速的搜索最近邻。 3)某些样本可能到两个核心对象的距离都小于ϵ,但是这两个核心对象由于不是密度直达,又不属于同一个聚类簇,那么如果界定这个样本的类别呢?一般来说,此时DBSCAN采用先来后到,先进行聚类的类别簇会标记这个样本为它的类别。也就是说DBSCAN的算法不是完全稳定的算法。 -
DBSCAN优缺点
\qquad 优点
相比K-Means,DBSCAN 不需要预先声明聚类数量。
可以对任意形状的稠密数据集进行聚类,相对的,K-Means之类的聚类算法一般只适用于凸数据集。
可以在聚类的同时发现异常点,对数据集中的异常点不敏感。
聚类结果没有偏倚,相对的,K-Means之类的聚类算法初始值对聚类结果有很大影响。
\qquad 缺点
当空间聚类的密度不均匀、聚类间距差相差很大时,聚类质量较差,因为这种情况下参数MinPts和Eps选取困难。
如果样本集较大时,聚类收敛时间较长,此时可以对搜索最近邻时建立的KD树或者球树进行规模限制来改进。
在两个聚类交界边缘的点会视乎它在数据库的次序决定加入哪个聚类,幸运地,这种情况并不常见,而且对整体的聚类结果影响不大(DBSCAN变种算法,把交界点视为噪音,达到完全决定性的结果。)
调参相对于传统的K-Means之类的聚类算法稍复杂,主要需要对距离阈值eps,邻域样本数阈值MinPts联合调参,不同的参数组合对最后的聚类效果有较大影响。
引用自:DBSCAN聚类算法
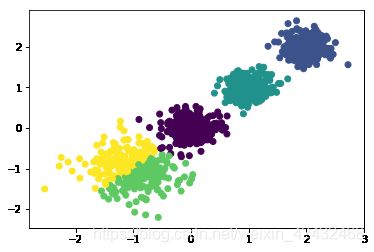
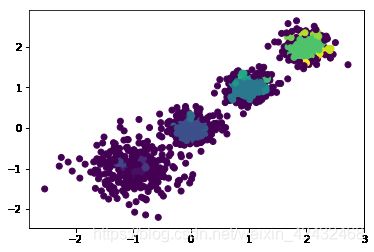
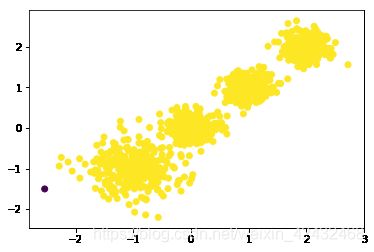
from sklearn.cluster import DBSCAN
for i in [0.05,0.1,0.2,0.5]:
print ('eps:',i)
y_pred = DBSCAN(eps=i, min_samples=5).fit_predict(train_data)
print (metrics.calinski_harabasz_score(train_data, y_pred)) # 评估聚类分数
plt.scatter(train_data[:, 0], train_data[:, 1], c=y_pred)
plt.show()
eps: 0.05
58.86262212608322
eps: 0.1
781.5837222306955
eps: 0.2
2208.5062803223173
eps: 0.5
5.03423151745993