Spark-NLP:大规模自然语言理解
自述
解读分析新项目的需求时,得出这几点要求:
- 需要处理大量的数据
- 需要自然语言处理
- 需要一定的效率
立志要成为一名框架师的我,就思考如何在新项目开启之前,确定需要哪些技术方案,是否要使用新的技术方案,新的技术框架,还是原有的组织技术。之前我个人做过大数据项目,使用的是Hadoop与Spark,也参与过NLP的项目,对此都有些了解,思考如何将这两点结合起来,使用开数据框架,分布式技术,分布在每台机器上,再使用NLP的处理逻辑。Spark流中放入NLP的逻辑处理,再把结果返回汇总,岂不美哉?
在我头疼需要我自己摸索的时候,发现已经有大佬们把这件事情已经做了,将Spark与NLP结果,出了一个Spark-NLP库。
Spark NLP 是一个构建在 Apache Spark ML 之上的自然语言处理库。
并且!重点!!! 最近更新的版本,已经支持Python,无需再学习其他语言了!
官方网站:https://nlp.johnsnowlabs.com/
有官方文档教你如何使用Spark-NLP,后续根据我的学习,出一个相关的介绍!
接下来带大家来看一篇介绍他们的文章《Spark NLP: Natural Language Understanding at Scale》,原文点击链接,就可以下载了!
下面的内容是根据文章翻译过的!
摘要
Spark NLP 是一个构建在 ApacheSparkML 之上的自然语言处理(NLP)库。它为可 以在分布式环境中轻松扩展的机器学习管道提供了简单、性能和准确的 NLP 注释。 Spark NLP 配备了 1100 种+,语言的 192 种+,预训练管道和模型。它支持几乎所有可以在集群中无缝使用的 NLP任务和模块。自 2020 年 1 月以来,Spark NLP 被下载了超 过 270 万次,经历了 9 倍的增长,被 54%的医疗保健组织用作该企业中世界上使用最广泛的 NLP 库。
关键词:火花,自然语言处理,深度学习,张量流,聚类
Keywords: spark, natural language processing, deep learning, tensorflow, cluster
1 Spark NLP库
自然语言处理(NLP)是许多数据科学系统中的一个关键组成部分,它必须理解或 解释一个文本。常见的用例包括问答、转述或总结、情绪分析、自然语言 BI、语言 建模和消除歧义。然而,NLP 总是只是一个更大的数据处理管道的一部分,由于在 这个过程中,越来越需要一体化解决方案来减轻文本预处理的负担大规模和连接点 之间的各个步骤解决数据与 NLP 的科学问题。一个好的 NLP 库应该能够正确地将自 由文本转换为结构化特性,并让用户训练他们自己的 NLP 模型,这些模型可以很容 易地输入下游的机器学习(ML)或深度学习(DL)管道。
Spark NLP 被开发为针对所有 NLP 任务的单一统一解决方案,是唯一可以在任何 Spark 集群中扩展训练和推理的库,利用转移学习和实现最新和最大的库NLP 研究中的算法和模型,同时提供关键的企业级解决方案。它是一个开源的自然 语言处理库,构建在 ApacheSpark 和 SparkML 之上。它提供了一个简单的 API 与 ML 管道集成,并得到了 JohnSnow 实验室公司的商业支持,这是一家屡获殊荣的美国 医疗人工智能和 NLP 公司。
Spark NLP 的注释器使用基于规则的算法、机器学习和深度学习模型,这些模型 使用张量流实现,张量流在准确性、速度、可伸缩性和内存利用率方面进行了大量 优化。此设置已与 ApacheSpark 紧密集成,以允许驱动程序节点使用驱动程序节点 上的所有可用内核运行整个训练。每个张量流组件都有一个 CuDA 版本,可在 GPU 上启用培训模型。SparkNLP 是用 Scala 编写的,它用 Python、Java、Scala 和 R - 提供了开源的 API,因此用户无需了解底层的实现细节(Tensor 流、Spark 等)。为 了使用它。由于它有一个活跃的发布周期(2019 年发布了 26 个新版本,2020 年发 布了 26 个),NLP 领域的最新趋势和研究被快速接受和实现,可以扩展到集群设置, 允许公共 NLP 管道运行比遗留库允许的固有设计限制快一个数量级。
SparkNLP 库有两个版本:开源版和企业版。开源版本使用最新的 DL 框架和研 究趋势,具有所有 NLP 库都可以期望的所有特性和组件。企业图书馆是获得许可的 (免费用于学术目的),旨在解决医疗保健领域的现实世界问题,并扩展了开源版 本。许可版本具有以下模块,以各种方式帮助研究人员和数据从业者:命名实体识 别(NER)、断言状态(否定范围)检测、关系提取、实体分辨率(SNOMED、RxNorm、 ICD10 等。),临床拼写检查,上下文解析器,文本 2SQL,去识别和混淆。每个版本中组件的高级概述(见图 4)。
2 对研究领域的影响
covid-19 大流行带来了关于病毒的学术研究的激增——在 2020 年1 月至 6月[1] 有 23634 份新出版物,从 6 月至 11 月 covid-19 开放研究数据集[2]加速到每周 8800 份。如此大量的出版物使得研究人员无法阅读每一份出版物,导致人们对应用自然 语言处理(NLP)和文本挖掘技术以实现半自动化文献综述的兴趣增加[3]。
与此同时,人们越来越需要对电子健康记录(EHRs)的自动文本挖掘,以找到新 研究指出的临床适应症。EHRs 是临床医生跟踪其患者护理的主要信息来源。输入到 这些系统中的信息可以在以电子方式输入值的结构化字段中找到。但这些记录中的 大多数时间信息都是非结构化的,这使得它在很大程度上无法进行统计分析[5]。 这些记录包括使用药物的原因、病人以前的疾病或过去治疗的结果,它们是生物医 学研究中最大的经验数据来源,允许重大科学发现高度相关的疾病,如癌症和阿尔 茨海默病[6]。尽管 NLP 研究和开创性的兴趣和进展,易于使用生产模型和工具在 生物医学和临床领域稀缺,是临床 NLP 研究人员实现最新算法的主要障碍之一的工 作流,并立即开始使用。另一方面,专门用于处理生物医学和临床文本的 NLP 工具包,如 MetaMap[7]和 cTAKES[8],通常不使用新的研究创新,如上面讨论的单词表 示或神经网络,因此产生不太准确的结果[9,10]。我们引入了 SparkNLP 作为解决 所有这些问题的一站式解决方案。
这种文本挖掘系统中的一个主要基石被称为实体识别(NER)——它被认为是回 答问题、主题建模、信息检索等的重要先驱[11]。在医学领域,NER 识别临床记录 中的第一个有意义的块,然后将其送入处理管道作为后续下游任务的输入,如临床 断言状态检测[12]、临床实体分辨率[13]和敏感数据[14]的去识别[14]。然而,临 床和药物实体的分割在生物医学神经和药物评估系统中被认为是一项困难的任务, 因为命名实体具有复杂的正字法结构[15]。从临床文本中得到的 NER 预测样本(见图 3)。
在临床 NLP 管道中的 NER 模型之后的下一步是根据其上下文为每个命名实体分 配一个断言状态。断言的状态解释了一个被命名的实体(例如。临床发现、程序、 实验室结果)通过给患者指定一个标签,如在场(“患者有糖尿病”)、缺席(“患 者否认恶心”)、有条件(“爬楼梯时呼吸困难”)或与他人相关(“抑郁症家族 史”)。在 covid-19 的背景下,应用准确的断言状态检测是至关重要的,因为大 多数患者将被测试和询问相同的症状和共病——因此将文本挖掘管道限制在没有 上下文的医学术语在实践中是没有用的。此类管道的流程图详(见图 1)。
在我们之前的研究[16]中,我们通过广泛的实验表明,SparkNLP 库中的 NER 模 块超过了 Stanza 报告的 8 个基准数据集中的生物医学 NER 基准,而没有使用像 BERT 那样的重上下文嵌入。使用修改版本的著名的 BiLSTM-CNN-CNrNER 架构[17]火花环 境,我们还提出,即使有了一个通用的手套嵌入(GloVe6B)和没有词汇特征,我们 能够在生物医学领域实现最先进的结果,产生更好的结果比西班牙 48 基准数据集。
在另一项研究[18]中,我们引入了一套预先训练的 NER 模型,这些模型都是在 使用相同的深度学习架构的生物医学和临床数据集上训练的。然后,我们说明了如 何从非结构化电子健康记录(EHR)和 covid-19 开放研究数据集(CORD-19)中提取知 识和相关信息,通过将这些模型结合在一个统一的和可扩展的管道中,并共享结果, 以说明从科学论文中提取有价值的信息。结果表明,CORD-19 中的论文包含了各种 各样的实体类型,这个新的 NLP 管道可以识别到,并且断言状态检测在这些实体上 是一个有用的过滤器(图 2)。所选实体类型中最常见的短语请见表 2。这对于下 游分析的丰富性是很好的兆头,它可以使用这些现在结构化和标准化的数据来完 成——如聚类、维度缩减、语义相似性、可视化或对身份相关概念的基于图的分析。 此外,为了评估管道工作的速度和规模利用计算集群,我们运行在本地模式和预测 管道相同的集群模式:并发现标记化快 20 倍,而实体提取比单机运行集群快 3.5 倍。
3 对工业和学术合作的影响
Spark NLP 的创建者,约翰雪实验室公司一直支持全球研究人员分发免费许可使 用所有许可模块在研究项目和研究生水平课程,在需要时提供实际支持,组织研讨 会和峰会收集杰出的演讲者和运行项目与顶级医药公司的研发团队来帮助他们解 锁潜在的非结构化文本数据埋在他们的生态系统。SparkNLP 已经为领先的医疗保健 和制药公司提供了动力,包括凯撒医疗公司、麦凯森公司、默克公司和罗氏公司。 由于 SparkNLP 也可以离线和部署在空气网络中,公司和医疗机构不需要担心公开受保护的运行状况信息(PHI)。有关这些项目和案例研究的详细信息可在第 21 页中找到。
4 致谢
我们感谢我们的同事和研究合作伙伴,他们为 Spark NLP 库的以前和当前的发展做出了贡献。 我们也感谢我们的用户和客户,他们通过他们的反馈和建议帮助我们改进了图书馆。
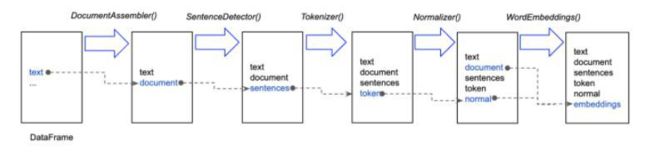
图 1:Spark NLP 管道的流程图。 当我们使用 Spark 数据框在管道上 fit() 时,它的文本列被送入 DocumentAssembler() 转换器和一个新列
文档被创建为任何 Spark 数据帧的 Spark NLP 的初始入口点。 然后,它的文档列被送入 SentenceDetector() 模块以将文本拆分为一个数组
句子并创建一个新列“句子”。 然后,将“sentences”列送入 Tokenizer(),对每个句子进行分词,并创建一个新列“token”。 然后,对令牌进行归一化(基本文本清理)并为每个令牌生成词嵌入。 现在数据已准备好输入 NER 模型,然后输入断言模型。

图 2:命名实体识别是医学文本挖掘管道的基本构建块,并为下游任务提供信息,例如断言状态、实体链接、去标识化和关系提取。
图 3:由在各种数据集上训练的临床 NER 模型预测的样本临床实体。 Spark NLP 企业版中有 40 多个预训练的 NER 模型。

图 4:Spark NLP 库有两个版本(开源和企业),每个版本都有一套预先训练好的模型和管道,无需进一步训练即可开箱即用数据集。

表 1:生物医学领域不同数据集的 NER 性能。 报告的所有分数都是微平均测试 F1,不包括 O。 Stanza 结果来自 [9] 中报道的论文,SciSpaCy 结果来自 [10] 中报道的 scispacy-medium 模型。 合并官方训练集和验证集用于训练,然后在原始测试集上评估模型。 出于可重复性的目的,我们使用 [22] 提供的这些数据集的预处理版本,也由 Stanza 使用。 表中的 Spark-x 前缀表示我们的实现。 粗体分数代表相应行中的最佳分数。

表 2:通过在 Spark NLP 中使用名为 jsl_ner_wip 的 NER 模型解析来自 CORD-19 数据集 [2] 的 100 篇文章预测的所选实体类型中最常见的 10 个术语。 从模型中获得预测,我们可以在不阅读论文的情况下获得有关论文中提到的最常见的疾病或症状或最常见的生命体征和心电图结果的一些有价值的信息。 根据该表,最常见的症状是咳嗽和炎症,而提到的最常见的药物成分是奥司他韦和抗生素。 我们也可以说心源性振荡和心室颤动是心电图的常见观察结果,而发烧和体温过低是最常见的生命体征。
参考文献
[1] J. A. T. da Silva, P. Tsigaris, M. Erfanmanesh, Publishing volumes in major databases related to covid-19, Scientometrics (2020) 1 – 12.
[2] L. L. Wang, K. Lo, Y. Chandrasekhar, R. Reas, J. Yang, D. Eide, K. Funk, R. Kinney, Z. Liu, W. Merrill, et al., Cord-19: The covid-19 open research dataset, ArXiv.
[3] X. Cheng, Q. Cao, S. Liao, An overview of literature on covid-19, mers and sars: Using text mining and latent dirichlet allocation, Journal of Information Science.
[4] A. Liede, R. K. Hernandez, M. Roth, G. Calkins, K. Larrabee, L. Nicacio, Validation of international classification of diseases coding for bone metastases in electronic health records using technology-enabled abstraction, Clinical epidemiology 7 (2015) 441.
[5] T. B. Murdoch, A. S. Detsky, The inevitable application of big data to health care, Jama 309 (13) (2013) 1351–1352.
[6] G. Perera, M. Khondoker, M. Broadbent, G. Breen, R. Stewart, Factors associated with response to acetylcholinesterase inhibition in dementia: a cohort study from a secondary mental health care case register in london, PloS one 9 (11) (2014) e109484.
[7] A. R. Aronson, F.-M. Lang, An overview of metamap: historical perspective and recent advances, Journal of the American Medical Informatics Association 17 (3) (2010) 229–236.
[8] G. K. Savova, J. J. Masanz, P. V. Ogren, J. Zheng, S. Sohn, K. C. KipperSchuler, C. G. Chute, Mayo clinical text analysis and knowledge extraction system (ctakes): architecture, component evaluation and applications, Journal of the American Medical Informatics Association 17 (5) (2010) 507–513.
[9] Y. Zhang, Y. Zhang, P. Qi, C. D. Manning, C. P. Langlotz, Biomedical and clinical english model packages in the stanza python nlp library, arXiv preprint arXiv:2007.14640.
[10] M. Neumann, D. King, I. Beltagy, W. Ammar, Scispacy: Fast and robust models for biomedical natural language processing, arXiv preprint arXiv:1902.07669.
[11] V. Yadav, S. Bethard, A survey on recent advances in named entity recognition from deep learning models, arXiv preprint arXiv:1910.11470.
[12] Ö. Uzuner, B. R. South, S. Shen, S. L. DuVall, 2010 i2b2/va challenge on concepts, assertions, and relations in clinical text, Journal of the American Medical Informatics Association 18 (5) (2011) 552–556.
[13] D. Tzitzivacos, International classification of diseases 10th edition (icd-10):: main article, CME: Your SA Journal of CPD 25 (1) (2007) 8–10.
[14] Ö. Uzuner, Y. Luo, P. Szolovits, Evaluating the state-of-the-art in automatic de-identification, Journal of the American Medical Informatics Association 14 (5) (2007) 550–563.
[15] S. Liu, B. Tang, Q. Chen, X. Wang, Effects of semantic features on machine learning-based drug name recognition systems: word embeddings vs. manually constructed dictionaries, Information 6 (4) (2015) 848–865.
[16] V. Kocaman, D. Talby, Biomedical named entity recognition at scale, arXiv preprint arXiv:2011.06315.8
[17] J. P. Chiu, E. Nichols, Named entity recognition with bidirectional lstmcnns, Transactions of the Association for Computational Linguistics 4 (2016)357–370.
[18] V. Kocaman, D. Talby, Improving clinical document understanding on covid-19 research with spark nlp, arXiv preprint arXiv:2012.04005.
[19] J. S. Labs, Apache Spark NLP for Healthcare: Lessons Learned Building Real-World Healthcare AI Systems, https://databricks.com/session_na20/apache-spark-nlp-for-healthcare-lessons-learned-building-real-world-healthcare-ai-systems,[Online; accessed 22-Jan-2021] (2021).
[20] J. S. Labs, NLP Case Studies, https://www.johnsnowlabs.com/nlp-case-studies/, [Online; accessed 22-Jan-2021] (2021).
[21] J. S. Labs, AI Case Studies, https://www.johnsnowlabs.com/ai-case-studies/, [Online; accessed 22-Jan-2021] (2021).
[22] X. Wang, Y. Zhang, X. Ren, Y. Zhang, M. Zitnik, J. Shang, C. Langlotz, J. Han, Cross-type biomedical named entity recognition with deep multi-task learning, Bioinformatics 35 (10) (2019) 1745–1752.