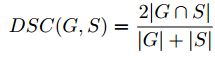AbdomenCT-1K: Is Abdominal Organ Segmentation A Solved Problem?
https://arxiv.org/abs/2010.14808
https://github.com/JunMa11/AbdomenCT-1K
AbdomenCT-1K: Is Abdominal Organ Segmentation A Solved Problem?
摘要:
在许多基准数据集上,看起来腹部主要器官的自动分割已经解决了。但实际上现有的腹部基准数据集都只包含single-center、single-phase、single-vendor、或single-disease,其他情况数据集上的泛化就不确定了。AbdomenCT-1k有超过1k 个来自12 medical centers的CT数据,这些数据包含multi-phase、multi-vendor和multi-disease cases。该数据集标注的器官是liver、kidney、spleen和pancreas,并在fully supervised、semi-supervised、weakly supervised和continual learning构建了对应的four organ segmentation benchmarks。
一、引言
器官分割之所以比较难。首先,因为软组织对比度通常比较低。齐次,有些器官存在复杂的形态学结构和异质性的病变(heterogeneous lesions)。另外,不同的扫描器和CT phases也会导致器官成像的显著差异。
图1、multi-center、multi-phase、multi-vender(多厂家)、multi-disease的腹部器官样例
最近的研究中Liver的DSC(Dice similarity coefficient)95%、Spleen的DSC是96.2%,以上结果都是在single-center、single-phase、single-vendor、和single-disease case,也就是说在其他多变的数据集上泛化能力就不可知了。所以作者才有了is abdominal organ segmentation a solved problem?
为此作者构建了一个large和diverse腹部CT器官分割数据集,AbdomenCT-1K。然后基于此数据集分析了现有解决方案的局限性。最后提四个benchmarks。
1.1现有腹部器官分割方法和基准数据集的局限性
实际的临床分割算法不应当只关注高精度,也应该关注在不同数据来源的泛化能力。当前的methods和benchmarks还存在一些局限性。
1)缺乏一个大规模和多样化的数据集。需要一个大规模、多样性的数据集评价算法的泛化能力。表1中可以看出,这些数据集要么比较小,要么来自于单一的医学中心。
表1:公开的腹部CT基准数据集
2)对SOTA方法没有一个综合行的评价。大多数存在的方法都关注于全监督学习,这些方法的训练和评价也都是在一个非常小的公开数据集上进行的。那这些方法在其他不同医学中心测试数据上的方法能力就不能确定了。
3)对最近出现的annotation-efficient分割任务缺乏benchmark。这些annotation-efficient方法比如无标记和弱标记学习。
4)对器官boundary-based的评价度量缺乏关注。许多的方法只关注于region-based度量(DSC),但是在临床上,边界准确性也很重要。
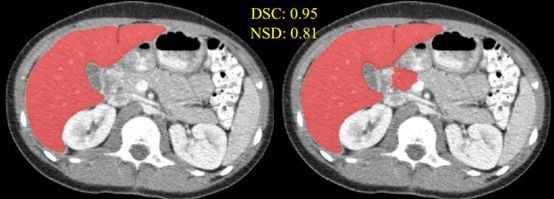
图5、对比Dice similarity coefficient(DSC)和normalized surface Dice
1.2论文贡献
对现存benchmark数据集进行扩展,如LiTS、MSD、KiTS、NIHPancreas等,创建一个大尺度腹部多器官CT数据集。也就是AbdomenCT-1K,里面包含了来自12个multi-center、multi-phase、multi-vendor和multi-disease的1112个CT扫描,这里面标注了liver、kidney、spleen、和pancreas。在表1和图3中可以看出原有数据集和扩增数据集的差异。为了回答腹部器官分割是不是已解决这个问题,作者使用了nnUnet作为分割方法。除了DSC外,另外追加了基于boundary的评估方法NSD(normalized surface Dice),主要是临床上对器官边界分割效果要求很重要。简单的情况下腹部器官分割可以说已经解决了,但实际临床上,腹部多器官分割依旧是一个未解决的问题。假如说测试数据来自于一家新的医学中心或者包含了一些未训练过的疾病。因此在第四节中揭示了为什么现有benchmark不能反映临床数据面临的挑战。因此作者基于AbdomenCT-1K同时提出4个benchmark,fully supervised learning、和三种annotation-efficient学习方法:semi-supervised learning、weakly supervised learning和continual learning。图2介绍了这4个benchmark的概述。
图2:全监督:①所有的训练数据都有标签。②主要在multi-center、-phase、-vendor和-disease上对其泛化能力进行评估;半监督:①训练集中有一部分是包含完整标记的,另一部分是未标记过的数据,②主要是为了评估使用未标记数据训练的效果;弱监督:①部分标记可能不全,例如间隔几层slice就漏标了。②主要是评价弱标签的能力;连续学习:①不同的数据集上包含不同的标签。②主要是评价模型学习新任务的扩充能力,也就是不遗忘之前学习到的。
图3、原始数据集和作者扩增的腹部数据及,liver(红色)、kidney(绿色)、spleen(蓝色)、pancreas(黄色)
主要的贡献:
- 构建了已知范围内最大的腹部多器官数据集AbdomenCT-1K,它包含来自12个医学中心的multi-phase、multi-vendor和multi-disease的1112个CT扫描,以及4446个器官勾画(liver、kidney、spleen和pancreas),比已知的数据集都要明显大得多。更重要的是,该数据让更多的研究者注意到了模型的泛化能力,这种泛化能力在临床上十分重要。
- 在此数据集上利用nnU-Net方法对liver、kidney、spleen、pancreas进行分割训练,并发现了一些已经解决和一些尚未解决的问题。
- 首次创建了四个新的腹部多器官分割基准,全监督、半监督、弱监督和连续学习。这些benchmarks可以为腹部多器官分割方法提供一个标准、公众的评价。并且使用SOTA方法(nnUnet)为每个任务提供了开箱即用的baseline解决方案。数据集、代码和训练模型可以从https://github.com/JunMa11/AbdomenCT-1K下载下来。
第2节,对现有的腹部器官分割方法和数据集简单的介绍。第3节,介绍创建的AbdomenCT-1K,第4节介绍利用SOTA方法nnU-Net在该数据集上的研究,并对已经解决和未解决的问题进行阐述。第5节介绍了腹部器官分割的fully supervised、semi-supervised、weakly supervised和continual learning方法。第6节结论。
2、相关工作
2.1腹部器官分割方法
腹部器官分割方法可以分为经典的基于模型的方法和现在的基于学习的方法。
基于模型的方法:通常将图像分割描述为energy functional最小化问题、shape template匹配或者是atlas方法,例如variational models、statistical shape models和atlas-based 方法。
Variational model:level set或者active contour models,提供了一种natural方式驱使曲线勾画出感兴趣的曲线。
Statistical shape models:active shape model,用一组边界点来表述目标形状,这些点又被点分布模型约束着。然后这些模型开始迭代形变这些点,用来使用新图像中的目标。
Atlas-based方法通常用标注的case来构建一个或者多个器官多atlas图谱,然后在利用atlas图像和target图像配准,将atlas标记融合到target图像。
虽然基于模型的方法有透明的原则和明确的公式,但是他们没办法分割出边界较弱或者对比度较低的图像。对于3D的CT扫描而言,这些方法的计算代价通常高昂。
基于学习的方法:通常从标注的图像中提取鉴别特征,以区分目标器官和其他组织。2015年的时候CNN方法已经引入到了腹部器官分割,该方法不依赖手工特征,也不依赖解刨对应关系,达到了SOTA性能。基于学习的方法又可以氛围supervised learning和annotation-efficient方法。
一类是单器官的监督分割方法,例如修改U-Net来分割liver和liver tumors。再比如shape-aware方法,该方法将目标器官的先验知识融入到CNN的backbone中,在liver分割任务中取得良好的性能。U-Net是一种很受欢迎的网络结构,其他的backbone也被提出并用在腹部器官分割中,例如progressive holistically-nested network(PHNN)和progressive semantically-nested networks(PSNNs)。相比liver分割而言,pancreas分割更具挑战,因此two-stage cascaded方法被提出,首先是pancreas的定位,然后一个新的网络来refine分割。另外一个level set regression网络来获取更精确的pancreas边界。入了一些经验设计网络结构外,通过设计高效可微神经结构搜索策略,Neural Architecture Search(NAS)神经结构搜索技术也被引入到器官分割当中。
另一类就是多器官的监督分割方法,早期就是单独利用FCN,后来的话慢慢的加入了预处理和后处理。相比单器官分割任务,多器官分割任务更具挑战性。如图1 所示,器官之间边界较弱,且不同器官存在大小差异。为了解决这个问题级联的网络被创建。Two-stage分割方法,第一个阶段首先计算器官分割的概率图,然后和原始图像组合进行第二阶段,segmentation probability map为第二阶段提供了spatial attention,从而在第二阶段增强目标器官的鉴别信息。还有一些其他的策略,主要是第一阶段网络扮演不同的角色。例如在第一阶段生成一个candidate region,然后送入第二阶段。或者第一阶段提取低分辨率的分割图。另有结论证明第一阶段网络中的中间层的特征可以为第二阶段提供有用信息。因此block level skip connections(BLSC)块级跳跃连接的across cascaded VNet,并且能够有效的提升性能。另外有些方法为了减少架构层数、核大小等方面的选择,提出了可学习的滤波系数和空间偏移的可训练3D卷积核,并展示了其在捕获大空间纹理及网络设计方面的优势。nnUnet在单器官和多器官(liver、kidney、pancreas和spleen)分割任务上都取得了state-of-the-art性能。
Annotation-efficient methods:semi-supervised learning、weakly supervised learning和continual learning得到了很多关注,主要是因为完整的标注数据获取起来实在太难了。因此除了fully supervised器官分割外,partially labelled受到了很多关注。
Semi-supervised learning主要的目的是将少量的有标记数据和大量的无标记数据相结合,是从无标记数据中挖掘知识的有效手段途径。在半监督方法中,基于伪标签的方法被认为是简单有效的解决方案。一个基于伪标签的半监督多器官分割方法,首先会在源数据集上训练一个teacher model,然后在使用该teacher model在未标记的数据集上分割出伪标签,最后将标记的数据和未标记的数据组合训练。另外还会利用其他的策略,例如除了从标记数据中计算dice loss之外,还会有一个assurance-based discriminator模块来监督对未标记的数据的学习。一个co-training strategy被提出用在无标签数据上。该框架可以在small single phase数据集上训练,也可以适应无标记的multi-center和multi-phase临床数据上。An uncertainty-aware multi-view co-training(UMCT)一种不确定性感知的多视图联合训练方法在多器官和pancreas数据集上取得了很好的性能。
Weakly supervised learning主要是对若标签的探索,例如slice-level annotations、sparse annotations、noisy annotations。在器官分割方面,一种用于livers、spleens、kidneys的classification forest-based weakly supervised器官分割方案的提出,这里面的标签都是在器官上的涂鸦。除此之外,image-level labels-based pancreas segmentation也在被探索。虽然weakly supervised learning在腹部器官分割研究上还十分有限,但在CV社区对于不同若标签分割有大量的研究,例如bounding boxes、point、scribbles、image-level labels。
Continual learning是在学习新任务的同时,不遗忘以前学习过的任务。也称之为life-long learning,incremental learning or sequential learning。虽然深度学习方法在很多引用获得了SOTA性能,但是神经网络会遭受灾难性的遗忘或者干扰。训练模型时使用新信息会干扰模型已经学习到的知识。所以旧任务性能可能会下降。在object recognition和classification中continual learning越来越受到关注。另外在计算机视觉界也提出了针对continual learning的数据集和benchmarks。例如object recognition dataset and benchmark CORe50,iCubWorld datasets和CVPR2020 CLVision challenge。但是在腹部器官分割里面好像还没有一个像样的continual learning相关的工作,因此利用这一新兴技术来处理器官分割任务还是有很大的需求。
2.2现有的腹部分割基准数据集
除了腹部器官分割的方法在发展以外,相应的benchmark数据集也在发展,比如越来越多的标注数据。表1展示了一些2010年以来的腹部器官CT分割基准数据集,接下来就会简单的介绍。
BTCV(Beyond The Cranial Vault)benchmark 数据集包含50个腹部数据,由Vanderbilt University Medical Center从转移性肝癌患者或术后腹疝患者中采集,该benchmark数据集有13个器官:spleen、right kidney、left kidney、gallbladder、esophagus、liver、stomach、aorta、inferior vena cava、portal vein and splenic vein、pancreas、right adrenal gland、and left adrenal gland。这些器官由两名经验丰富的本科生手工标记,并由放射科医生进行验证。
NIH Pancreas dataset,由 US National Institutes of Health(NIH) Clinical Center采集,包含80组增强CT,这些CT扫描的大小是512x512,spacing是变化的,层厚1.5~2.5mm。这里面17名是健康的肾脏捐赠者,剩余的65名是由放射科医生从没有腹部病变和胰腺癌变的患者中挑选出来的。这些胰腺器官,由一名医学生手动一层一层画出来的,然后由经验丰富的放射科医生进行验证/修改。
VISCERAL Anatomy Benchmark由120个CT和MR组成,来自4种不同的成像方式和视野组成,每一组有20个,加载一起就是80个。这写数据被手动勾画,建立起一个standard Gold Corpus,一共有1295个结构和1760个landmarks。
LiTS(Liver Tumor Segmentation)数据集包含131个liver和liver tumor训练数据,70个测试数据,in-plane分辨率是0.5~1.0mm,层厚是0.45~6.0mm。这些数据从7个医学中心收集,包含了各种原发癌症,如结肠直肠癌、乳腺癌、肺癌的转移性肝病。放射科医生对肝脏和肿瘤进行标注。
MSD(Medical Segmentation Decathlon)pancreas dataset 有281个pancreas和tumor标注的训练数据,以及139个测试数据。数据集由Memorial Sloan Kettering Cancer Center(New York, USA)提供。本组患者接受了胰腺肿物切除术,包括导管内黏液性肿瘤、胰腺神经内分泌肿瘤或胰腺导管腺癌,胰腺实质和胰腺肿块(囊肿或肿瘤)由腹部放射科专家在每个切片上手工标注。
MSD Spleen dataset包含41个有spleen标注的训练数据,和20个测试数据。同样由Memorial Sloan Kettering Cancer Center(New York, USA)提供。该数据集中的患者接受了肝转移的化疗。Spleen采用基于水平集level-set-based的方法半自动分割semi-automatically,然后由腹部放射专家手动调整。
Multi-organ Abdominal CT Reference Standard Segmentations有90组数据8个器官组成。这些数据从Cancer Imaging Archive(TCIA) Pancreas-CT和Beyond the Cranial Vault(BTCV)(除了duodenum), 未分割的器官由一名影响研究员在一名认证放射科医生的监督下手工标记的。
CHAOS(Combined Healthy Abdominal Organ Segmentation)由20个liver标记的训练数据和20个测试数据组成,有Dokuz Eylul University(DEU) hospital(Izmir, Turkey)。不同于其他数据集,这里面的40个liver数据来自于健康的人群。
KiTS(Kidney Tumor Segmentation)包含210个kidney和kidney tumor标注的训练数据和90个测试数据。有University of Minnesota Medical Center(Minnesota, USA)提供。本数据集中的患者因一个或多个肾脏肿瘤接受了部分或根治性肾切除术。肾脏和肿瘤标注由医学生在临床主任的监督下提供。
CT-ORG是一个包含140个CT图像的多样化数据集,包含6个器官分类,其中131个是专门的CT和9是PET-CT检查的CT成分。这些CT图像来自8个不同的医疗中心。根据一个或多个标记器官的病变情况纳入患者。大多数图像显示肝脏病变,包括良性和恶性的。
3 ABDOMENCT-1K
3.1数据集动机和详细信息
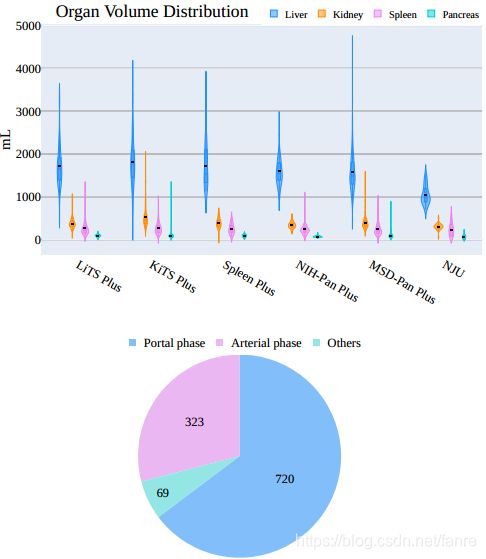
一般的数腹部器官分割数据集在多样性和规模上都存在一定的局限性。而这里提供的数据集更接近于真实场景,规模更大且多样性。跟关注于多器官分割,如liver、kidney、spleen和pancreas。这里面还包含了更多的疾病情况,AbdomenCT-1K,有1112个CT扫描组成,这些数据来自于5个公开的数据集:LiTS(201)、KiTS(300)、MSD Spleen(61)、Pancreas(420)、NIH Pancreas(80)以及从南京大学采集的50组数据。来自南京大学数据集的50张CT扫描图胰腺癌20例,结肠癌20例,肝癌10例。Plain phase平相、artery phase动脉相和protal phase门脉相扫描数分别是18、18和14。CT扫描的分辨率是512×512,spacing大小任意,层厚在1.25- 5mm之间,通过GE多探测器螺旋获取CT。NIH Pancreas and KiTS dataset的许可分别是 Creative Commons license CC-BY和CC-BY-NC-SA 4.0。LiTS, MSD Pancreas, and MSD Spleen datasets知识共享许可Creative Commons license CC-BY-SA 4.0。在这些许可下,我们可以修改数据集,并以任何格式共享或重新分发它们。
原始数据集只提供单个器官的注释,而我们的数据集包含每个数据集中所有情况下的四个器官的注释,如图3所示。为了与原始数据集区分,我们将多器官注释称为plus数据集(例如,多器官LiTS数据集称为LiTS plus数据集)。图4显示了AbdomenCT-1K的器官体积和对比相分布。其他信息(如CT扫描仪、Hounsfield单位(HU)值的分布、图像大小和图像间距。)在补充(补充表1)中。
图4:器官volume和contrast phase在AbdomenCT-1K上的分布
3.2标注
如果现有数据存在标注,那就用以前的标注,如果没有的话,就用训练过的单器官模型来预测,然后,然后,15名初级注释员(1至5年经验)在两名经过认证的放射科医生的监督下,使用ITK-SNAP 3.6手工细化分割结果。最后,由一位具有10年以上经验的资深放射科医师对注释进行验证和完善。所有的注释都应用于axial图像。为了降低评分者之间注释的可变性,我们引入了三种层次策略来提高标签的一致性。具体地说:
- 在标注之前,所有的评分者都需要学习现有的器官标注协议,以确保评分者与现有数据集的标注协议一致;
- 在标注时,对现有数据集中明显的标注错误进行修正,最终由一位经验丰富的资深放射科医师(腹部专业10年以上)对所有标注进行检查和修改;
- 经过注释,我们训练了five-fold U-Net模型,找出可能的分割错误。低DSC或NSD评分的病例由资深放射科医师进行复核
另外,我们邀请两位放射科医生在南京大学数据集中选取了50个病例进行标记,并在表2中显示了它们的inter-rater variability。
表2:两名放射科医生评分者间差异的定量分析。
3.3Backbone network
虽然有很多UNet存在很多的变体,但实际上如果相应的pipeline设计得当,基本的U-Net网络依旧难以超越。特别是,nnU-Net(no-new-U-Net)在给定的数据集提出自适应预处理策略和网络架构(即池数、卷积核大小和步数)。无需手动调优,nnU-Net在19个公开的国际细分比赛中,比大多数专门的深度学习管道,取得更好的性能,并在49个任务中设置新的SOTA。目前,nnU-Net在许多分割任务中仍然是SOTA方法。因此这里的backbone网络依旧是nnU-Net,具体来说,batch size=2、随机梯度下降做为优化器,并且初始学习率是0.01、nesterov momentum是0.99。为了避免过拟合,在训练的时候使用标准的数据增强,例如旋转、放缩、高斯噪声、伽马校正等。损失函数是Dice和cross-entropy的组合,因为这种组合方式有很好的鲁棒性。训练在NVIDIA TITAN V100或2080Ti GPUs上进行1000次迭代。
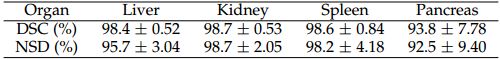
3.4评价度量
一个是region-based的Dice similarity coefficient(DSC),用来评价region overlap。另一个是boundary-based的Normalized surface Dice(NSD)用来评价在指定tolerance τ下两个surface到底有多接近。这些度量都在[0,1],高分代表高性能。G、S分别代表ground truth和segmentation result。|∂G|和|∂S|代表分割的voxels个数。
![]()
![]()
![]()
分别代表ground truth和segmentation surface在tolerance τ下的边界区域。文章中tolerance τ=1mm。
一般都是用DSC来做评价,NSD是可有作为其补充,图5给出了一个肝脏分割的例子说明NSD的特性,肝脏右侧边界存在明显的分割误差。但是DSC得分仍然很高,不能很好地反映边界误差,而NSD对这个边界误差很敏感,因此得分较低。在许多临床任务中,如术前计划和器官移植,边界误差是至关重要的,因此应该被取出。引入NSD的另一个好处是,它忽略了小的边界偏差,因为小的观察者间误差也是不可避免的,而且在放射科医生进行器官分割时往往与临床无关。http://medicaldecathlon.com/files/Surface_distance_based_measures.ipynb
图5对比Dice similarity coefficient(DSC)和normalized surface Dice(NSD)
4全监督器官分割的大规模研究
腹部器官分割是目前流行的分割任务之一。现有benchmark主要集中在fully supervised的分割任务上,并且建立在训练用例和测试用例来自同一个医疗中心的单中心数据集上,最先进的(SOTA)方法(nnU-Net)取得了非常高的准确性。在本节中,我们将评估plus数据集上的SOTA方法,以说明性能是否可以推广到多中心数据集
4.1单器官分割
现有的腹部器官分割基准主要是单器官分割,如KiTS、MSDSpleen和NIH Pancreas分别只关注kidney、spleen、pancreas分割。这些基准中的训练和测试集都来自于同一个医疗中心,目前的SOTA方法在一些分割任务(kidney、spleen、pancreas)中准确性(从DSC角度)已经达到了人类水平。然而,目前还不清楚这种出色的性能是否能推广到来自第三方医疗中心的新数据集。在本小节中,我们在原始训练集中随机选择80%的case进行训练,其余20%的case和三个新数据集作为测试集,这样可以进行数据集内和数据集间的定量比较。
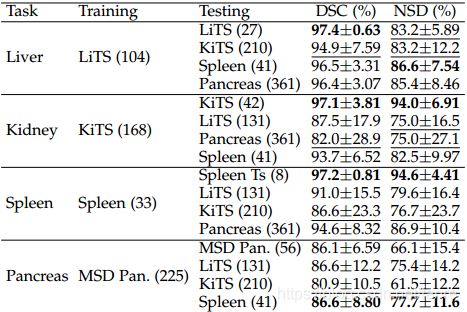
表3:单器官分割量化结果。每个分割任务都有一个来自与训练集相同数据源的测试集和三个来自新医疗中心的测试集。粗体和下划线的数字分别表示最好和最差的结果。
图6:单器官分割任务中不同器官的分割性能(DSC和NSD)Violin图
表3为各器官的量化的分割结果,图6为相应的violin图。可以发现:
- 在liver分割中,SOTA方法在三个新的测试数据集上取得了94.9% ~ 96.5%的DSC分数,表明了其良好的泛化能力。与LiTS (27)的DSC分数相比,KiTS(210)的DSC分数下降了2.5%。主要原因是KiTS(210)的CT扫描是在 arterial phase 获得的,而LiTS的大部分CT扫描是在portal phase获得的。Pancreas(361)和Spleen(41)的DSC评分与LiTS(27)比较接近,但NSD评分要好得多,说明LiTS(27)的分割结果在边界附近误差较大。这是因为,LiTS大部分患者都是肝癌,而Pancreas(361)和Spleen(41)数据的肝脏是正常的。
- 对于kidney的分割,与在KiTS(42)上DSC和NSD评分较高,在其他三个数据集上的性能显著下降,DSC和NSD分别高达15%和19%,尤其是LiTS(131)和Pancreas(361)。主要原因是其他三个数据集中大多数病例的CT phases与KiTS不同。
- 在spleen分割中,其他三个数据集的DSC和NSD评分也有所下降,尤其是KiTS(210)数据集,DSC和NSD评分分别下降了10.6%和17.9%,说明SOTA方法在不同spleen 的泛化效果不佳。
- 对于胰腺分割,由于CT phases的不同,KiTS(210)的表现也有显著下降。值得注意的是,与MSD Pan(56)相比,LiTS(131)和Spleen(41)获得了相似的DSC分数,但NSD评分有很大的改善,分别为9.3%和11%,因为这两个数据集中的大多数病例都是健康的胰腺。结果表明,胰腺分割模型对胰腺健康病例的泛化效果优于胰腺病理病例,尤其是基于边界的NSD度量。
综上所述,当前的SOTA单器官分割方法在训练集和测试集来自同一分布时可以达到非常高的性能(尤其是对DSC),但当测试集来自新的医疗中心时,性能会下降。
4.2多器官分割
在这一小节中,我们重点评价了SOTA方法(nnU-Net)在多器官分割任务中的泛化能力。具体来说,我们进行了四组实验。在每一组中,我们在一个带有四种器官注释的数据集上训练nnU-Net,并在另外三个新数据集上测试训练模型。需要注意的是,每组的训练集和测试集来自不同的医疗中心。
表4:全监督多器官分割的平均DSC和NSD定量结果。粗体和下划线的数字分别表示最好和最差的结果
表4显示了每个器官的定量分割结果10。可以看出。
- 在liver和脾脏的spleen结果中DSC评分比较稳定,在所有实验中均达到90%+。而NSD评分在不同测试集之间波动较大,spleen为77.4% ~ 92.1%,spleen为86.0% ~ 97.0%。
- 不同测试集的kidney分割结果中,DSC和NSD评分差异很大。例如,在第一组实验中,nnU-Net在Pancreas Plus(361)和Spleen Plus(41)数据集上的平均肾脏DSC得分分别为96.0%和85.6%,NSD得分分别为92.4%和78.9%,性能差距为10%+
- 胰腺分割结果在所有实验中都低于其他器官,说明胰腺分割仍然是一个具有挑战性的问题。
图7展示了一些分割良好的案例和具有挑战性的案例。可以观察到,分割良好的案例,器官边界清晰,对比良好,器官内无严重伪影或病变。与分割良好的案例相比,挑战性的案例通常有异质性病变,如肝脏病变(图7 (d)-第一行)和胰腺病变(图7 (d)-第三行)。此外,噪声还会降低图像质量,如图7 (d)-第二行所示。
图7:在大规模的全监督多器官分割研究中,来自测试集的良好分割和具有挑战性的例子。
4.3腹部器官分割是一个解决的问题吗?
如果认为对liver、kidney和spleen这是一个已解决的问题,那么需要满足以下条件:
- 使用DSC评价,主要关注region-based分割错误
- 训练集和测试集数据分布一致。
- 测试集中的案例是微不足道的,也就是说案例中没有严重疾病或者图像质量较低。
作者认为腹部器官分割还是一个未解决的问题,注意是因为:
- 使用NSD评价,主要关注boundary-based分割错误
- 测试集来自新的医疗中心,其数据分布与训练集不同
- 测试集的样本有看不见的或严重的疾病,图像质量低,如异质病变、噪声等,而训练集没有或只有很少的类似样本。
如2.2节所述,现有的腹部器官分割基准无法反映这些具有挑战性的情况。因此,在这项工作中,我们建立了新的细分基准,可以覆盖这些挑战。现有的基准在社区中得到了广泛的关注,在当前的测试集和相关的评估度量(即DSC)中几乎没有改进的空间。因此,我们希望我们的新细分基准将带来新的见解,并再次引起广泛的关注
5.在全监督、半监督、弱监督、连续学习上新的腹部ct器官分割基准
我们新的腹部器官分割基准旨在包括更具挑战性的设置。我们特别关注:
- 不仅要评估区域相关的分割误差,还要评估边界相关的分割误差,因为边界误差在许多临床应用中是至关重要的,如器官移植的手术计划。
- 评估分割方法对新medical centers and CT phases样本的泛化能力。评估分割方法对新medical centers and CT phases样本的泛化能力。
- 评估分割方法对未见和重症疾病样本的泛化能力
除了完全监督分割基准,我们还建立了我们所知的第1个针对半监督学习、弱监督学习和持续学习的腹部器官分割基准,这些都是目前活跃的研究课题,可以减少对注释的依赖。
在每个基准测试中,我们选择50个挑战案例和50个随机案例作为测试集,这对未来用户评估他们的方法是友好的,因为它在推理过程中不会花费太多的时间。更重要的是,最后的表现不容易被容易的情况所偏颇。我们还引入了一个新的数据集作为公共测试集,它可以在四个基准之间进行同一基准的比较。此外,对于每个基准,我们已经用SOTA方法开发了一个强大的基线,这可以成为对这些任务感兴趣的研究人员的一种开箱即用的方法。
5.1完全监督腹部器官分割基准
全监督分割是一个长期而流行的研究课题。在这个基准中,我们专注于多器官分割(肝、肾、脾、胰腺),旨在解决第4节中大规模研究中未解决的问题。
5.1.1任务设置
训练集和测试集的选择动机: 一个在完全监督的器官中,通常需要大量的训练集分割。因此,我们选择 MSD Pan. Plus (281)作为训练集中的基础数据集,因为它拥有最多的训练案例。在MSD Pan. Plus (281)基础上,将不同的案例添加到训练集,构建两个子任务,如表5所示。
- 子任务1。训练集由MSD Pan. Plus (281)和NIH Pan. Plus (80) 组成,所有CT扫描都来自 portal phase。我们使用5.1.2节中的基线模型来预测LiTS Plus, KiTS Plus, and Spleen Plus中所有剩余的case。然后选取DSC和NSD平均值最低的50个case作为测试集。这些cases通常异构性病变,边界不清,分割难度大,但在临床实践中也很重要。
- 子任务2。 NIH Pan. Plus (80)被LiTS Plus的40个案例和KiTS Plus的40个案例所取代,这些案例与测试集具有相似的phases。通过这种方式,我们可以评估在训练和测试集中包含 shared contrast phases是否能够提高性能。
我们使用5.1.2节中的基线模型来推断所有剩余的案例,并选择100个案例作为最终的测试集,其中包括平均DSC和NSD得分最低的50个具有挑战性的案例,以及50个随机选取的案例。最重要的是子任务上的训练集和测试集是没有交集的。
表5:全监督多器官分割基准的任务设置和定量基线结果。
5.1.2基线和结果
基线建立在3D nnU-Net上,这是一种用于多器官分割的SOTA方法。表5列出每一子任务各器官的详细结果。可以发现,由于子任务2中引入了shared contrast phases的情况,子任务1中所有器官的性能都低于子任务2中的性能。虽然完全监督的腹部器官分割似乎已经解决了问题(如肝、肾和脾的分割),因为SOTA方法已经达到了专家间的精度,但我们在一个大型和多样化的数据集上的研究表明,腹部器官分割仍然是一个未解决的问题,尤其是在有挑战性的情况下。
每个器官的violin图见补充图2。对于DSC分数,虽然肝脏分割violin图的高DSC分数和低离散分布,表明了良好的性能,但其他器官结果退化。对于NSD分数,从得到的分数和从violin图中观察到的离散分布表明四个器官的分割性能都不理想。值得指出的是,对于肝脏分割,两个子任务的DSC得分都在95%以上,表明在ground truth与被分割区域的区域重叠方面,分割性能很好。两个子任务的NSD得分分别为83%和85.8%,说明边界区域存在更多的分割错误,需要进一步改进。这一现象进一步证明了应用NSD评价分割结果的必要性。
图8显示了每个子任务中一些具有挑战性的例子的分割结果。可见,SOTA法不能很好地推广到病变器官。例如,图8中的第一行显示了脂肪肝的病例,其中肝脏比健康病例的颜色更深。SOTA法不能完全分割肝脏。脾(蓝色)分割结果在这种情况下也很差。此外,第二,第三和第四行分别为肾(绿色)和脾(蓝色)肿瘤。在分割结果中存在严重的欠分割和分割错误。这些具有挑战性的案例仍然是腹部器官分割未解决的问题,在目前公开的基准中没有突出。
图8:完全监督分割基准测试集的挑战性示例。
5.2半监督腹部器官分割基准
半监督学习是利用无标记数据、减少标注需求的有效方法,是当前研究的热点。在自然图像/视频分割领域已经有几个基准[94],[95]。然而,医学图像分割领域还没有相关的基准。因此,我们建立了这个基准来探索如何使用未标记的数据来提高腹部器官分割的性能。
5.2.1任务设置
训练集和测试集的选择动机: 该半监督任务使用MSD Spleen Plus, LiTS Plus, KiTS Plus, MSD Pancreas Plus, and NIH Pancreas Plus作为训练和测试数据集。半监督任务致力于减轻手工注释的负担。在这个场景中,有一小部分标记数据和大量未标记数据可用。因此,我们将最小的子集Spleen Plus(41例)作为标记的训练集。为了显示半监督方法在利用大量未标记数据方面的优越性,从剩余子集中选择大约10-20倍的数据量(400-800例)作为未标记训练集。我们使用Section 5.2.2中的基线模型来推断所有剩余的案例,并选择100个案例作为最终的测试集,其中包括平均DSC和NSD得分最低的50个具有挑战性的案例,以及50个随机选取的案例。
表6给出了由2个子任务组成的半监督分割基准设置。作为对比,我们从完全监督的下界任务开始,其中模型仅在包含41个标注良好的MSD Spleen Plus 案例上训练。上界任务也受到完全监督,涉及额外的800个标记案例。准确地说,上界训练集中,来自 MSD Spleen Plus的41例,来自MSD and NIH Pancreas Plus的400例,来自LiTS Plus的145例,来自KiTS Plus的250例,来自 MSD Spleen Plus测试集的5例。在下界子任务和上界子任务的基础上,在接下来的半监督子任务中逐渐引入了不带标签的情况。为了评估未标记数据及其数量对多器官分割的影响,我们精心设计了关于未标记数据来源和数量的2个子任务。具体来说,子任务1利用了来自MSD and NIH Pancreas Plus的400例未标记病例,此外,子任务2利用了来自 LiTS plus, KiTS plus, and MSD Spleen Plus 测试集的400例未标记病例。两个子任务在一致测试集上进行评估,以进行公平的比较
5.2.2基线和结果
受半监督图像分类noisy-student learning method 和半监督城市场景分割任务的成功启发,我们开发了一种noisy-student learning method 的半监督腹部器官分割方法,包括五个主要步骤:
- 步骤1。在手工标记的数据上训练一个教师模型。
- 步骤2。通过教师模型生成未标记数据的伪标签。
- 步骤3。在手工和伪标记数据上训练学生模型。
- 步骤4。在步骤3中对手工标记的数据微调学生模型。
- 第5步。回到第2步,将教师模型替换为学生模型,并根据所需次数进行迭代。
在实验中,我们教师模型和学生模型都是使用3D nnU-Net。结果如表7所示。由于训练时标记的案例数量不同,下界子任务与上界子任务之间存在性能差距。当涉及到无标记数据时,性能在平均DSC和NSD上逐渐提高,说明该方法可以利用无标记的情况来提高多器官分割性能。
表7:半监督基准的定量多器官分割结果。
图9:来自半监督分割基准测试集的挑战性示例。
图9展示了每个子任务的3个挑战性例子的分割结果。可以观察到,我们的半监督方法能够通过利用未标记的数据来减少误分类。第一行和第三行分别显示有大的肾脏肿瘤和肝内胆管扩张的病例。病理改变对肾脏分割构成了极大的挑战。第二行演示了脾脏与肝脏具有相似的外观,当训练数据有限时,它往往被识别为肝脏。我们还可以发现分割误差是可以逐渐修正的利用更多未标记的数据。从Supplementary Figure 3中分割结果的violin图可以看出,当未标记数据的数量增加时,四个器官的表现都有增加的趋势。
5.3弱监督腹部器官分割基准
这个基准测试是为了探索如何使用弱注释来生成完整的分割结果。对于分割任务,有几种不同的弱注释策略,如随机涂鸦、边界框、极端点和稀疏标签。稀疏标签是放射科医师在手工描绘器官时最常用的器官分割弱标注。在这个基准测试中,我们在训练集中提供了 slice-level的稀疏标签,其中只有部分切片(30%)得到了良好的标注。
5.3.1任务设置
训练集和测试集的选择动机: 我们选择Spleen Plus (41)作为训练集,因为它的训练案例最少。与使用其他数据集(如KiTS Plus (210), LiTS Plus (131))相比,这种选择更符合实际情况,因为在许多医疗中心,训练集只有有限的充分注释的病例。
弱监督器官分割基准包含三个子任务,如表8所示,其中只有一小部分切片以大致均匀的间隔进行标注。我们以大致均匀的间隔生成稀疏标签,因为在实践中,human-raters通常对稀疏标签进行注释,然后插入未标记的切片。具体来说,我们设置了5%、15%和30%三种不同的标注率,与现有的脑组织分割工作相似。我们使用5.3.2节的基线模型来推断所有剩余的案例,并选择100个案例作为最终的测试集,其中包括50个平均DSC和NSD得分最低具有挑战性的案例,以及50个随机选取的案例。
表8:弱监督腹部器官分割的任务设置和定量基线结果。
5.3.2基线和结果
我们的基线方法建立在2D nnUNet和完全连接条件随机场(CRF)的组合之上,这是由[100]中提出的方法驱动的,其中解决了缺失注释的挑战。[100]的主要思想是用有限的标记图像训练一个pixel-wise的分类(分割)网络,然后对未标记的图像进行分割,得到初始分割结果,然后使用全连通的CRF进行改善步骤。全连接CRF在许多分割任务中得到了广泛的应用(如肝脏和肝脏肿瘤分割、脑肿瘤分割),是一种细化分割结果的有效方法。我们的新基线也遵循这个想法,并有以下三个主要步骤。
- 步骤1。利用稀疏标签训练2D U-Net
- 步骤2。通过推理测试用例得到分割概率图;
- 步骤3。使用全连通的CRF来细化分割结果,其中一元势为概率图,成对势为CT衰减评分定义的三个高斯核势。
表8为四个器官的平均DSC和NSD评分,表9为每个器官的详细分割结果。正如预期的那样,训练案例的标注率越高,基线方法的分割性能越好。只有15%的注释,基线可以达到平均DSC评分超过90%的肝脏分割。这些结果可以激励我们采用基于深度学习的策略来减少人工注释的工作量和时间。在Supplementary Figure 4中,我们展示了不同标注比率下分割结果的violin图。15%到30%的标注比率带来的性能增益小于5%到15%的性能增益,这表明简单地添加注释并不总是带来线性性能改进。
表9:定量多器官分割结果在弱监督基准。
此外,可以发现使用CRF并没有带来显著的性能改善。在2018年著名的脑肿瘤分割(BraTS)挑战中,获胜的解决方案也发现了类似的现象。虽然结果不如预期,但它们为传统的基于能量的细分方法提供了新的机遇和挑战。具体来说,给定最初(不准确)的CNN分割结果,如何或什么样的能量模型可以持续提高分割精度?所有相关的结果,包括训练的模型、使用的CRF代码和超参数设置、分割结果及其概率图,将在未来的这一方向的研究中公开。
5.4连续腹部器官分割基准
持续学习是一个新兴的研究课题,受到了广泛的关注。目的是探索我们应该如何扩大训练的分割模型来学习新的任务而不忘记所学的任务。这类任务有几个术语,例如:continual learning, incremental learning, life-long learning or online learning. 。在本文中,我们使用持续学习来表示这类任务,这在现有文献中得到了广泛的应用。据我们所知,在CVPR 2020中,建立了第一个用于图像分类的持续学习基准。然而,医学图像分割仍然缺乏一个公开的持续学习基准。因此,我们建立了腹部器官分割的持续学习基准,并开发了基线解决方案。
5.4.1任务设置
训练集和测试集的选择动机: 使用原始单器官数据集KiTS(210)、脾脏(41)和MSD胰腺(281)作为训练集。为了评估这些方法的泛化能力,我们选择MSD Pancreas Ts (139) 作为只有肝脏注释的训练集,而不是LiTS(131)数据集,因为LiTS(131)是一个多中心数据集,更适合作为测试集。我们使用5.4.2节中的基线模型来推断所有剩余的案例,并选择100个案例作为最终的测试集,其中包括平均DSC和NSD得分最低的50个具有挑战性的案例,以及50个随机选取的案例。如表10所示,训练集包含4个数据集,每个数据集中只标注了一个器官。其中,MSD Pancreas Ts (139), KiTS (210), Spleen (41), and MSD Pancreas Ts (139) 的标签分别为肝、肾、脾和胰腺。总之,本任务需要建立一个单器官标注训练集的多器官分割模型。还应该注意的是,切换到新任务时,不能访问以前任务的数据集。例如,如果使用KiTS(210)数据集构建了肾脏分割模型,那么在使用 Spleen (41)数据集对模型进行扩增时,这个数据集将不可用。
表10:持续学习的任务设置和定量基线结果。
5.4.2基线和结果
在众所周知的学习不遗忘的激励下,我们开发了一种embarrassingly简单但有效的持续学习方法作为基线,包括以下四个步骤:
- 步骤1。基于 MSD Pancreas Ts(139)数据集单独训练肝脏分割 nnU-Net模型。
- 步骤2。利用训练后的肝脏分割模型推断KiTS(210),得到伪肝脏标签。因此,KiTS(210)中的每个病例都有肝脏和肾脏的标记。然后,我们使用新的标签训练一个能同时分割肝脏和肾脏的nnUNet模型。
- 步骤3。使用step2训练的模型推断MSD Pancreas(41),得到肝脏和肾脏的伪标签。因此, Spleen (210)中每个病例都有肝、肾和脾的标记。然后,我们使用新的标签训练一个nnU-Net模型,可以分割三个器官。
- 步骤4。使用step3训练的模型推断MSD Pancreas (281) ,得到肝、肾、脾伪标签。因此,在MSD Pancreas (281) 中,每个病例都有肝脏、肾脏、脾脏和胰腺的标签。最后,利用新标签训练神经网络,得到最终的多器官分割模型。
表10为四个器官的平均DSC和NSD评分,表11为每个器官的详细结果。综上所述,在全监督的分割结果中,单器官数据集的学习性能低于完整标注的学习(表4、表5),说明模型在切换到新任务时仍然容易忘记之前的部分任务。每个器官的分割表现的violin图如图5所示。肝脏分割的DSC和NSD评分最好,分布紧凑,离群值少,而胰腺分割的评分较低。NSD得分低且分布分散,由于各种病理变化的影响,其边界误差相对较高,如图10所示。
表11:持续学习多器官定量分割结果。
图10:连续学习多器官分割基准测试中具有挑战性的例子。
5.5常用测试集的评价与比较
上述测试集在四个基准测试中是不同的。为了在基准之间进行全面的比较,我们引入了一个通用的测试集(如3.1节所述的NJU数据集),其中包含50例腹部CT病例.
表12给出了四个基准的共同测试集的定量结果。观察到,全监督方法在肾、脾、胰腺分割中获得了最好的平均DSC和NSD评分,因为它使用了许多标记病例。然而,由于注释的负担,在临床实践中往往难以获得所需的注释量。因此,问题就在于什么是需要的高效注释方法。子任务2中的semi-supervised方法只有41个标记病例和800对肝脏无标号的情况下达到最佳的性能和整体性能非常接近最好的全面监督方法标签(361例),这表明使用大型和多样化的未标记的情况下可以显著提高性能。弱监督方法的性能最低,但它需要的注释负担最少。
表12:四个基准的共同测试集的定量结果。
6.结论
在这项工作中,我们引入了 AbdomenCT-1K,它包括 multi-center, multi-phase, multi-vendor, and multi-disease cases。虽然SOTA方法在肝脏、肾脏和脾脏分割等几个已有的基准测试中取得了前所未有的性能,但我们的大规模研究显示,如第4节所示,一些问题仍未解决。特别是当评价指标为DSC,测试集的数据分布与训练集相似,且测试集中没有未见疾病的疑难病例时,SOTA方法可以获得较好的分割结果。然而,对于许多具有挑战性的病例,如从不同的扫描仪或临床中心获得的新new CT phases、严重疾病的病例,SOTA方法无法泛化其在未见数据集上的巨大性能。
为了解决未解决的问题,我们建立了四个新的腹部器官分割基准,包括全监督、半监督、弱监督和持续学习。与现有流行的全监督腹部器官分割基准(如LiTS、MSD和KiTS)不同,我们的新基准有三个主要特点:
- 每个基准中的测试用例来自多个不同的CT扫描仪和医疗中心。
- 选择具有挑战性的病例(如未见或罕见疾病),并纳入我们的测试集,如巨大肿瘤病例。
- 我们不仅关注基于区域的度量(DSC),还强调与边界相关的度量(NSD),因为边界误差在许多腹部器官手术(如肿瘤切除和器官移植)的术前计划中是至关重要的
主要的限制是我们只关注四个大的腹部器官的分割。然而,在我们的数据集中还存在更困难的器官和没有注释的病变。为了解决这一问题,我们对50例有8个额外器官的病例进行了注释,包括食道、胆囊、胃、主动脉、腹腔干、下腔静脉、右肾上腺和左肾上腺。对于病变,原始数据集中没有详细的病理信息。单纯的CT扫描很难做出明确准确的诊断,因为鉴别(恶性)肿瘤通常需要病理检查。作为替代,我们包含了原始的单器官肿瘤掩膜,并通过标注所有其他可能的肿瘤,提供了663例的伪肿瘤标记,可用于噪声标记学习。
基于深度学习的分割方法已经取得了巨大的成功。我们希望我们的庞大和多样化的数据集和开箱即用的基线方法有助于推动腹部器官分割走向真正的临床实践。