高斯混合模型(GMM)
高斯混合模型
k-means 聚类模型非常简单并且易于理解,但是它的简单性也为实际应用带
来了挑战。特别是在实际应用中,k-means 的非概率性和它仅根据到簇中心点的距离来指
派簇的特点将导致性能低下。这一节将介绍高斯混合模型,该模型可以被看作是k-means
思想的一个扩展,但它也是一种非常强大的聚类评估工具。还是从标准导入开始:
%matplotlib inline
import matplotlib.pyplot as plt
import seaborn as sns; sns.set()
import numpy as np
高斯混合模型(GMM)为什么会出现:k-means
算法的缺陷
下面来介绍一些k-means 算法的不足之处,并思考如何改进我们的聚类模型。就像前一节
所看到的,只要给定简单且分离性非常好的数据,k-means 就可以找到合适的聚类结果。
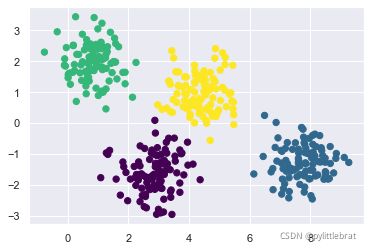
例如,只要有简单的数据簇,k-means 算法就可以快速给这些簇作标记,标记结果和通过
肉眼观察到的簇的结果十分接近:
from sklearn.datasets import make_blobs
X, y_true = make_blobs(n_samples=400, centers=4,
cluster_std=0.60, random_state=0)
X = X[:, ::-1] # 交换列是为了方便画图
#用k-means标签画出数据
from sklearn.cluster import KMeans
kmeans = KMeans(4, random_state=0)
labels = kmeans.fit(X).predict(X)
plt.scatter(X[:, 0], X[:, 1], c=labels, s=40, cmap='viridis');
簇似乎有一小块区域重合,因此我们对重合部分的点将被分配到哪个簇不是很有信心。不
幸的是,k-means 模型本身也没有度量簇的分配概率或不确定性的方法(虽然可以用数据
重抽样方法bootstrap 来估计不确定性)。因此,我们必须找到一个更通用的模型。
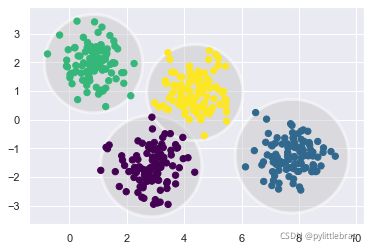
理解k-means 模型的一种方法是,它在每个簇的中心放置了一个圆圈(在更高维空间中是
一个超空间),圆圈半径根据最远的点与簇中心点的距离算出。这个半径作为训练集分配
簇的硬切断(hard cutoff),即在这个圆圈之外的任何点都不是该簇的成员。可以用以下函
数将这个聚类模型可视化:
from sklearn.cluster import KMeans
from scipy.spatial.distance import cdist
def plot_kmeans(kmeans, X, n_clusters=4, rseed=0, ax=None):
labels = kmeans.fit_predict(X)
# 画出输入数据
ax = ax or plt.gca()
ax.axis('equal')
ax.scatter(X[:, 0], X[:, 1], c=labels, s=40, cmap='viridis', zorder=2)
# 画出k-means模型的表示
centers = kmeans.cluster_centers_
radii = [cdist(X[labels == i], [center]).max() for i, center in enumerate(centers)]
for c, r in zip(centers, radii):
ax.add_patch(plt.Circle(c, r, fc='#CCCCCC', lw=3, alpha=0.5, zorder=1))
kmeans = KMeans(n_clusters=4, random_state=0)
plot_kmeans(kmeans, X)
k-means 有一个重要特征,它要求这些簇的模型必须是圆形:k-means 算法没有内置的方法
来实现椭圆形的簇。因此,如果对同样的数据进行一些转换,簇的分配就会变得混乱:
rng = np.random.RandomState(13)
X_stretched = np.dot(X, rng.randn(2, 2))
kmeans = KMeans(n_clusters=4, random_state=0)
plot_kmeans(kmeans, X_stretched)
通过肉眼观察,可以发现这些变形的簇并不是圆形的,因此圆形的簇拟合效果非常糟糕。
总之,k-means 对这个问题有点无能为力,只能强行将数据拟合至4 个圆形的簇,但却导
致多个圆形的簇混在一起、相互重叠,右下部分尤其明显。有人可能会想用PCA(详情请
参见5.9 节)先预处理数据,从而解决这个特殊的问题。但实际上,PCA 也不能保证这样
的全局操作不会导致单个数据被圆形化。
k-means 的这两个缺点——类的形状缺少灵活性、缺少簇分配的概率——使得它对许多数
据集(特别是低维数据集)的拟合效果不尽如人意。
你可能想通过对k-means 模型进行一般化处理来弥补这些不足,例如可以通过比较每个点
与所有簇中心点的距离来度量簇分配的不确定性,而不仅仅是关注最近的簇。你也可能想
通过将簇的边界由圆形放宽至椭圆形,从而得到非圆形的簇。实际上,这正是另一种的聚
类模型——高斯混合模型——的两个基本组成部分。
一般化E-M:高斯混合模型
一个高斯混合模型(Gaussian mixture model,GMM)试图找到多维高斯概率分布的混合
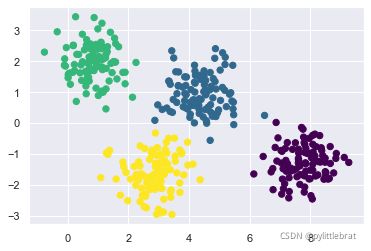
体,从而获得任意数据集最好的模型。在最简单的场景中,GMM 可以用与k-means 相同
的方式寻找类:
from sklearn.mixture import GaussianMixture as GMM
gmm = GMM(n_components=4).fit(X)
labels = gmm.predict(X)
plt.scatter(X[:, 0], X[:, 1], c=labels, s=40, cmap='viridis');
但由于GMM 有一个隐含的概率模型,因此它也可能找到簇分配的概率结果——在Scikit-
Learn 中用predict_proba 方法实现。这个方法返回一个大小为[n_samples, n_clusters]
的矩阵,矩阵会给出任意点属于某个簇的概率:
probs = gmm.predict_proba(X)
print(probs[:5].round(3))
[[0.469 0.531 0. 0. ]
[0. 0. 0. 1. ]
[0. 0. 0. 1. ]
[0. 1. 0. 0. ]
[0. 0. 0. 1. ]]
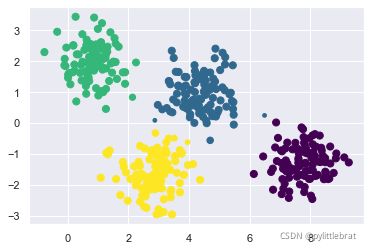
我们可以将这个不确定性可视化,用每个点的大小体现预测的不确定性,使其成正比.在簇边界上的点反映了簇分配的不确定性:
size = 50 * probs.max(1) ** 2 # 平方强调差异
plt.scatter(X[:, 0], X[:, 1], c=labels, cmap='viridis', s=size);
高斯混合模型本质上和k-means 模型非常类似,它们都使用了期望最大化方法,具体实现
如下。
- 选择初始簇的中心位置和形状。
- 重复直至收敛。
- 期望步骤(E-step):为每个点找到对应每个簇的概率作为权重。
- 最大化步骤(M-step):更新每个簇的位置,将其标准化,并且基于所有数据点的权重来确定形状。
最终结果表明,每个簇的结果并不与硬边缘的空间(hard-edged sphere)有关,而是通过高
斯平滑模型实现。正如在k-means 中的期望最大化方法,这个算法有时并不是全局最优解,
因此在实际应用需要使用多个随机初始解。
下面创建一个可视化GMM 簇位置和形状的函数,该函数用gmm 的输出结果画出椭圆:
from matplotlib.patches import Ellipse
def draw_ellipse(position, covariance, ax=None, **kwargs):
"""Draw an ellipse with a given position and covariance"""
ax = ax or plt.gca()
# Convert covariance to principal axes
if covariance.shape == (2, 2):
U, s, Vt = np.linalg.svd(covariance)
angle = np.degrees(np.arctan2(U[1, 0], U[0, 0]))
width, height = 2 * np.sqrt(s)
else:
angle = 0
width, height = 2 * np.sqrt(covariance)
# Draw the Ellipse
for nsig in range(1, 4):
ax.add_patch(Ellipse(position, nsig * width, nsig * height,
angle, **kwargs))
def plot_gmm(gmm, X, label=True, ax=None):
ax = ax or plt.gca()
labels = gmm.fit(X).predict(X)
if label:
ax.scatter(X[:, 0], X[:, 1], c=labels, s=40, cmap='viridis', zorder=2)
else:
ax.scatter(X[:, 0], X[:, 1], s=40, zorder=2)
ax.axis('equal')
w_factor = 0.2 / gmm.weights_.max()
for pos, covar, w in zip(gmm.means_, gmm.covars_, gmm.weights_):
draw_ellipse(pos, covar, alpha=w * w_factor)
gmm = GMM(n_components=4, random_state=42)
# plot_gmm(gmm, X)