论文小结——基于聚类演化随机森林的阿尔兹海默症的多模态数据分析
系列文章目录
基于多模态成像遗传学数据来预测帕金森病相关基因和大脑区域的新型GERNNE方法
基于聚类演化随机森林的阿尔兹海默症的多模态数据分析
- 系列文章目录
- 前言
- 一、全局总览
- 二、方法与流程
-
- 1.流程
- 2.数据预处理
- 3.融合特征构建
- 4.构建聚类演化随机森林(CERF)
- 5.聚类演化随机森林(CERF)学习过程
-
- 均值分类
- 参数优化
- 三、实验结果
-
- 1.融合特征构造结果
- 2.各最优参数
-
- 最优决策树数量和聚类演化次数
- 最佳融合特征提取
- 3.异常脑区和基因提取
- 四、总结与展望
前言
这篇博客只是我总结自己对这篇论文的理解,还存在许多理解不到位和理解错误的地方,还请大家批评和指正。
这篇论文题目为:
Multimodal Data Analysis of Alzheimer’s Disease Based on Clustering Evolutionary Random Forest
X. -a. Bi, X. Hu, H. Wu and Y. Wang, “Multimodal Data Analysis of Alzheimer’s Disease Based on Clustering Evolutionary Random Forest,” in IEEE Journal of Biomedical and Health Informatics, vol. 24, no. 10, pp. 2973-2983, Oct. 2020, doi: 10.1109/JBHI.2020.2973324.
提示:以下是本篇文章正文内容,下面案例可供参考
一、全局总览
本篇论文作者
面临的两个问题与挑战:
1. 从小样本的高维信息提取重要信息
2. 缺乏全面的框架
解决与应对方法:
1. 聚类演化随机森林(CERF)
2. AD(阿尔兹海默症)多模态数据综合分析框架
创新点:
- 聚类演化随机森林方法
成果:
1. 发现脑异常区
2. 检测致病基因
局限性:
1.多模态数据融合还有进展空间
2.如何设计合适的指标来捕捉fMRI(功能磁共振)与基因数据之间的相关性
二、方法与流程
1.流程
- 多模态数据预处理
- 相关分析方法
- 融合特征矩阵
- 聚类演化随机森林
- 样本分类
- 致病基因的提取
2.数据预处理
这些数据是从ADNI数据库中获取的,ADNI数据库是阿尔茨海默病的一个大型公共数据库(链接:ADNI)。大量PET数据,MRI数据和AD患者的基因数据都可以在这里获得。本研究以37例AD患者和35例HC为实验样本,每个样本均有静态的fMRI和SNP数据。
数据预处理方面不是我学习的方向,所以我不能参上我的理解。
用DPARSF对fMRI数据进行标准预处理,如切片定时校正和头部运动调整等。FMRI数据预处理的具体步骤如下:
1. 将原始数据转换为Nifti格式文件。
2. 删除所有样本的前10个时间卷,以确保扫描仪的磁梯度场稳定。
3. 对剩余卷执行片定时校正。
4. 调整头部运动以保证每个样本的大脑处于相同的位置。
5. 使用EPI模板对图像进行规范化。
6. 用高斯核平滑图像的噪声,半高宽为6mm。
7. 利用线性模型消除了物体运动、白质和全局信号等协变量信号干扰。
8. 用0.01Hz到0.08Hz的频率范围对功能时间序列进行滤波。
9. 通过Ilumina Omni 2.5m微芯片获得了样品的SNP信息。
10. Plink用于对SNP数据执行预处理,具体步骤如下:
将样本呼叫率阈值设为95%,以评价基因数据的总体质量。
将基因分型阈值、最小等位基因频率和Hardy-Weinberg平衡检验分别设置为99.9%、4%和1E-4,以消除质量较差的SNPs。
3.融合特征构建
实现的目的:检测基因与脑区之间的相关性
- 首先采用AAL(自动解剖标记模板),将fMRI数据分割成90个脑区,得到脑区时间序列。
- SNP基因根据相应基因分组,Msnp基因数大于阈值len都保留
- 将SNP中的四种碱基(A、T、C和G)转换成不同的数字(如1、2、3和4),然后通过转换的方法得到基因的数字序列。
- 把基因序列和大脑时间序列的长度调整为2len并分别采用Pearson相关分析、典型相关分析(CCA)和距离相关分析(DCA)作为构建脑区基因对的候选方法。
4.构建聚类演化随机森林(CERF)
CERF的功能:应对小样本问题,处理高维融合特征。
内容:将聚类进化和随机森林相结合,实现了自适应集成学习。通过对随机森林中决策树的分层聚类,从高维特征中逐步筛选出HC和AD之间最容易识别的特征。

引入聚类演化的思想,通过重复聚类演化,提高分类器的多样性和准确性。
假设样本集是S={x,y}Nn=1。然后,训练集A={xAa,yAa}nAa=1,验证集B={xBb,yBb}nBb=1和测试集C={xCc,yCc}nCc=1按5:3:2的比例从样本中随机选取。xAa={BG1a,BG2a,… BGma}和yAa={−1,+1},xAa表示训练集合A中的一个样本,BGma指示m-样本的特征,yAa指示示例的相应类标签“+1”为HC,“−1”为AD。
总结一句话,将总样本按照5:3:2的比例随机分为训练集A,验证集B,测试集C。+1为正常,-1为阿尔兹海默症。
xAa表示训练集合A中的一个样本,BGma指示m-样本的特征,
由于本文没有给出代码,所以我觉的自己能力不够,复现不出来。下面的公式只能理解大概,没有代码,确实不能理解的很透彻。主要还是学习整体的框架流程:
- G=fix(√m)
特征G和正常样本都是从训练集中随机抽选的
G特征数量
m集合样本总数
fix(x)四舍五入函数
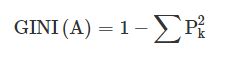
利用GINI函数找到不同特征的最优分类点,并构造一个决策树。
P_K是分类结果为K的概率。

BGm 表示m-所有样本的特征
这里算的是当特征值BGm是j时候的基尼指数
N 是A组的样本总量,n1和n2分别是样本子集A1和A2中的样本数
-
然后计算所有特征值的基尼系数。BGm,选择与最小基尼系数对应的值作为最优分类点。还计算了所有特征的最优二进制分类点。2, 3公式构造了一个决策树。
-
上述决策树的构造重复了P次。得到P决策树,并组装成初始随机森林。
-
文章这里使用不一致度量、关联度量和kappa相似性度量来检测相似性。
-
不一致度量

计算不一致度量,我认为差异度更好听一些。
这里
T_ij:训练集上dti和dtj正确分类的样本数量
Ri_j: 训练集上只有dti正确分类的样本数量
Rj_i: 训练集上只有dtj正确分类的样本数量
F_ij:训练集上,dti和dtj分类都不正确的样本数量
DM_ij越小,代表两个分类器相似性越高。
5.聚类演化随机森林(CERF)学习过程
输入:实验数据集{X,Y}
输出:聚类进化随机森林
初始化{X,Y},D,E,I.
{X,Y}是实验数据集,
D是初始决策树的数目。
E是聚类进化的步长。
I是集群进化的时代。
数据集分为训练集,验证集,测试集
I=1
为K=1至D:
选择{{rm{x,Y}}_{rm{tra_k}
随机选择一个特征子集训练
随机选择样本
决策树分类精度的
→检验k
最终达到
随机森林=决策树集合
从公式(7)–(9)
决策树间相似性的计算
随机林中的决策树聚类
保持决策树在每个簇中的最高精度→删除低效树
D=n−ie,i=i+1
{随机森林}新的=保留决策树的集合
直到{随机森林}的精度新的达到顶峰
均值分类
即通过得到的随机森林集合里所有的分类器一起分类,综合所有分类结果,等权重,如果超过一般认为是正样本就预测为正。
参数优化
- 决策树的数目和聚类演化的次数
在[a,b],[c,d]之间,通过训练找到最优值
[300,500]和[1,25]
决策树的数目初始值300,
聚类演化的次数初始值1。
三、实验结果
1.融合特征构造结果
AAL模板分成90个脑区
对每个脑区提取平均时间序列
同时SNP提取序列长度30以上的36个基因,并截取序列前60碱基对
然后通过三种相关分析方法就算相关系数,其中pearson方法性能最好。
2.各最优参数
最优决策树数量和聚类演化次数

最优进化次数与初始决策树数量的关系曲线
最终随机森林的分类准确率(决策树初始数为340,聚类进化次数为7)接近90%。
最佳融合特征提取
提取最终随机林中每个决策树选择的所有“脑区-基因对”,并计算每个“脑区-基因对”的频率。频率越高,HC与AD的差异越大。因此,频率最高的400对“脑区-基因对”被认为是“重要的脑区-基因对”,其频率大于12的特征列于下表。
用前290个“重要脑区-基因对”构建子集时,随机林的分类准确率最高达91.3%。因此,前290对“脑区-基因对”是“最佳脑区-基因对”。

基于不同子集的传统随机森林的精确性。
3.异常脑区和基因提取

主要的异常脑区。不同脑区的权重显示在图9(A)。不同脑区在大脑中的位置显示在图9(B)。脑区结节大小越大,AD与脑区的相关性越明显。

主要致病基因的频率。基因频率越高,AD与AD的相关性越显著。
四、总结与展望
总的来说和我上次读的论文大致没啥区别。除了用决策树代替ANN,研究的PD变成了AD,其他基本不变。但是这篇论文,里面存在一些不知道什么语言的类似这种
begin{equation*} {\rm{FB}}{{\rm{G}}{\rm{j}}} = \sum\limits{{\rm{i}} = 1}^{\rm{m}} {{\rm{FB}}{{\rm{G}}_{{\rm{i}},
看的我很难受,不好理解。其他方面我理解的还可以。
如果想看好这篇论文,可以看我之前的博客。
可学习性一般,对我这种新手没有太大学习价值。
首先没代码,就没法复现。
其次,在没代码的基础上,就没办法吃透。
最终,只能学到一种方法思路,框架。但是现阶段还难以用上。
最好还是学有代码的论文。




