基于U-Net的递归残差卷积神经网络在医学图像分割中的应用
转载:
版权声明:本文为博主原创文章,遵循 CC 4.0 BY-SA 版权协议,转载请附上原文出处链接和本声明。
本文链接:https://blog.csdn.net/weixin_45723705/article/details/107097488
英文原文链接: Recurrent Residual Convolutional Neural Network based on U-Net (R2U-Net) for Medical Image Segmentation
摘要
基于深度学习(DL)的语义分割方法在过去的几年里已经提供了最先进的性能。更具体地说,这些技术已经成功地应用到医学图像的分类、分割和检测任务中。一种深度学习技术—U-Net,已经成为这些应用中最流行的技术之一。本文提出了一种基于U-Net的递归卷积神经网络(RCNN)和基于U-Net模型的递归残差卷积神经网络(RRCNN),分别命名为RU-Net和R2U-Net网。该模型利用了U网络、残差网络和RCNN的优点。对于分割任务,这些提出的体系结构有几个优点。首先,在训练深层架构时,剩余单元会有所帮助。第二,利用递归残差卷积层进行特征积累,保证了分割任务更好的特征表示。第三,它允许我们在相同网络参数的情况下,设计出更好的U-Net结构,以更好地进行医学图像分割。在视网膜血管分割、皮肤癌分割和肺部病变分割等三个基准数据集上对所提出的模型进行了测试。实验结果表明,与包括UNet和残差U-Net(ResU-Net)等等效模型相比,该算法在分割任务方面具有更好的性能。
索引项—医学成像,语义分割,卷积神经网络,U-Net,残差U-Net,RU-Net,R2U-Net。
I.简介
目前,DL为图像分类[1]、分割[2]、检测和跟踪[3]以及字幕[4]提供了最先进的性能。自2012年以来,人们提出了一些深卷积神经网络(DCNN)模型,如AlexNet[1]、VGG[5]、GoogleNet[6]、Remailture Net[7]、DenseNet[8]和CapsuleNet[9][65]。基于DL的方法(尤其是CNN)为分类和分割任务提供了最先进的性能,原因有以下几个:首先,**函数解决了DL方法中的训练问题。其次,dropout有助于使网络正规化。第三,有几种有效的优化技术可以用来训练CNN模型[1]。然而,在大多数情况下,任务和任务的分类都是用单标度数据集来评估的。另外,小的体系结构上的变体模型用于语义图像分割任务。例如,全连接卷积神经网络(FCN)也为计算机视觉中的图像分割任务提供了最新的结果[2]。FCN的另一个变体也被提出,称为SegNet[10]。
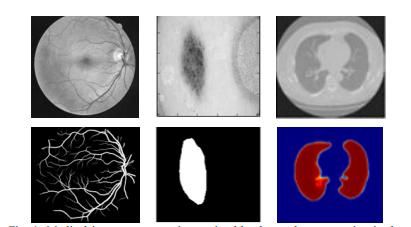
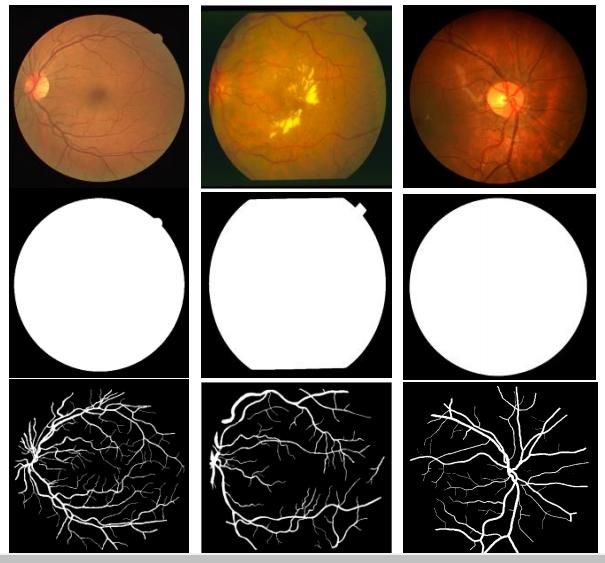
图1:医学图像分割:左侧为视网膜血管分割,中间为皮肤癌病灶分割,右侧为肺部分割。
由于DCNNs在计算机视觉领域的巨大成功,该方法的不同变体被应用于不同的医学成像模式,包括分割、分类、检测、配准和医学信息处理。医学成像来自不同的成像技术,如计算机断层扫描(CT)、超声波、X射线和磁共振成像(MRI)。计算机辅助诊断(CAD)的目标是获得更快、更好的诊断结果,以保证对大量患者进行更好的治疗。此外,无需人工参与的高效自动处理可减少人为错误,也可减少总体时间和成本。由于人工分割方法的缓慢和繁琐,人们对计算机算法的需求很大,这种算法能够在不需要人工干预的情况下快速准确地进行分割。然而,医学图像分割存在数据匮乏、类不平衡等局限性。在大多数情况下,用于培训的大量标签(通常为数千个)不可用,原因如下[11]。对数据集进行标记需要这一领域的专家,这是昂贵的,而且需要大量的努力和时间。有时,会应用不同的数据转换或增强技术(数据加噪、旋转、平移和缩放)来增加可用标记样本的数量[12、13和14]。另外,基于图像块的方法被用来解决类不平衡问题。在这项工作中,我们评估了所提出的方法,包括基于图像块的方法和基于整个图像的方法。然而,要从基于图像块的方法切换到处理整个图像的基于像素的方法,我们必须意识到类不平衡问题。在语义分割的情况下,图像背景被分配一个标签,前景区域被分配一个目标类。因此,分类不平衡问题就迎刃而解了。文献[13,14]中引入了交叉熵损失和骰子相似性两种先进的分类和分割任务训练方法。
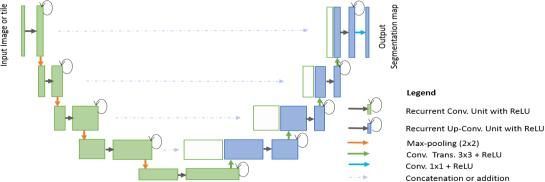
图2.U-Net体系结构由卷积编码和解码单元组成,以图像为输入,生成相应像素类的分割特征映射。
此外,在医学图像处理中,全局定位和上下文调制经常用于定位任务。在识别任务中,每个像素被分配一个类别标签,该标签具有与目标病灶轮廓相关的期望边界。为了确定这些目标病灶的边界,我们必须强调相关像素点。医学成像中的目标检测[15,16]就是一个例子。在DL革命之前,有几种传统的机器学习和图像处理技术可用于医学图像分割任务,包括基于直方图特征的幅度分割[17]、基于区域的分割方法[18]和图切割方法[19]。然而,近年来,在医学图像分割、病变检测和定位领域,利用DL的语义分割方法变得非常流行[20]。此外,基于DL的方法被称为通用学习方法,其中单个模型可以有效地用于不同的医学成像模式,如MRI、CT和X射线。
根据最近的一项调查,DL方法应用于几乎所有的医学成像模式[20,21]。此外,在不同医学影像学模式下,发表的关于分割任务的论文数量最多[20,21]。文献[22]提出了一种基于DCNN的脑肿瘤分割与检测方法。从体系结构的角度来看,用于分类任务的CNN模型需要一个编码单元并提供类概率作为输出。
在分类任务中,我们使用**函数和子采样层进行卷积运算,从而降低特征映射的维数。当输入样本穿过网络的各个层时,特征映射的数量增加,但是特征映射的维数降低。这在图2中的模型的第一部分(绿色)中显示。由于特征映射的数量在深层增加,网络参数的数量也相应增加。最后,在网络的末端应用Softmax运算来计算目标类的概率。
与分类任务不同,分割任务的体系结构需要卷积编码和解码单元。该编码单元用于将输入图像编码成数量更多、维数更低的映射。解码单元用于执行上卷积(反卷积)操作,以生成与原始输入图像具有相同维数的分割图。因此,与分类任务的体系结构相比,分割任务的体系结构通常需要几乎两倍的网络参数。因此,设计高效的DCNN体系结构来实现任务的分割,以减少网络参数的数量,从而获得更好的性能。
本研究展示了两种改进的分割模型,一种使用递归卷积网络,另一种使用递归残差卷积网络。为了实现我们的目标,我们对所提出的模型在不同的医学想象模式上进行了评估,如图1所示。这项工作的贡献可概括如下:
图3.具有卷积编码和解码单元的RU-Net体系结构,使用基于递归卷积层(RCL)的U-Net体系结构。剩余单元与RCL一起用于R2U网络体系结构。
1) 介绍了两种新的医学图像分割模型RU-Net和R2U-Net。
2) 实验在视网膜血管分割、皮肤癌分割和肺部分割三种不同的医学影像模式上进行。
3) 对提出的模型进行了性能评价,分别对基于斑片的视网膜血管分割任务和端到端图像分割方法进行了皮肤病变和肺部的分割任务。
4) 与最近提出的最先进的方法进行比较,这些方法在具有相同网络参数的等效模型中显示出优越的性能。
论文的结构安排如下:第二节论述了相关工作。第三节介绍了所提出的RU-Net和R2U-Net模型的结构。第四节解释了数据集、实验和结果。第五节讨论了本文的结论和未来的发展方向。
II. 相关工作
语义分割是一个非常活跃的研究领域,它使用DCNNs对图像中的每个像素进行单独分类,这是由计算机视觉和医学成像领域中不同的具有挑战性的数据集推动的[23,24,25]。在深度学习革命之前,传统的机器学习方法主要依赖于手工设计的特征,这些特征用于独立地对像素进行分类。在过去的几年里,已经有很多模型被提出,这些模型已经证明了更深的网络更适合于识别和分割任务[5]。然而,由于梯度消失问题,训练非常深的模型是困难的,这是通过实现**函数,如校正线性单元(ReLU)或指数线性单元(ELU)[5,6]。这个问题的另一个解决方案是由何凯明等人提出的,一个深度残差模型克服了这个问题,利用自身映射来促进训练过程[26]。
此外,基于FCN的CNNs分割方法为自然图像分割提供了优越的性能[2]。其中一种基于图像图像块的体系结构称为随机体系结构,它的计算量非常大,包含约134.5百万的网络参数。这种方法的主要缺点是大量的像素重叠和多次执行相同的卷积。使用递归神经网络(RNN)改进了FCN的性能,该网络在非常大的数据集上进行了微调[27]。DeepLab的语义图像分割是最先进的执行方法之一[28]。SegNet由两部分组成,一部分是编码网络,即13层VGG16网络[5],对应的解码网络使用像素级分类层。本文的主要贡献是解码器对其低分辨率输入特征映射进行上采样的方法[10]。后来,2015年提出了一个改进版的SegNet,称为贝叶斯SegNet[29]。这些体系结构中的大多数都是使用计算机视觉应用程序进行探索的。然而,有一些针对医学图像分割的深度学习模型,因为它们考虑了数据不足和类不平衡问题。
最早也是最流行的语义医学图像分割方法之一被称为“U-Net”[12]。基本U-Net模型的示意图如图2所示。根据结构,网络主要由两部分组成:卷积编解码单元。基本的卷积运算是在网络的两个部分中执行ReLU**之后执行的。对于编码单元中的下采样,执行2×2最大池操作。在解码阶段,执行卷积转置(表示上卷积或反卷积)操作以对特征映射进行上采样。第一个版本的U-Net被用来裁剪和复制从编码单元到解码单元的特征映射。U-Net模型为分割任务提供了几个优点:首先,该模型允许同时使用全局位置和上下文。第二,它使用很少的训练样本,并且为分割任务提供了更好的性能[12]。第三,一个端到端的流水线在前向过程中处理整个图像并直接生成分割图。这确保了U-Net保留了输入图像的完整上下文,这是与基于图像块的分割方法相比的一个主要优势[12,14]。
然而,U-Net不仅局限于医学成像领域的应用,现在这种模型也大量应用于计算机视觉任务[30,31]。同时,已经提出了不同的U-Net模型变体,包括一个非常简单的U-Net变体,用于基于CNN的医学影像数据分割[32]。在这个模型中,对U-Net的原始设计做了两个修改:首先,从网络的一个部分到另一个部分,将多个分割图和前向特征映射的组合(按元素方向)求和。特征映射来自编码和解码单元的不同层,最后在编码和解码单元之外执行求和(元素级)。作者报告说,与U-Net相比,训练期间的性能有了很大的提高,收敛性更好,但是在测试阶段使用特征的总和时没有观察到任何好处[32]。然而,这个概念证明了特征求和对网络性能的影响。跳过连接对于生物医学图像分割任务的重要性已经用U-Net和残差网络进行了经验评估[33]。2016年提出了一种深度轮廓感知网络,称为深度轮廓感知网络(deep ContourAware Networks,DCAN),它可以使用层次结构提取多层次上下文特征,用于组织学图像的精确腺体分割,并显示出非常好的分割性能[34]。此外,Nabla网络:一种类似深挖掘的卷积结构在2017年被提出用于分割[35]。
其他基于U-Net的深度学习方法也被提出用于三维医学图像分割任务。用于体积分割的3D-Unet体系结构从稀疏标注的体积图像中学习[13]。提出了一种基于体图像的端到端三维医学图像分割系统,称为V-net,它由一个具有残差连接的FCN组成[14]。本文还介绍了骰子丢失层[14]。此外,文献[36]提出了一种三维深度监督的医学图像自动分割方法。High-Res3DNet在2016年被提议使用残差网络进行3D分割任务[37]。2017年,提出了一种基于CNN的脑肿瘤分割方法,该方法使用3D-CNN模型和完全连接的CRF[38]。2016年,在X-net中提出了残存体素的胰腺分割。这种架构利用了残差网络和来自不同层的特征图的总和[40]。
另外,本文提出了两种基于U-Net体系结构的语义分割模型。所提出的基于U网络的递归卷积神经网络模型被称为RU网络,如图3所示。此外,我们还提出了一个基于残差RCNN的U-Net模型,称为R2U-Net。以下部分提供了这两个模型的体系结构细节。
III. RU-NET和R2U-NET 网络结构
受深度残差模型[7]、RCNN[41]和UNet[12]的启发,我们提出了两种分割任务的模型RU-Net和R2U-Net。这两种方法利用了最近开发的三种深度学习模型的优点。RCNN及其变体已经在使用不同基准的对象识别任务上表现出了优异的性能[42,43]。根据文献[43]中改进的残差网络,递归残差卷积运算可以从数学上加以证明。循环卷积层(RCL)的操作是相对于根据RCNN表示的离散时间步长执行的[41]。让我们考虑残差RCNN(RRCNN)块的l层中的x输入样本和位于RCL中第k特征映射的输入样本中(i,j)处的像素。另外,假设O(l,ijk)网络的输出在时间步长t处。输出可以表示为:
这里(f(i,j),l))()和(r(i,j),l))(t-1)分别是标准卷积层和第l个RCL的输入。(f,k)和(r,k)值分别为标准卷积层的权重和kth要素图的RCL,(k)为偏差。RCL的输出被送入标准ReLU**函数,并表示为:
ℱ(,)表示RCNN单元第l层的输出。ℱ(,)的输出分别用于RU网模型的卷积编解码单元中的下采样层和上采样层。在R2U网络的情况下,RCNN单元的最终输出通过图4(d)所示的剩余单元。我们考虑RRCNN块的输出是+1,计算如下:
这里,表示RRCNN块的输入样本。+1样本被用作R2U-Net编解码卷积单元中的直接后续子采样或上采样层的输入。然而,用于剩余单元的特征映射的数量和特征映射的尺寸与图4(d)中所示的RRCNN块中的相同。
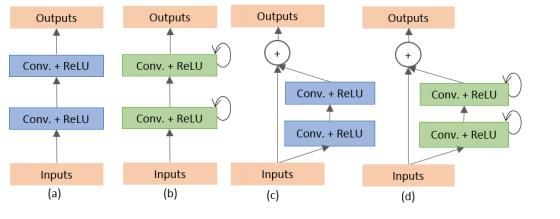
图4.卷积和递归卷积单元的不同变体(a)前卷积单元,(b)递归卷积块(c)残余卷积单元,以及(d)递归残余卷积单元(RRCU)。
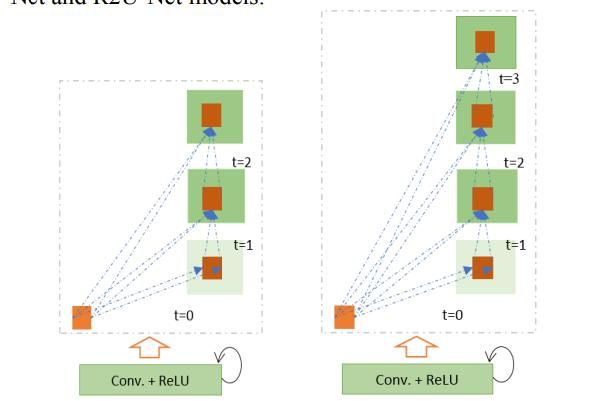
所提出的深度学习模型是图4(b)和(d)所示的堆叠卷积单元的构建块。本文中评估了四种不同的体系结构。首先,将具有前向卷积层和特征串联的U-Net应用于U-Net的原始版本[12]中的裁剪和复制方法。该模型的基本卷积单元如图4(a)所示。其次,使用具有残差连通性的前向卷积层的U-Net,它通常被称为残差U-Net(ResU-Net),如图4(c)[14]所示。第三种结构是具有前向递归卷积层的U-Net,如图4(b)所示,称为RU-Net。最后,最后一个架构是具有递归卷积层的U-Net,如图4(d)所示,它被称为R2U网。图5示出了展开的RCL层相对于时间步长的图示。这里t=2(0~2)是指递归卷积运算,它包括一个单独的卷积层和两个随后的递归卷积层。在这个实现中,我们对RU-Net和R2U-Net模型的特征映射应用了从编码单元到解码单元的级联。
图5.t=2(左)和t=3(右)的展开循环卷积单元。
与U-Net模型相比,所提出的模型之间的差异有三个方面。该结构由与U-Net相同的卷积编解码单元组成。然而,在编码和解码单元中,RCLs和具有残差单元的RCLs来代替常规的前向卷积层。具有残差单元的RCLs有助于开发更有效的深层模型。其次,在两个模型的RCL单元中都包含了有效的特征积累方法。基于CNN的医学图像分割方法显示了从网络的一部分到另一部分的特征积累的有效性。在这个模型中,元素级特征求和是在U-Net模型之外进行的[32]。该模型只在训练过程中以更好的收敛性来体现效益。然而,由于模型中的特征积累,我们提出的模型显示了训练和测试阶段的好处。针对不同时间步长的特征积累保证了更好、更强的特征表示。因此,它有助于提取非常低层次的特征,这些特征对于不同的医学成像模式(如血管分割)的分割任务至关重要。第三,我们从基本的U-Net模型中删除了裁剪和复制单元,只使用连接操作,从而形成了一个更加复杂的体系结构,从而获得了更好的性能。
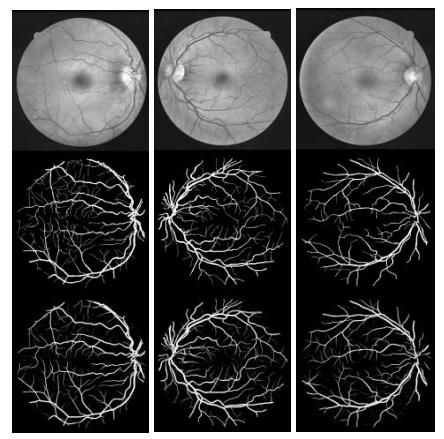
图6.来自训练数据集的示例图像:DRIVE数据集的左列、STARE数据集的中间列和CHASE-DB1数据集的右列。第一行显示原始图像,第二行显示视野(FOV),第三行显示目标输出。
与U-Net相比,使用所提出的体系结构有几个优点。首先是网络参数数量方面的效率。与U-Net相比,RU-Net和R2U-Net的网络参数数目相同,RU-Net和R2U-Net在分割任务上表现出更好的性能。重复操作和残差操作不会增加网络参数的数量。然而,它们确实对培训和测试性能有着显著的影响。以下章节[43]中的一组实验证明了这一点。这种方法也可以推广,因为它可以很容易地应用于基于Seg-Net[10]、3DU-Net[13]和V-Net14]的深度学习模型,提高了分割任务的性能。
IV. 实验步骤与结果
为了证明RU-Net和R2U-Net模型的性能,我们在三个不同的医学影像数据集上对它们进行了测试。这些包括从视网膜图像中分割血管(如图6所示的DRIVE、STARE和CHASE_DB1)、皮肤癌病灶分割和2D图像中的肺分割。对于这个实现,Keras和TensorFlow框架在一台拥有56G RAM和NIVIDIA GEFORCE GTX-980 Ti的GPU机器上进行实验。
A、 数据库摘要
1)血管分割
我们在三种不同的视网膜血管分割数据集上进行了实验,包括DRIVE、STARE和CHASH_DB1。驱动数据集由40幅彩色视网膜图像组成,其中20幅用于训练,其余20幅用于测试。每个原始图像的大小为565×584像素[44]。为了开发一个方形数据集,图像被裁剪成只包含第9列到第574列的数据,这使得每个图像都有565×565像素。在这个实现中,我们考虑了从驱动器数据集中的20个映像中随机选择的190000个图像块,其中171000个图像块用于培训,剩下的19000个图像块用于验证。对于图7所示的所有三个数据集,每个图像块的大小都是48×48。第二个数据集STARE包含20个彩色图像,每个图像的大小为700×605像素[45,46]。由于样本数量较少,因此经常使用两种方法对该数据集进行训练和测试。首先,训练有时是从所有20幅图像中随机选取样本[53]。
图7.左侧的示例图像块和相应的显示在右侧的图像块输出。
图8.使用R2U-Net的驱动器数据集的实验输出:第一行显示输入图像的灰度级,第二行显示基本真实,第三行显示实验输出。
另一种方法是“leave-one-out”方法,即对每个图像进行测试,并对剩余的19个样本进行训练[47]。因此,训练和测试之间没有重叠。在这个实现中,我们对STARE数据集使用了“leave-one-out”方法。CHASH_DB1数据集包含28个彩色视网膜图像,每个图像的大小为999×960像素[48]。该数据集中的图像采集自14名学龄儿童的左右眼。数据集被分成两组,其中样本是随机选择的。20个样本集用于培训,其余8个样本用于测试。
由于输入数据的维数大于整个驱动器数据集,我们考虑了STARE和CHASE_DB1的20个图像中总共250000个补丁。在这种情况下,225000个补丁用于培训,其余25000个补丁用于验证。由于二进制FOV(如图6第二行所示)不适用于STARE和CHASE_DB1数据集,因此我们使用类似于[47]中描述的技术来生成FOV掩码。基于补丁的方法的一个优点是,补丁允许网络访问像素的局部信息,这对整体预测有影响。此外,它确保输入数据的类是平衡的。在整个图像上随机地对输入面片进行采样,整个图像还包括视场的外部区域。
2) 皮肤癌分割
此数据集取自2017年举行的Kaggle皮肤病变分割竞赛[49]。该数据集共包含2000个样本。它包括1250个训练样本、150个验证样本和600个测试样本。每个样本的原始大小为700×900,在本次实现中将其重新缩放为256×256。训练样本包括原始图像,以及对应的包含癌症或非癌症病变的目标二值图像。对于目标病灶外的像素,目标像素用255或0的值表示。
3) 肺分割
2017年在Kaggle Data Science Bowl举办的肺结节分析(LUNA)竞赛旨在通过2D和3D CT图像发现肺部病变。所提供的数据集由534个二维样本组成,这些样本具有用于肺分割的相应标签图像[50]。在这项研究中,70%的图像用于训练,剩下的30%用于测试。原始图像的大小是512×512,但是在这个实现中,我们将图像大小调整到256×256像素。
B、 定量分析方法
为了定量分析实验结果,考虑了几个性能指标,包括准确度(AC)、敏感性(SE)、特异性(SP)、F1分数、Dice系数(DC)和Jaccard相似性(JS)。为此,我们还使用变量真阳性(TP)、真阴性(TN)、假阳性(FP)和假阴性(FN)。整体精度用式(4)计算,灵敏度用式(5)计算。
此外,使用以下等式计算特异性。

根据[51],DC用公式(7)表示。这里GT表示真实数值,SR表示分割结果:

JS用式(8)表示,如[52]所示。

然而,曲线下面积(AUC)和接收器工作特性(ROC)曲线是医学图像分割任务的常用评价指标。在这个实验中,我们利用这两种分析方法来评估所提出的方法的性能,同时考虑到现有的最新技术。

图9.RU-Net,R2U-Net模型,ResU-Net和U-Net的训练精度。
C、 结果
1)利用DRIVE数据集对视网膜血管进行分割。
用所提出的R2U网络模型实现的精确分割结果如图8所示,图9和10显示使用驱动器数据集时的培训和验证精度。这些图表明,与U-Net和ResU-Net相比,提出的R2U-Net和RU-Net模型在训练和验证阶段都提供了更好的性能。
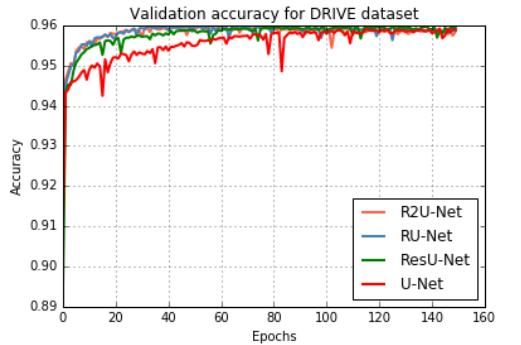
图10.根据ResU-Net和UNet验证所提出模型的准确性。
2) STARE数据集上的视网膜血管分割
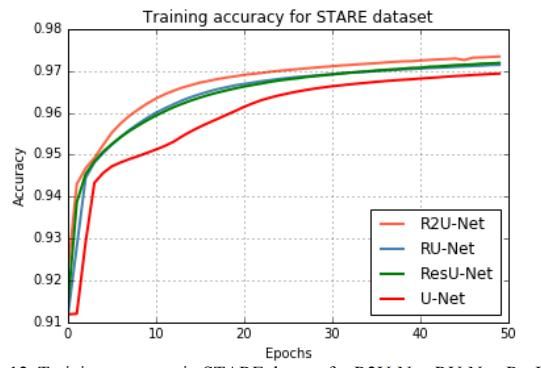
使用STARE数据集时R2U网络的实验输出如图11所示。STARE数据集的训练和验证精度如图所示。分别是12和13。
在R2U的训练中,它的表现比所有其他型号都要好。此外,图13中的验证精度表明,与等效的UNet和ResU-Net模型相比,RU-Net和R2U-Net模型提供了更好的验证精度。因此,性能证明了所提出的分割任务方法的有效性。

图11.使用R2U-Net的STARE数据集的实验输出:第一行显示标准化后的输入图像,第二行显示基本真实,第三行显示实验输出。
图12.R2U-Net、RU-Net、ResU-Net和U-Net在STARE数据集中的训练精度
图13.在STARE数据集中,R2U-Net、RU-Net、ResUNet和U-Net的验证精度。
3) CHASE_DB1
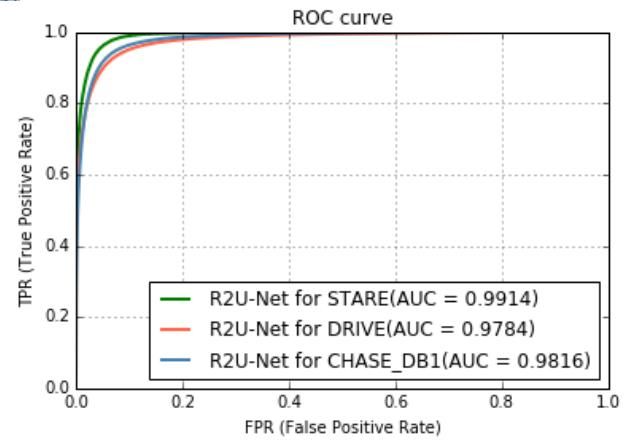
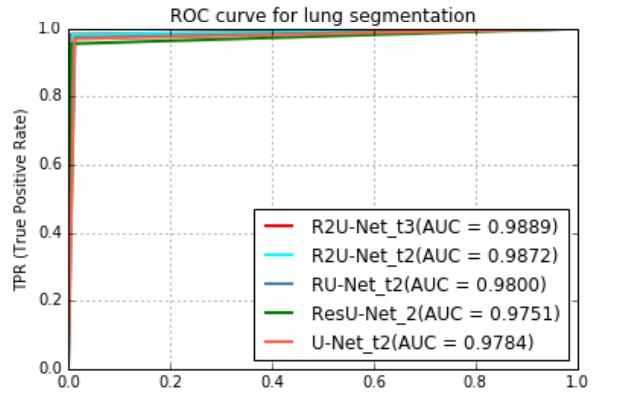
对于定性分析,R2U-Net的示例输出如图14所示。对于定量分析,结果如表1所示。从表中可以得出结论,在所有情况下,所提出的RU-Net和R2U-Net模型在AUC和准确性方面都表现出更好的性能。图15显示了三个视网膜血管分割数据集上R2U网络模型的最高AUC的ROC。
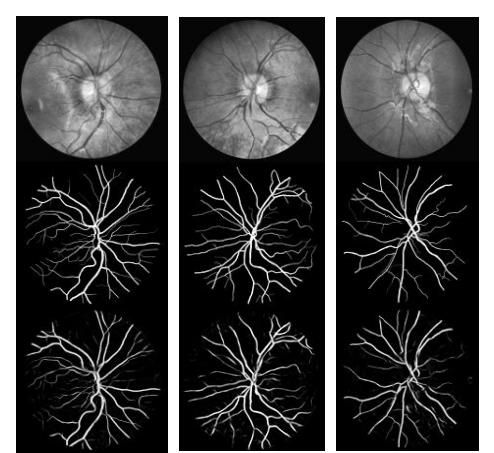
图14.CHASE_DB1数据集的定性分析。利用R2U-Net对8个测试样本进行分割输出。第一行显示输入图像,第二行显示基本真实数据,第三行显示使用R2U-Net的分割输出。
图15.用于视网膜血管分割的AUC是R2U-Net实现的最佳性能。
表1.视网膜血管分割方法的实验结果,以及与其他传统基于深度学习的方法的比较
在这个实现中,这个数据集是用均值减法进行预处理,并根据标准差进行归一化。我们使用ADAM优化技术,学习率为2×10-4,二进制交叉熵损失。此外,我们还计算了训练和验证阶段的均方误差。在这种情况下,10%的样本在培训期间用于验证,batch大小为32和150个epochs。
比较了R2U-Net和RU-Net模型与ResU-Net和U-Net模型在基于图像的端到端分割中的训练精度。结果如图16所示。验证精度如图17所示。在这两种情况下,与等效的U-Net和ResU-Net模型相比,本文提出的模型表现出更好的性能。这清楚地证明了所提出的模型在端到端图像分割任务中的鲁棒性。
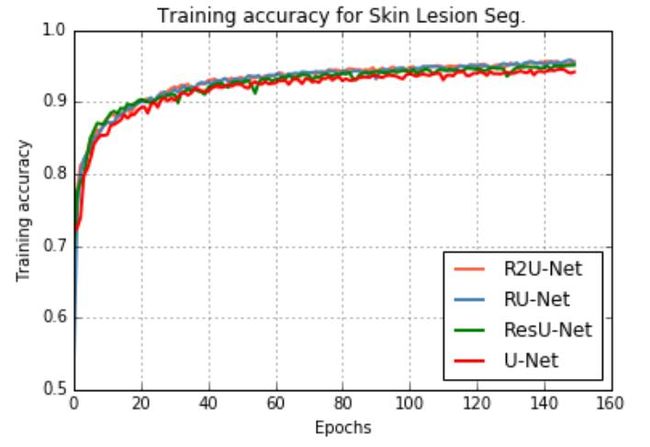
图16.皮损分割的训练精度。
图17.验证皮肤病变分割的准确性。
将本实验的定量结果与现有方法进行比较,如表2所示。来自测试阶段的一些示例输出如图18所示。第一列显示输入图像,第二列显示实际情况,第三列显示网络输出,第四列显示阈值为0.5的后处理后的最终输出。图18显示了精确的分割结果。
表2.所提出的皮肤癌病灶分割方法的实验结果及与其他现有方法的比较。JACCARD相似性评分(JSC)。
图18.这一结果表明,对于t=3的皮肤癌分割任务,R2-UNet的定性评估。第一列为输入样本,第二列为基本真实值,第三列显示网络输出,第四列显示0.5阈值后的最终结果。
5) 肺分割
肺分割是分析肺部相关疾病的重要手段,可应用于肺癌的分割和肺模式的分类,以识别其他问题。本实验采用ADAM优化器,学习速率为2×10-4。我们使用了二进制交叉熵损失,并在训练和验证过程中计算了均方误差。在本案例中,使用了16个batches的150个epoches样本进行验证。表三显示了本文所提出的模型相对于等效的U-Net和ResU-Net模型的表现如何。实验结果表明,在网络参数数目相同的情况下,该模型的性能优于U-Net和ResU-Net模型。
表3.RU-NET和R2U-NET肺部分割模型的实验结果以及与RESU-NET和U-NET模型的比较。
图19.当t=2和t=3时,ROC-AUC用于皮肤分割的四种模型。
此外,许多模型在分割任务期间**很难正确定义类边界**[64]。然而,如果我们观察图20所示的实验输出,第三列中的输出在边界上显示了不同的命中图,可以用来定义肺部区域的边界,而基本真实情况往往具有平滑的边界。另外,如果我们在第二行观察所提出方法的输入、基本真实和输出,则可以观察到,所提出方法的输出显示出更好的分割效果,并且具有适当的轮廓。图21示出了具有auc的ROC。最高的AUC通过当t=3时的额R2U网络方法实现。
图20.肺分割数据集R2U-NET性能的定性评估:第一列输入图像,第二列基本真实,第三列输出。
图21.ROC曲线在t=2和t=3的四种肺分割模型中的应用。
D、 评估
大多数情况下,对网络进行以下架构的不同分割任务评估:1→64→128→256→512→256→128→64→1,需要4.2百万的网络参数;1→64→128→256→512→256→256→128→64→64,分别需要8.5百万的网络参数。但是,我们还使用以下结构对U-Net、ResU-Net、RU-Net和R2U-Net模型进行了实验:1→16→32→64→128→64→32→16→1。在本例中,我们使用了t=3的时间步长,它指的是一个前向卷积层,然后是三个后续的递归卷积层。将该网络应用于皮肤和肺部病变的分割。虽然相对于递归卷积层中的时间步长,网络参数的数量增加了一点点,但是在表II和III的最后几行中可以清楚地看到性能的进一步提高,我们评估了基于视网膜血管分割的斑块模型和基于端到端图像的皮肤和肺部病变分割方法。
在这两种情况下,所提出的模型在所有三个数据集的AUC和准确性方面都优于现有的最新方法,包括ResU-Net和U-Net。表IV显示了针对不同时间步长具有不同网络参数数量的网络结构。在测试阶段,STARE、CHASE_DB和DRIVE数据集的处理时间分别为每个样本6.42、8.66和2.84秒。另外,皮肤癌和肺癌的分割分别需要0.22秒和1.145秒。
表4.网络参数的结构和数量。
E. 时间开销
在视网膜图像血管分割、皮肤癌分割和肺部分割中每个样本测试的时间开销分别为:
表5.测试阶段的时间开销
V. 结论与展望
在这篇文章中,我们提出了一个使用递归卷积神经网络和递归残差卷积神经网络的U-Net结构的扩展。提出的模型分别称为“RU-Net”和“R2U-Net”。这些模型在医学影像学领域有三种不同的应用,包括视网膜血管分割、皮肤癌病灶分割和肺部分割。实验结果表明,与现有的包括U-Net和残差U-Net(或ResU-Net)模型在内的方法相比,所提出的RU-Net和R2U-Net模型在相同网络参数的情况下具有更好的分割性能。此外,实验结果表明,这些模型不仅能保证在训练期间和测试阶段都有更好的表现。在未来,我们将探索相同的架构,从编码单元到解码单元,采用一种新颖的特征融合策略。
参考文献
[1] Krizhevsky, Alex, Ilya Sutskever, and Geoffrey E. Hinton. "ImageNet classification with deep convolutional neural networks." Advances in neural information processing systems. 2012.
[2] Long, J., Shelhamer, E., & Darrell, T. (2015). Fully convolutional networks for semantic segmentation. In Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition (pp. 3431- 3440).
[3] Wang, Naiyan, et al. “Transferring rich feature hierarchies for robust visual tracking.” arXiv preprint arXiv: 1501.04587 (2015).
[4] Mao, Junhua, et al. “Deep captioning with multimodal recurrent neural networks (m-rnn).” arXiv preprint arXiv: 1412.6632 (2014)
[5] Simonyan, Karen, and Andrew Zisserman. “Very deep convolutional networks for large-scale image recognition.” arXiv preprint arXiv: 1409.1556 (2014).
[6] Szegedy, Christian, et al. “Going deeper with convolutions.” Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. 2015.
[7] He, Kaiming, et al. “Deep residual learning for image recognition.” Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. 2016.
[8] Huang, Gao, et al. “Densely connected convolutional networks.” arXiv preprint arXiv:1608.06993 (2016).
[9] Sabour, Sara, Nicholas Frosst, and Geoffrey E. Hinton. “Dynamic routing between capsules.” Advances in Neural Information Processing Systems. 2017.
[10] Badrinarayanan, Vijay, Alex Kendall, and Roberto Cipolla. “Segnet: A deep convolutional encoder-decoder architecture for image segmentation.” arXiv preprint arXiv:1511.00561(2015).
[11] Ciresan, Dan, et al. “Deep neural networks segment neuronal membranes in electron microscopy images.” Advances in neural information processing systems. 2012.
[12] Ronneberger, Olaf, Philipp Fischer, and Thomas Brox. “U-net: Convolutional networks for biomedical image segmentation.” International Conference on Medical image computing and computer-assisted intervention. Springer, Cham, 2015.
[13] Çiçek, Özgün, et al. “3D U-Net: learning dense volumetric segmentation from sparse annotation.” International Conference on Medical Image Computing and Computer-Assisted Intervention. Springer International Publishing, 2016.
[14] Milletari, Fausto, Nassir Navab, and Seyed-Ahmad Ahmadi. “V-net: Fully convolutional neural networks for volumetric medical image segmentation.” 3D Vision (3DV), 2016 Fourth International Conference on. IEEE, 2016.
[15] Yang, Dong, et al. “Automated anatomical landmark detection ondistal femur surface using convolutional neural network.” Biomedical Imaging (ISBI), 2015 IEEE 12th International Symposium on. IEEE, 2015.
[16] Cai, Yunliang, et al. “Multi-modal vertebrae recognition using transformed deep convolution network.” Computerized Medical Imaging and Graphics 51 (2016): 11-19.
[17] Ramesh, N., J-H. Yoo, and I. K. Sethi. “Thresholding based on histogram approximation.” IEE Proceedings-Vision, Image and Signal Processing 142.5 (1995): 271-279.
[18] Sharma, Neeraj, and Amit Kumar Ray. “Computer aided segmentation of medical images based on hybridized approach of edge and region based techniques.” Proceedings of International Conference on Mathematical Biology’, Mathematical Biology Recent Trends by Anamaya Publishers. 2006.
[19] Boykov, Yuri Y., and M-P. Jolly. “Interactive graph cuts for optimal boundary & region segmentation of objects in ND images.” Computer Vision, 2001. ICCV 2001. Proceedings. Eighth IEEE International Conference on. Vol. 1. IEEE, 2001.
[20] Litjens, Geert, et al. “A survey on deep learning in medical image analysis.” arXiv preprint arXiv:1702.05747 (2017).
[21] Greenspan, Hayit, Bram van Ginneken, and Ronald M. Summers. “Guest editorial deep learning in medical imaging: Overview and future promise of an exciting new technique.” IEEE Transactions on Medical Imaging 35.5 (2016): 1153-1159.
[22] Havaei, Mohammad, et al. “Brain tumor segmentation with deep neural networks.” Medical image analysis 35 (2017): 18-31. [23] G. Brostow, J. Fauqueur, and R. Cipolla, “Semantic object classes in video: A high-definition ground truth database,” PRL, vol. 30(2), pp. 88– 97, 2009.
[23] S. Song, S. P.
[24] Lichtenberg, and J. Xiao, “Sun rgb-d: A rgb-d scene understanding benchmark suite,” in Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, pp. 567–576, 2015.
[25] Kistler, Michael, et al. “The virtual skeleton database: an open access repository for biomedical research and collaboration.” Journal of medical Internet research 15.11 (2013).
[26] He, Kaiming, et al. “Identity mappings in deep residual networks.” European Conference on Computer Vision. Springer International Publishing, 2016.
[27] S. Zheng, S. Jayasumana, B. Romera-Paredes, V. Vineet, Z. Su, D. Du, C. Huang, and P. H. Torr, “Conditional random fields as recurrent neural networks,” in Proceedings of the IEEE International Conference on Computer Vision, pp. 1529–1537, 2015.
[28] L.-C. Chen, G. Papandreou, I. Kokkinos, K. Murphy, and A. L. Yuille, “Semantic image segmentation with deep convolutional nets and fully connected CRFs,” in ICLR, 2015.
[29] Kendall, Alex, Vijay Badrinarayanan, and Roberto Cipolla. “Bayesian segnet: Model uncertainty in deep convolutional encoder-decoder architectures for scene understanding.” arXiv preprint arXiv:1511.02680 (2015).
[30] Zhang, Zhengxin, Qingjie Liu, and Yunhong Wang. “Road Extraction by Deep Residual U-Net.” arXiv preprint arXiv:1711.10684 (2017).
[31] Li, Ruirui, et al. “DeepUNet: A Deep Fully Convolutional Network for Pixel-level Sea-Land Segmentation.” arXiv preprint arXiv:1709.00201 (2017).
[32] Kayalibay, Baris, Grady Jensen, and Patrick van der Smagt. “CNN-based Segmentation of Medical Imaging Data.” arXiv preprint arXiv:1701.03056 (2017).
[33] Drozdzal, Michal, et al. “The importance of skip connections in biomedical image segmentation.” International Workshop on LargeScale Annotation of Biomedical Data and Expert Label Synthesis. Springer International Publishing, 2016.
[34] Chen, Hao, et al. “Dcan: Deep contour-aware networks for accurate gland segmentation.” Proceedings of the IEEE conference on Computer Vision and Pattern Recognition. 2016
[35] McKinley, Richard, et al. “Nabla-net: A Deep Dag-Like Convolutional Architecture for Biomedical Image Segmentation.” International Workshop on Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Springer, Cham, 2016.
[36] Q. Dou, L. Yu, H. Chen, Y. Jin, X. Yang, J. Qin, and P.-A. Heng, “3D deeply supervised network for automated segmentation of volumetric medical images,” Medical Image Analysis, vol. 41, pp. 40–54, 2017.
[37] Li, Wenqi, et al. “On the Compactness, Efficiency, and Representation of 3D Convolutional Networks: Brain Parcellation as a Pretext Task.” International Conference on Information Processing in Medical Imaging. Springer, Cham, 2017.
[38] Kamnitsas, Konstantinos, et al. “Efficient multi-scale 3D CNN with fully connected CRF for accurate brain lesion segmentation.” Medical image analysis 36 (2017): 61-78.
[39] Roth, Holger R., et al. “Deeporgan: Multi-level deep convolutional networks for automated pancreas segmentation.” International Conference on Medical Image Computing and Computer-Assisted Intervention. Springer, Cham, 2015.
[40] Chen, Hao, et al. “Voxresnet: Deep voxelwise residual networks for volumetric brain segmentation.” arXiv preprint arXiv:1608.05895 (2016).
[41] Liang, Ming, and Xiaolin Hu. “Recurrent convolutional neural network for object recognition.” Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. 2015.
[42] Alom, Md Zahangir, et al. “Inception Recurrent Convolutional Neural Network for Object Recognition.” arXiv preprint arXiv:1704.07709 (2017).
[43] Alom, Md Zahangir, et al. “Improved Inception-Residual Convolutional Neural Network for Object Recognition.” arXiv preprint arXiv:1712.09888 (2017).
[44] Staal, Joes, et al. “Ridge-based vessel segmentation in color images of the retina.” IEEE transactions on medical imaging23.4 (2004): 501-509.
[45] Hoover, A. D., Valentina Kouznetsova, and Michael Goldbaum. “Locating blood vessels in retinal images by piecewise threshold probing of a matched filter response.” IEEE Transactions on Medical imaging 19.3 (2000): 203-210.
[46] Zhao, Yitian, et al. “Automated vessel segmentation using infinite perimeter active contour model with hybrid region information with application to retinal images.” IEEE transactions on medical imaging 34.9 (2015): 1797-1807.
[47] Soares, João VB, et al. “Retinal vessel segmentation using the 2-D Gabor wavelet and supervised classification.” IEEE Transactions on medical Imaging 25.9 (2006): 1214-1222.
[48] Fraz, Muhammad Moazam, et al. “Blood vessel segmentation methodologies in retinal images–a survey.” Computer methods and programs in biomedicine 108.1 (2012): 407-433.
[49] https://challenge2017.isic-archive.com
[50] https://www.kaggle.com/kmader/finding-lungs-in-ct-data/data.
[51] Dice, Lee R. “Measures of the amount of ecologic association between species.” Ecology 26.3 (1945): 297-302.
[52] Jaccard, Paul. “The distribution of the flora in the alpine zone.” New phytologist 11.2 (1912): 37-50.
[53] Cheng, Erkang, et al. “Discriminative vessel segmentation in retinal images by fusing context-aware hybrid features.” Machine vision and applications 25.7 (2014): 1779-1792.
[54] Azzopardi, George, et al. “Trainable COSFIRE filters for vessel delineation with application to retinal images.” Medical image analysis 19.1 (2015): 46-57.
[55] Roychowdhury, Sohini, Dara D. Koozekanani, and Keshab K. Parhi. Blood vessel segmentation of fundus images by major vessel extraction and subimage classification." IEEE journal of biomedical and health informatics 19.3 (2015): 1118-1128.
[56] Liskowski, Paweł, and Krzysztof Krawiec. “Segmenting Retinal Blood Vessels With Deep Neural Networks.” IEEE transactions on medical imaging 35.11 (2016): 2369-2380.
[57] Li, Qiaoliang, et al. “A cross-modality learning approach for vessel segmentation in retinal images.” IEEE transactions on medical imaging 35.1 (2016): 109-118.
[58] Marín, Diego, et al. “A new supervised method for blood vessel segmentation in retinal images by using gray- level and moment invariants-based features.” IEEE Transactions on medical imaging 30.1 (2011): 146-158.
[59] Fraz, Muhammad Moazam, et al. “An ensemble classification-based approach applied to retinal blood vessel segmentation.” IEEE Transactions on Biomedical Engineering 59.9 (2012): 2538-2548.
[60] Fraz, Muhammad Moazam, et al. “Delineation of blood vessels in pediatric retinal images using decision trees-based ensemble classification.” International journal of computer assisted radiology and surgery 9.5 (2014): 795-811.
[61] Burdick, Jack, et al. “Rethinking Skin Lesion Segmentation in a Convolutional Classifier.” Journal of digital imaging (2017): 1-6.
[62] Codella, Noel CF, et al. “Skin lesion analysis toward melanoma detection: A challenge at the 2017 international symposium on biomedical imaging (isbi), hosted by the international skin imaging collaboration (isic).” arXiv preprint arXiv:1710.05006 (2017).
[63] Li, Yuexiang, and Linlin Shen. “Skin Lesion Analysis Towards Melanoma Detection Using Deep Learning Network.” arXiv preprint arXiv:1703.00577 (2017).
[64] Hsu, Roy Chaoming, et al. “Contour extraction in medical images using initial boundary pixel selection and segmental contour following.” Multidimensional Systems and Signal Processing 23.4 (2012): 469-498.
[65] Alom, Md Zahangir, et al. “The History Began from AlexNet: A Comprehensive Survey on Deep Learning Approaches.” arXiv preprint arXiv:1803.01164 (2018).