数据挖掘--关联规则实验
实验一 有趣的频繁项集
案例简介:
有时我们并不想寻找所有频繁项集 , 而只对包含某个特定元素项 的项集感兴趣。我们会寻找毒蘑菇中的一些公共特征, 利用这些特征 就能避免吃到那些有毒的蘑菇。UCI 的机器学习数据集合中有一个关于肋形蘑菇的 23 种特征的数据集 , 每一个特征都包含个标称数据值。我们必须将这些标称值转化为一个集合。幸运的是, 已经有人已经做 好了这种转换。Roberto Bayardo 对 UCI 蘑菇数据集进行了解析 , 将每 个蘑菇样本转换成一个特征集合(mushroom.dat) 。其中 , 枚举了每个特征的所有可能值, 如果某个样本包含特征 , 那么该特征对应的整数值被 包含数据集中。数据集的前几行如下所示:
1. 数据准备
读取数据集“ mushroom.dat ”文件,并只选择 有毒蘑菇的数据 。 注意删除存在缺失值的行数据
2. 数据建模
调用 aprior 或 FP 树算法,其中选择支持度阈值 0.4 。
3. 分析结果输出
只输出与毒蘑菇相关的属性值构成的频繁项集 ( 例如, [“2”,”59”])
4. Python代码实现
import pandas as pd
from mlxtend.preprocessing import TransactionEncoder
from mlxtend.frequent_patterns import apriori,association_rules
df = pd.read_csv("C:/Users/wcj770801/Desktop/Python/Mushroom.txt",header=None,sep =' ')
ind = (df.iloc[:,0]==2)
b = df.loc[ind]
b= b.iloc[:,:-1].values.tolist()
te_ary = te.fit(b).transform(b)
df = pd.DataFrame(te_ary, columns=te.columns_)
fre_item = apriori(df, min_support=0.6) 实验二 中医证型关联规则挖掘
案例简介:
恶性肿瘤俗称癌症,当前已成为危害我国居民生命健康的主要杀手。 应用中医药治疗恶性肿瘤已成为公认的综合治疗方法之一,且中医药 治疗乳腺癌有着广泛的适应证和独特的优势。从整体出发,调整机体 气血、阴阳、脏腑功能的平衡,根据不同的临床证候进行辨证论治。 确定“先证治”的方向: 即后续证侯尚未出现之前,需要截断恶化 病情的哪些后续证侯。发现中医症状间的关联关系和诸多症状间的规 律性,并且依据规则分析病因、预测病情发展以及为未来临床诊治供有鉴。这样,在治疗患者的过程中,医生可以有效地减少西医 治疗的毒副作用,为后续治疗打下基础并且还能够帮助乳腺癌患者 在手术后恢复体质,改善生存质量,有利于提高患者的生存机率。请
根据提供的数据实现以下目标。
1) 借助三阴乳腺癌患者的病理信息,挖掘患者的症状与中医证型之
间的关联关系。
2) 对截断治疗提供依据,挖掘潜性证素。
由于患者在围手术期、围化疗期、围放疗期和内分泌治疗期等各 个病程阶段,基本都会出现特定的临床症状,故而可以运用中医截断 疗法进行治疗,在辨病的基础上围绕各个病程的特殊证候先证而治。 截断扭转的主要观点是强调早期治疗,力图快速控制病情,截斯病情 的特殊证候先证而治. 截断扭转的主要观点是强调早期治疗,力图快速控制病情,截斯病情邪变深人,扭转阻止疾病恶化。
目前,患者的临床病理信息大部分都记录在纸张上,包含了患者的基 本信息、具体患病目前,患者的临床病理信息大部分都记录在纸张上, 包含了患者的基本信息、具体患病信息等,很少会将患者的患病信息存放于系统中,因此进行数据分析时会面临数据缺乏的情况。针对这种状况,采用问卷调查的方式收集数据; 运用数据挖掘技术对收集的 数据进况. 行数据探索与预处理,形成建模数据 : 采用关联规则算法, 挖掘各中医证素与乳腺癌 TNM 分期之间的关系,其中乳腺癌 TNM 分期是乳腺癌分期基本原则,I 期较轻, IV 期较严重。探索不同分期阶段的三阴乳腺癌患者的中医证素分布规律,以及采用截断病变发 展、先期干预的治疗思路,指导三阴乳腺癌的中医临床治疗。
1. 数据准备
本案例采用调查问卷的形式对数据进行搜集,数据获取的具体过程如
下。
1) 拟定调查问卷表并形成原始指标表。
2) 定义纳人标准与排除标准。
3) 将收集回来的问卷表整理成原始数据。
2. 数据预处理
本案例中数据预处理过程包括数据清洗、属性规约和数据变换。 数据来源于问卷调查,因此在数据预处理开始阶段,需要把纸质的问 卷整理成原始数据集。针对原始数据集,经过数据预处理,形成建模数据集。
(1)数据清洗
在收回的问卷中,存在无效的问卷,为了便于模型分析,需要对其进行处理。在经过问卷有效性条件筛选后,将有效问卷整理成原始数据,共 930 条记录。
(2)属性规约
本案例收集到的数据共有 73 个属性,为了更有效地对其进行挖掘,将其中冗余属性与挖掘任务不相关属性剔除。因此选取其中 6 种证型得分、TNM 分期的属性值构成数据集,见下表。
(3) 数据变换
本章数据变换主要采用属性构造和数据离散化两种方法对数据 进行处理。首先通过属性构造,获得证型系数,然后通过聚类算法对数据进行离散化处理,形成建模数据。
○
① 属性构造
为了更好地反映出中医证素分布的特征,采用证型系数代替具体
单证型的证素得分,证型相关系数计算公式为 : 证型系数 = 该证型得分 1 该证型总分。数据详见 ( cancer.xls )
②数据离散化
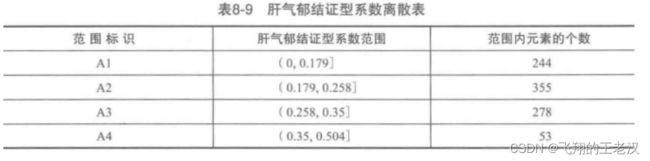
由于 Apriori 关联规则算法无法处理连续型数值变量,为了将原 始数据格式转换为适合建模的格式,需要对数据进行离散化。本章采用聚类算法对各个证型系数进行离散化处理,将每个属性聚成 4 类,其离散化后的数据格式见下表。
( 提示 : 利用 pandas 模块中的 cut 方法 ) 注意删除存在缺失值的行数据!!
3. 数据建模
本案例的目标是探索乳腺癌患者 TNM 分期与中医证型系数之间 的关系,因此采用关联规则算法,挖掘它们之间的关联关系。
(1) Apriori 算法主要用于寻找数据集中项之间的关联关系。它 揭示了数据项间的未知关系,基于样本的统计规律,进行关联规则挖掘。根据所挖掘的关联关系,可以从一个属性的信息来推断另一个属性的信息。当置信度达到某一阈值时,就可以认为规则成立。
最小支持度 6% 、最小置信度 75%
注意: 根据上述运行结果,我们得出了 5 个关联规则,如 ----H4 ,它的意思是 A3,F4=>H4 ,类似的, D---4---A2 的意思是 D2 , F3 , H4=>A2 。但是,并非所有关联规则都有意义的,我们 只在乎那些以 H 为规则 结果的规则 。
(2) 计算 有效蕴含表达式 的 不平衡比和 Kluc 度量。
4. 结果分析
1) A3 、 F4=>H4 支持度最大,达到 7.85% , 置信度最大,达到87.96%, 说明肝气郁结证型系数处于 (0.258 , 0.35] , 肝肾阴虚证型系数处于( 0.353 , 0.607] 范围内, TNM 分期诊断为 H4 期的可能性为 87.96% ,而这种情况发生的可能性为 7.85% 。
2) C3 、 F4=>H4 支持度 7.53% , 置信度 87.5% , 说明冲任失调证型系数处于(0.201,0.288] ,肝肾阴虚证型系数处于 ( 0.353 , 0.607] 范围内,TNM 分期诊断为 H4 期的可能性为 87.5% ,而这种情况发生的可能性为 7.53% 。
3) B2 、 F4=>H4 支持度 6.24% ,置信度 79.45% ,说明热毒蕴结证型系数处于(0.15,0.296] ,肝肾阴虚证型系数处于 ( 0.353 , 0.607] 范围内,TNM 分期诊断为 H4 期的可能性为 79. 45% ,而这种情况发生的可能性为 6.24% 。
综合以上分析, TNM 分期为 H4 期的三阴乳腺癌患者证型主要为肝肾阴虚证、热毒蕴结证、肝气郁结证和冲任失调,H4 期患者肝肾阴虚证和肝气郁结证的临床表现较为突出,其置信度最大达到
87.96% 。
4.Python代码实现
import pandas as pd
from mlxtend.preprocessing import TransactionEncoder
from mlxtend.frequent_patterns import apriori,association_rules
te = TransactionEncoder()
df = pd.read_excel("C:/Users/wcj770801/Desktop/Python/cancer.xls")
df["肝气郁结证型系数"] = pd.cut(df["肝气郁结证型系数"], [0,0.179,0.258,0.35,0.504], labels=["A1", "A2", "A3", "A4"],include_lowest=True)
df["热毒蕴结证型系数"] = pd.cut(df ["热毒蕴结证型系数"], [0,0.15,0.296,0.485,0.78], labels=["B1", "B2", "B3", "B4"],include_lowest=True)
df["冲任失调证型系数"] = pd.cut(df["冲任失调证型系数"], [0,0.201,0.288,0.415,0.61], labels=["C1", "C2", "C3", "C4"],include_lowest=True)
df["气血两虚证型系数"] = pd.cut( df["气血两虚证型系数"], [0,0.172,0.251,0.357,0.552], labels=["D1", "D2", "D3", "D4"],include_lowest=True)
df["脾胃虚弱证型系数"] = pd.cut( df["脾胃虚弱证型系数"], [0,0.154,0.256,0.375,0.526], labels=["E1", "E2", "E3", "E4"],include_lowest=True)
df["肝肾阴虚证型系数"] = pd.cut( df["肝肾阴虚证型系数"], [0,0.178,0.261,0.353,0.607], labels=["F1", "F2", "F3", "F4"],include_lowest=True)
b = df.values.tolist()
te_ary = te.fit(b).transform(b)
df = pd.DataFrame(te_ary, columns=te.columns_)
fre_itemset = apriori(df, min_support=0.06,use_colnames=True)
rules =association_rules(fre_itemset, metric='confidence', min_threshold=1, support_only=False)
consequents = rules['consequents'] #frozenset() 返回一个冻结的集合,冻结后集合不能再添加或删除任何元素。
idx = consequents.apply(lambda x: 'H4' in set(x))
aim_rules = rules[idx]
print(aim_rules)