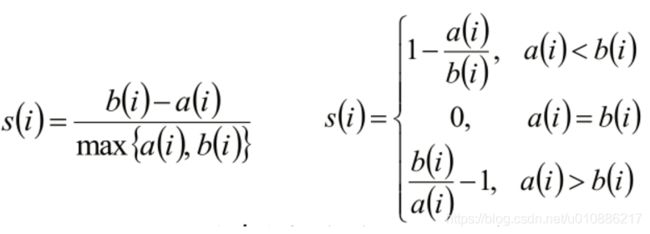机器学习(1):K-MEANS聚类算法
一、聚类简介
1.无监督问题:我们手里没有标签了
2.聚类:相似的东西分到一组
3.难点:如何评估,如何调参
二、基本概念:
1.K:要得到簇的个数,需要指定K
2.质心:均值,即向量各维取平均
3.距离的度量:常用欧几里得距离和余弦相似度(先标准化)
4.优化目标
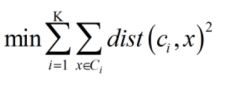
(1)Ci代表第i个簇的中心
(2)x是属于Ci的点
(3)dist:为distance距离,即欧几里得距离
(4)目标为:每一个点到该簇的距离之和,以及各个簇中心之和,为最小值!
5.注意:
(1)标准化
比如X轴0.01,0.02,0.03偏差比较小;Y轴,105,161偏差比较大。为了消除值本身大小的影响,所以需要将X/Y的范围都归类到[0,1]或者[-1,1]。
(2)一般用欧几里得距离
三、工作流程
1.初始化:假设K=3,即三个初始化点,C1,C2,C3。初始化三个点。

3.更新质心C1,C2,C3:依据2中的分类,求取每个分类的质心,作为新的C1',C2',C3。
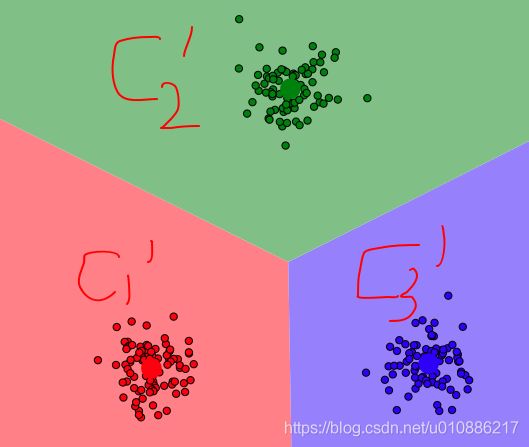
4.循环2到3
5.结束:当C1,C2,C3和C1',C2',C3'偏差满足要求停止。即,满足优化目标为止!
四、优势和劣势
1.优势
简单,快速,适合常规数据集
2.劣势
(1)K值难确定
(2)复杂度与样本呈线性关系
(3)很难发现任意形状的簇
五、聚类可视化
可视化网址过程【老外的网址,不错!】
(1)k-means
https://www.naftaliharris.com/blog/visualizing-k-means-clustering/
(2)dbscan
https://www.naftaliharris.com/blog/visualizing-dbscan-clustering/
六、实例
针对数据data.txt
name calories sodium alcohol cost
0 Budweiser 144 15 4.7 0.43
1 Schlitz 151 19 4.9 0.43
2 Lowenbrau 157 15 0.9 0.48
3 Kronenbourg 170 7 5.2 0.73
4 Heineken 152 11 5.0 0.77
5 Old_Milwaukee 145 23 4.6 0.28
6 Augsberger 175 24 5.5 0.401.python代码实现
1.获取数据
# beer dataset
import pandas as pd
beer = pd.read_csv('data.txt', sep=' ')
beer
2.抽取数据X
X = beer[["calories","sodium","alcohol","cost"]]
print(X)
3.K-means clustering创建实例
from sklearn.cluster import KMeans
km = KMeans(n_clusters=3).fit(X)
km2 = KMeans(n_clusters=2).fit(X)
4.结果:获取分类结果
km.labels_
5.加载到原始数据
beer['cluster'] = km.labels_
beer['cluster2'] = km2.labels_
beer.sort_values('cluster')2.绘图验证
(1)求每一类中心点:聚合函数groupby
1.获取中心值
from pandas.tools.plotting import scatter_matrix
%matplotlib inline
cluster_centers = km.cluster_centers_
cluster_centers_2 = km2.cluster_centers_
2.groupby:求取三个类簇的聚类中心点值
(1)
beer.groupby("cluster").mean()
(2)
beer.groupby("cluster2").mean()
结果:
calories sodium alcohol cost cluster
cluster2
0 91.833333 10.166667 3.583333 0.433333 1.333333
1 150.000000 17.000000 4.521429 0.520714 0.000000
3.重新排序
centers = beer.groupby("cluster").mean().reset_index() #按照对应分组类别
结果:
centers为
cluster calories sodium alcohol cost cluster cluster2
0 0 70.00 10.5 2.600000 0.420000 1 1
1 1 150.00 17.0 4.521429 0.520714 0 0
2 2 102.75 10.0 4.075000 0.440000 1 2
(2)绘图验证
绘制两个特征的图以及中心点
%matplotlib inline
import matplotlib.pyplot as plt
plt.rcParams['font.size'] = 14
import numpy as np
colors = np.array(['red', 'green', 'blue', 'yellow'])
---------
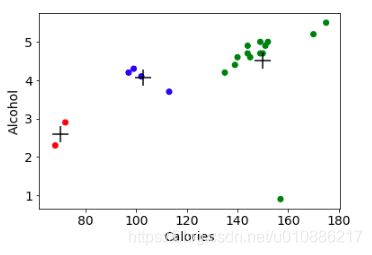
plt.scatter(beer["calories"], beer["alcohol"],c=colors[beer["cluster"]])
plt.scatter(centers.calories, centers.alcohol, linewidths=3, marker='+', s=300, c='black')
plt.xlabel("Calories")
plt.ylabel("Alcohol")
结果:
(3)两两特征分析
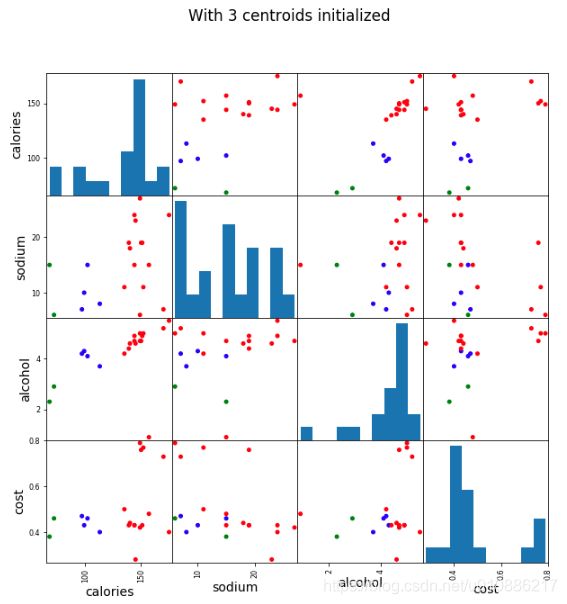
(a)三个簇
scatter_matrix(beer[["calories","sodium","alcohol","cost"]],s=100, alpha=1, c=colors[beer["cluster"]], figsize=(10,10))
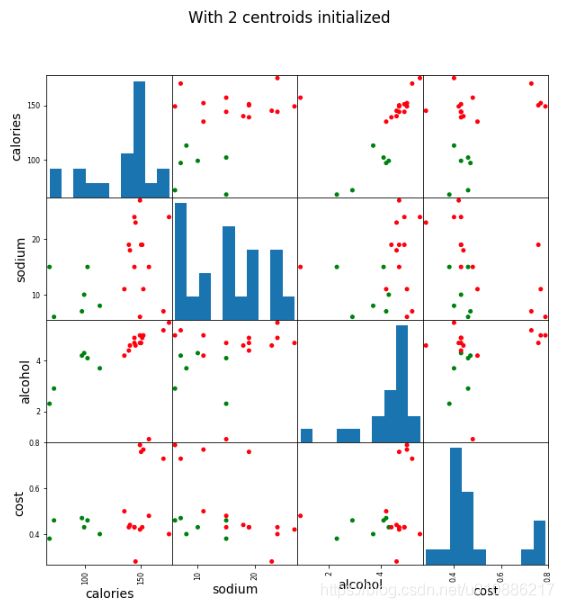
plt.suptitle("With 3 centroids initialized")(2)两个簇
scatter_matrix(beer[["calories","sodium","alcohol","cost"]],s=100, alpha=1, c=colors[beer["cluster2"]], figsize=(10,10))
plt.suptitle("With 2 centroids initialized")结果:
七、归一化改进Scaled data
1.归一化
四个特征值基本都在[-2,2]之间,消除差异
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
X_scaled2.KMeans拟合
km = KMeans(n_clusters=3).fit(X_scaled)3.结果添加到原始数据上
beer["scaled_cluster"] = km.labels_
beer.sort_values("scaled_cluster")4.求取每个簇的中心点
beer.groupby("scaled_cluster").mean()5.绘制两两特征的关系
pd.scatter_matrix(X, c=colors[beer.scaled_cluster], alpha=1, figsize=(10,10), s=100)6.优劣势
(1)优势:使每个特征的权重相似,不至于忽略较小,但是比较重要的特征。
(2)劣势:可能有的特征比较大,而且非常重要。这样会抹杀掉该特征的重要性。
八、聚类评估:轮廓系数(Silhouette Coefficient )
1.公式
(1)单点的轮廓系数
(2)将所有点的轮廓系数求平均,就是该聚类结果总的轮廓系数
2.概念:
(1)ai:计算样本i到同簇其他样本的平均距离ai。ai 越小,说明样本i越应该被聚类到该簇。将ai 称为样本i的簇内不相似度。
(2)bi:计算样本i到其他某簇Cj 的所有样本的平均距离bij,称为样本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:bi =min{bi1, bi2, ..., bik}
(3)结果:轮廓系数si
si接近1,则说明样本i聚类合理
si接近-1,则说明样本i更应该分类到另外的簇
若si 近似为0,则说明样本i在两个簇的边界上。
(4)将所有点的轮廓系数求平均,就是该聚类结果总的轮廓系数!
3.代码实现
from sklearn import metrics
score = metrics.silhouette_score(X,beer.cluster) #归一化前的结果
score_scaled = metrics.silhouette_score(X,beer.scaled_cluster) #归一化后的结果
print(score_scaled, score)
结果:
0.673177504646 0.179780680894
解释:
归一化后反而变差,因为,可能会消除部分重要的特征的值。
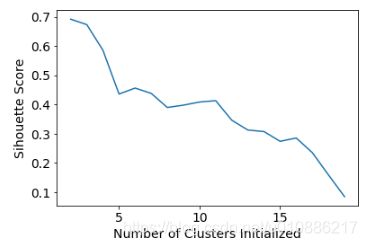
4.选择k值可以,循环看轮廓系数得分,选择轮廓系数偏高的值
scores = []
for k in range(2,20):
labels = KMeans(n_clusters=k).fit(X).labels_
score = metrics.silhouette_score(X, labels)
scores.append(score)
scores5.绘图
plt.plot(list(range(2,20)), scores)
plt.xlabel("Number of Clusters Initialized")
plt.ylabel("Sihouette Score")
结果:
[0.6917656034079486,
0.6731775046455796,
0.5857040721127795,
0.4355716067265817,
0.4559182167013378,
0.43776116697963136,
0.38946337473126,
0.39746405172426014,
0.4081599013899603,
0.41282646329875183,
0.3459775237127248,
0.31221439248428434,
0.30707782144770296,
0.2736836031737978,
0.2849514001174898,
0.23498077333071996,
0.1588091017496281,
0.08423051380151177]
6.绘图
plt.plot(list(range(2,20)), scores)
plt.xlabel("Number of Clusters Initialized")
plt.ylabel("Sihouette Score")