sklearn学习谱聚类
原文链接
1. sklearn谱聚类概述
\qquad 在sklearn的类库中,sklearn.cluster.SpectralClustering实现了基于Ncut的谱聚类,没有实现基于RatioCut的切图聚类。同时,对于相似矩阵的建立,也只是实现了基于K邻近法和全连接法的方式,没有基于 ϵ \epsilon ϵ-邻近法的相似矩阵。最后一步的聚类方法则提供了两种,K-Means算法和 discretize算法。
\qquad 对于SpectralClustering的参数,我们主要需要调参的是相似矩阵建立相关的参数和聚类类别数目,它对聚类的结果有很大的影响。当然其他的一些参数也需要理解,在必要时需要修改默认参数。
2. SpectralClustering重要参数与调参注意事项
\qquad 下面我们就对SpectralClustering的重要参数做一个介绍,对于调参的注意事项会一起介绍。
\qquad 1)n_clusters:代表我们在对谱聚类切图时降维到的维数,同时也是最后一步聚类算法聚类到的维数。也就是说sklearn中的谱聚类对这两个参数统一到了一起。简化了调参的参数个数。虽然这个值是可选的,但是一般还是推荐调参选择最优参数。
\qquad 2) affinity: 也就是我们的相似矩阵的建立方式。可以选择的方式有三类,第一类是 ‘nearest_neighbors’即K邻近法。第二类是’precomputed’即自定义相似矩阵。选择自定义相似矩阵时,需要自己调用set_params来自己设置相似矩阵。第三类是全连接法,可以使用各种核函数来定义相似矩阵,还可以自定义核函数。最常用的是内置高斯核函数’rbf’。其他比较流行的核函数有‘linear’即线性核函数, ‘poly’即多项式核函数, ‘sigmoid’即sigmoid核函数。如果选择了这些核函数, 对应的核函数参数在后面有单独的参数需要调。自定义核函数我没有使用过,这里就不多讲了。affinity默认是高斯核’rbf’。一般来说,相似矩阵推荐使用默认的高斯核函数。
\qquad 3) 核函数参数gamma: 如果我们在affinity参数使用了多项式核函数 ‘poly’,高斯核函数‘rbf’, 或者’sigmoid’核函数,那么我们就需要对这个参数进行调参。
\qquad 多项式核函数中这个参数对应 K ( x , z ) = ( γ x ⋅ z + r ) d K(x,z) = (\gamma x·z + r)^d K(x,z)=(γx⋅z+r)d中的 γ \gamma γ,一般需要通过交叉验证选择一组合适的 γ , r , d \gamma,r,d γ,r,d
\qquad 高斯核函数中这个参数对应 K ( x , z ) = e x p ( γ ∣ ∣ x − z ∣ ∣ 2 ) K(x,z)=exp(\gamma||x-z||^2) K(x,z)=exp(γ∣∣x−z∣∣2)中的 γ \gamma γ,一般需要通过交叉验证选择合适的 γ \gamma γ
\qquad sigmoid核函数中这个参数对应 K ( x , z ) = t a n h ( γ x ⋅ z + r ) K(x,z) = tanh(\gamma x·z + r) K(x,z)=tanh(γx⋅z+r)中的 γ \gamma γ,一般需要通过交叉验证选择一组合适的 γ , r \gamma,r γ,r
γ \qquad\gamma γ默认值为1.0,如果我们affinity使用’nearest_neighbors’或者是’precomputed’,则这么参数无意义。
\qquad 4)核函数参数degree:如果我们在affinity参数使用了多项式核函数 ‘poly’,那么我们就需要对这个参数进行调参。这个参数对应 K ( x , z ) = ( γ x ⋅ z + r ) d K(x,z) = (\gamma x·z + r)^d K(x,z)=(γx⋅z+r)d中的d,默认是3。一般需要通过交叉验证选择一组合适的 γ , r , d \gamma,r,d γ,r,d
\qquad 5)核函数参数coef0: 如果我们在affinity参数使用了多项式核函数 ‘poly’,或者sigmoid核函数,那么我们就需要对这个参数进行调参。
\qquad 多项式核函数中这个参数对应 K ( x , z ) = ( γ x ⋅ z + r ) d K(x,z) = (\gamma x·z + r)^d K(x,z)=(γx⋅z+r)d中的 r r r。一般需要通过交叉验证选择一组合适的 γ , r , d \gamma,r,d γ,r,d
\qquad sigmoid核函数中这个参数对应 K ( x , z ) = t a n h ( γ x ⋅ z + r ) K(x,z) = tanh(\gamma x·z + r) K(x,z)=tanh(γx⋅z+r)中的 r r r,一般需要通过交叉验证选择一组合适的 γ , r \gamma,r γ,r
\qquad coef0默认为1.
\qquad 6)kernel_params:如果affinity参数使用了自定义的核函数,则需要通过这个参数传入核函数的参数。
\qquad 7 ) n_neighbors: 如果我们affinity参数指定为’nearest_neighbors’即K邻近法,则我们可以通过这个参数指定KNN算法的K的个数。默认是10.我们需要根据样本的分布对这个参数进行调参。如果我们affinity不使用’nearest_neighbors’,则无需理会这个参数。
\qquad 8)eigen_solver:在降维计算特征值特征向量的时候,使用的工具。有 None, ‘arpack’, ‘lobpcg’, 和‘amg’4种选择。如果我们的样本数不是特别大,无需理会这个参数,使用’'None暴力矩阵特征分解即可,如果样本量太大,则需要使用后面的一些矩阵工具来加速矩阵特征分解。它对算法的聚类效果无影响。
\qquad 9)eigen_tol:如果eigen_solver使用了arpack’,则需要通过eigen_tol指定矩阵分解停止条件。
\qquad 10)assign_labels:即最后的聚类方法的选择,有K-Means算法和 discretize算法两种算法可以选择。一般来说,默认的K-Means算法聚类效果更好。但是由于K-Means算法结果受初始值选择的影响,可能每次都不同,如果我们需要算法结果可以重现,则可以使用discretize。
\qquad 11)n_init:即使用K-Means时用不同的初始值组合跑K-Means聚类的次数,这个和K-Means类里面n_init的意义完全相同,默认是10,一般使用默认值就可以。如果你的n_clusters值较大,则可以适当增大这个值。
\qquad 从上面的介绍可以看出,需要调参的部分除了最后的类别数n_clusters,主要是相似矩阵affinity的选择,以及对应的相似矩阵参数。当我选定一个相似矩阵构建方法后,调参的过程就是对应的参数交叉选择的过程。对于K邻近法,需要对n_neighbors进行调参,对于全连接法里面最常用的高斯核函数rbf,则需要对gamma进行调参。
3.SpectralClustering实例
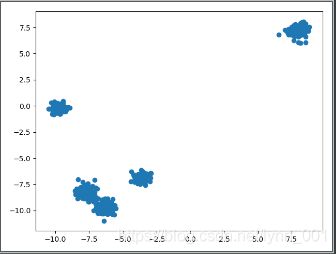
\qquad 首先我们生成500个个6维的数据集,分为5个簇。由于是6维,这里就不可视化了,代码如下:
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
X, y = make_blobs(n_samples=500, n_features=6, centers=5, cluster_std=[0.4, 0.3, 0.4, 0.3, 0.4], random_state=11)
plt.scatter(X[:, 0], X[:, 1], marker='o')
plt.show()
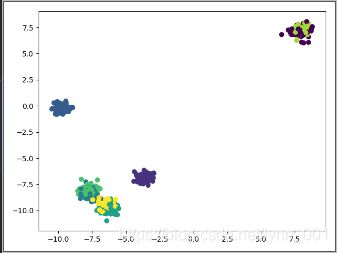
y_pred = SpectralClustering().fit_predict(X)
# Calinski-Harabasz Score 14907.099436228204
print("Calinski-Harabasz Score", metrics.calinski_harabaz_score(X, y_pred))
plt.scatter(X[:, 0], X[:, 1], c=y_pred)
plt.show()
\qquad 由于我们使用的是高斯核,那么我们一般需要对n_clusters和gamma进行调参。选择合适的参数值。代码如下:
for i, gamma in enumerate((0.01, 0.1, 1, 10)):
for j, k in enumerate((3, 4, 5, 6)):
y_pred = SpectralClustering(n_clusters=k, gamma=gamma).fit_predict(X)
print("Calinski-Harabasz Score with gamma=", gamma, "n_clusters=", k, "score:",
metrics.calinski_harabaz_score(X, y_pred))
"""
Calinski-Harabasz Score with gamma= 0.01 n_clusters= 3 score: 1979.7709609161868
Calinski-Harabasz Score with gamma= 0.01 n_clusters= 4 score: 3154.0184121901602
Calinski-Harabasz Score with gamma= 0.01 n_clusters= 5 score: 23410.63894999139
Calinski-Harabasz Score with gamma= 0.01 n_clusters= 6 score: 19303.734087657893
Calinski-Harabasz Score with gamma= 0.1 n_clusters= 3 score: 1979.7709609161868
Calinski-Harabasz Score with gamma= 0.1 n_clusters= 4 score: 3154.0184121901607
Calinski-Harabasz Score with gamma= 0.1 n_clusters= 5 score: 23410.638949991386
Calinski-Harabasz Score with gamma= 0.1 n_clusters= 6 score: 19427.96189435911
Calinski-Harabasz Score with gamma= 1 n_clusters= 3 score: 450.692778360567
Calinski-Harabasz Score with gamma= 1 n_clusters= 4 score: 120.1243266675767
Calinski-Harabasz Score with gamma= 1 n_clusters= 5 score: 23410.638949991386
Calinski-Harabasz Score with gamma= 1 n_clusters= 6 score: 633.021945343848
Calinski-Harabasz Score with gamma= 10 n_clusters= 3 score: 42.777268645847606
Calinski-Harabasz Score with gamma= 10 n_clusters= 4 score: 42.40099368087282
Calinski-Harabasz Score with gamma= 10 n_clusters= 5 score: 30.558274478353223
Calinski-Harabasz Score with gamma= 10 n_clusters= 6 score: 47.37991118563843
"""
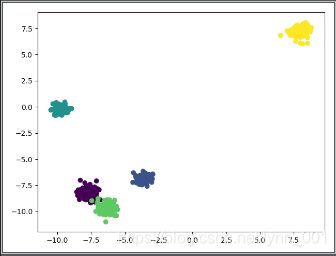
\qquad 可见最好的n_clusters是5,而最好的高斯核参数是1或者0.1,或者0.01。
\qquad 将n_clusters=5,gamma=0.1可视化
y_pred = SpectralClustering(n_clusters=5, gamma=0.1).fit_predict(X)
plt.scatter(X[:, 0], X[:, 1], c=y_pred)
plt.show()