系统聚类
系统聚类,即层次聚类法。先计算样本之间的距离,每次将距离最近的点合并到同一个类。然后,再计算类与类之间的距离,将距离最近的类合并为一个大类。最终经过不停的合并,直到合成了一个类。
正如样本之间的距离可以有不同的定义方法一样(欧氏距离、曼哈顿距离、马氏距离等),类与类之间的距离也有各种定义。例如可以定义类与类之间的距离为两类之间最近样本的距离,或者定义为两类之间最远样本的距离,也可以定义为两类重心之间的距离等等。类与类之间用不同的方法定义距离,就产生了不同的系统聚类方法。常用的系统聚类方法,即最短距离法、最长距离法、中间距离法、重心法、类平均法、可变类平均法、相似法、离差平方和法。系统聚类分析尽管方法很多,但归类的步骤基本上是一样的,所不同的仅是类与类之间的距离有不同的定义方法,从而得到不同的计算距离的公式。
原理详细讲解和网页(JS)计算实现,见银河统计系统聚类法 - 数据挖掘算法。
R和Python计算实现见下文。
目录概览
1)R语言实战
2)Python实战
3)R语言函数总结
4)Python函数总结
- Data - 鸢尾花iris数据集介绍
Iris也称鸢尾花卉数据集,是一类多重变量分析的数据集。通过花萼长度,花萼宽度,花瓣长度,花瓣宽度4个属性预测鸢尾花卉属于(Setosa,Versicolour,Virginica)三个种类中的哪一类。鸢尾花(iris)是数据挖掘常用到的一个数据集,包含150种鸢尾花的信息,每50种取自三个鸢尾花种之一(setosa,versicolour或virginica)。每个花的特征用下面的5种属性描述萼片长度(Sepal.Length)、萼片宽度(Sepal.Width)、花瓣长度(Petal.Length)、花瓣宽度(Petal.Width)、类(Species)。
| ID | Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species |
|---|---|---|---|---|---|
| 1 | 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 2 | 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 3 | 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 4 | 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 5 | 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| 6 | 5.4 | 3.9 | 1.7 | 0.4 | setosa |
| 7 | 4.6 | 3.4 | 1.4 | 0.3 | setosa |
| 8 | 5.0 | 3.4 | 1.5 | 0.2 | setosa |
| 9 | 4.4 | 2.9 | 1.4 | 0.2 | setosa |
| 10 | 4.9 | 3.1 | 1.5 | 0.1 | setosa |
| 11 | 5.4 | 3.7 | 1.5 | 0.2 | setosa |
| 12 | 4.8 | 3.4 | 1.6 | 0.2 | setosa |
| 13 | 4.8 | 3.0 | 1.4 | 0.1 | setosa |
| 14 | 4.3 | 3.0 | 1.1 | 0.1 | setosa |
| 15 | 5.8 | 4.0 | 1.2 | 0.2 | setosa |
| 16 | 5.7 | 4.4 | 1.5 | 0.4 | setosa |
| 17 | 5.4 | 3.9 | 1.3 | 0.4 | setosa |
| 18 | 5.1 | 3.5 | 1.4 | 0.3 | setosa |
| 19 | 5.7 | 3.8 | 1.7 | 0.3 | setosa |
| 20 | 5.1 | 3.8 | 1.5 | 0.3 | setosa |
| 21 | 5.4 | 3.4 | 1.7 | 0.2 | setosa |
| 22 | 5.1 | 3.7 | 1.5 | 0.4 | setosa |
| 23 | 4.6 | 3.6 | 1.0 | 0.2 | setosa |
| 24 | 5.1 | 3.3 | 1.7 | 0.5 | setosa |
| 25 | 4.8 | 3.4 | 1.9 | 0.2 | setosa |
| 26 | 5.0 | 3.0 | 1.6 | 0.2 | setosa |
| 27 | 5.0 | 3.4 | 1.6 | 0.4 | setosa |
| 28 | 5.2 | 3.5 | 1.5 | 0.2 | setosa |
| 29 | 5.2 | 3.4 | 1.4 | 0.2 | setosa |
| 30 | 4.7 | 3.2 | 1.6 | 0.2 | setosa |
| 31 | 4.8 | 3.1 | 1.6 | 0.2 | setosa |
| 32 | 5.4 | 3.4 | 1.5 | 0.4 | setosa |
| 33 | 5.2 | 4.1 | 1.5 | 0.1 | setosa |
| 34 | 5.5 | 4.2 | 1.4 | 0.2 | setosa |
| 35 | 4.9 | 3.1 | 1.5 | 0.2 | setosa |
| 36 | 5.0 | 3.2 | 1.2 | 0.2 | setosa |
| 37 | 5.5 | 3.5 | 1.3 | 0.2 | setosa |
| 38 | 4.9 | 3.6 | 1.4 | 0.1 | setosa |
| 39 | 4.4 | 3.0 | 1.3 | 0.2 | setosa |
| 40 | 5.1 | 3.4 | 1.5 | 0.2 | setosa |
| 41 | 5.0 | 3.5 | 1.3 | 0.3 | setosa |
| 42 | 4.5 | 2.3 | 1.3 | 0.3 | setosa |
| 43 | 4.4 | 3.2 | 1.3 | 0.2 | setosa |
| 44 | 5.0 | 3.5 | 1.6 | 0.6 | setosa |
| 45 | 5.1 | 3.8 | 1.9 | 0.4 | setosa |
| 46 | 4.8 | 3.0 | 1.4 | 0.3 | setosa |
| 47 | 5.1 | 3.8 | 1.6 | 0.2 | setosa |
| 48 | 4.6 | 3.2 | 1.4 | 0.2 | setosa |
| 49 | 5.3 | 3.7 | 1.5 | 0.2 | setosa |
| 50 | 5.0 | 3.3 | 1.4 | 0.2 | setosa |
| 51 | 7.0 | 3.2 | 4.7 | 1.4 | versicolor |
| 52 | 6.4 | 3.2 | 4.5 | 1.5 | versicolor |
| 53 | 6.9 | 3.1 | 4.9 | 1.5 | versicolor |
| 54 | 5.5 | 2.3 | 4.0 | 1.3 | versicolor |
| 55 | 6.5 | 2.8 | 4.6 | 1.5 | versicolor |
| 56 | 5.7 | 2.8 | 4.5 | 1.3 | versicolor |
| 57 | 6.3 | 3.3 | 4.7 | 1.6 | versicolor |
| 58 | 4.9 | 2.4 | 3.3 | 1.0 | versicolor |
| 59 | 6.6 | 2.9 | 4.6 | 1.3 | versicolor |
| 60 | 5.2 | 2.7 | 3.9 | 1.4 | versicolor |
| 61 | 5.0 | 2.0 | 3.5 | 1.0 | versicolor |
| 62 | 5.9 | 3.0 | 4.2 | 1.5 | versicolor |
| 63 | 6.0 | 2.2 | 4.0 | 1.0 | versicolor |
| 64 | 6.1 | 2.9 | 4.7 | 1.4 | versicolor |
| 65 | 5.6 | 2.9 | 3.6 | 1.3 | versicolor |
| 66 | 6.7 | 3.1 | 4.4 | 1.4 | versicolor |
| 67 | 5.6 | 3.0 | 4.5 | 1.5 | versicolor |
| 68 | 5.8 | 2.7 | 4.1 | 1.0 | versicolor |
| 69 | 6.2 | 2.2 | 4.5 | 1.5 | versicolor |
| 70 | 5.6 | 2.5 | 3.9 | 1.1 | versicolor |
| 71 | 5.9 | 3.2 | 4.8 | 1.8 | versicolor |
| 72 | 6.1 | 2.8 | 4.0 | 1.3 | versicolor |
| 73 | 6.3 | 2.5 | 4.9 | 1.5 | versicolor |
| 74 | 6.1 | 2.8 | 4.7 | 1.2 | versicolor |
| 75 | 6.4 | 2.9 | 4.3 | 1.3 | versicolor |
| 76 | 6.6 | 3.0 | 4.4 | 1.4 | versicolor |
| 77 | 6.8 | 2.8 | 4.8 | 1.4 | versicolor |
| 78 | 6.7 | 3.0 | 5.0 | 1.7 | versicolor |
| 79 | 6.0 | 2.9 | 4.5 | 1.5 | versicolor |
| 80 | 5.7 | 2.6 | 3.5 | 1.0 | versicolor |
| 81 | 5.5 | 2.4 | 3.8 | 1.1 | versicolor |
| 82 | 5.5 | 2.4 | 3.7 | 1.0 | versicolor |
| 83 | 5.8 | 2.7 | 3.9 | 1.2 | versicolor |
| 84 | 6.0 | 2.7 | 5.1 | 1.6 | versicolor |
| 85 | 5.4 | 3.0 | 4.5 | 1.5 | versicolor |
| 86 | 6.0 | 3.4 | 4.5 | 1.6 | versicolor |
| 87 | 6.7 | 3.1 | 4.7 | 1.5 | versicolor |
| 88 | 6.3 | 2.3 | 4.4 | 1.3 | versicolor |
| 89 | 5.6 | 3.0 | 4.1 | 1.3 | versicolor |
| 90 | 5.5 | 2.5 | 4.0 | 1.3 | versicolor |
| 91 | 5.5 | 2.6 | 4.4 | 1.2 | versicolor |
| 92 | 6.1 | 3.0 | 4.6 | 1.4 | versicolor |
| 93 | 5.8 | 2.6 | 4.0 | 1.2 | versicolor |
| 94 | 5.0 | 2.3 | 3.3 | 1.0 | versicolor |
| 95 | 5.6 | 2.7 | 4.2 | 1.3 | versicolor |
| 96 | 5.7 | 3.0 | 4.2 | 1.2 | versicolor |
| 97 | 5.7 | 2.9 | 4.2 | 1.3 | versicolor |
| 98 | 6.2 | 2.9 | 4.3 | 1.3 | versicolor |
| 99 | 5.1 | 2.5 | 3.0 | 1.1 | versicolor |
| 100 | 5.7 | 2.8 | 4.1 | 1.3 | versicolor |
| 101 | 6.3 | 3.3 | 6.0 | 2.5 | virginica |
| 102 | 5.8 | 2.7 | 5.1 | 1.9 | virginica |
| 103 | 7.1 | 3.0 | 5.9 | 2.1 | virginica |
| 104 | 6.3 | 2.9 | 5.6 | 1.8 | virginica |
| 105 | 6.5 | 3.0 | 5.8 | 2.2 | virginica |
| 106 | 7.6 | 3.0 | 6.6 | 2.1 | virginica |
| 107 | 4.9 | 2.5 | 4.5 | 1.7 | virginica |
| 108 | 7.3 | 2.9 | 6.3 | 1.8 | virginica |
| 109 | 6.7 | 2.5 | 5.8 | 1.8 | virginica |
| 110 | 7.2 | 3.6 | 6.1 | 2.5 | virginica |
| 111 | 6.5 | 3.2 | 5.1 | 2.0 | virginica |
| 112 | 6.4 | 2.7 | 5.3 | 1.9 | virginica |
| 113 | 6.8 | 3.0 | 5.5 | 2.1 | virginica |
| 114 | 5.7 | 2.5 | 5.0 | 2.0 | virginica |
| 115 | 5.8 | 2.8 | 5.1 | 2.4 | virginica |
| 116 | 6.4 | 3.2 | 5.3 | 2.3 | virginica |
| 117 | 6.5 | 3.0 | 5.5 | 1.8 | virginica |
| 118 | 7.7 | 3.8 | 6.7 | 2.2 | virginica |
| 119 | 7.7 | 2.6 | 6.9 | 2.3 | virginica |
| 120 | 6.0 | 2.2 | 5.0 | 1.5 | virginica |
| 121 | 6.9 | 3.2 | 5.7 | 2.3 | virginica |
| 122 | 5.6 | 2.8 | 4.9 | 2.0 | virginica |
| 123 | 7.7 | 2.8 | 6.7 | 2.0 | virginica |
| 124 | 6.3 | 2.7 | 4.9 | 1.8 | virginica |
| 125 | 6.7 | 3.3 | 5.7 | 2.1 | virginica |
| 126 | 7.2 | 3.2 | 6.0 | 1.8 | virginica |
| 127 | 6.2 | 2.8 | 4.8 | 1.8 | virginica |
| 128 | 6.1 | 3.0 | 4.9 | 1.8 | virginica |
| 129 | 6.4 | 2.8 | 5.6 | 2.1 | virginica |
| 130 | 7.2 | 3.0 | 5.8 | 1.6 | virginica |
| 131 | 7.4 | 2.8 | 6.1 | 1.9 | virginica |
| 132 | 7.9 | 3.8 | 6.4 | 2.0 | virginica |
| 133 | 6.4 | 2.8 | 5.6 | 2.2 | virginica |
| 134 | 6.3 | 2.8 | 5.1 | 1.5 | virginica |
| 135 | 6.1 | 2.6 | 5.6 | 1.4 | virginica |
| 136 | 7.7 | 3.0 | 6.1 | 2.3 | virginica |
| 137 | 6.3 | 3.4 | 5.6 | 2.4 | virginica |
| 138 | 6.4 | 3.1 | 5.5 | 1.8 | virginica |
| 139 | 6.0 | 3.0 | 4.8 | 1.8 | virginica |
| 140 | 6.9 | 3.1 | 5.4 | 2.1 | virginica |
| 141 | 6.7 | 3.1 | 5.6 | 2.4 | virginica |
| 142 | 6.9 | 3.1 | 5.1 | 2.3 | virginica |
| 143 | 5.8 | 2.7 | 5.1 | 1.9 | virginica |
| 144 | 6.8 | 3.2 | 5.9 | 2.3 | virginica |
| 145 | 6.7 | 3.3 | 5.7 | 2.5 | virginica |
| 146 | 6.7 | 3.0 | 5.2 | 2.3 | virginica |
| 147 | 6.3 | 2.5 | 5.0 | 1.9 | virginica |
| 148 | 6.5 | 3.0 | 5.2 | 2.0 | virginica |
| 149 | 6.2 | 3.4 | 5.4 | 2.3 | virginica |
| 150 | 5.9 | 3.0 | 5.1 | 1.8 | virginica |
1)R语言实战
1.1 R语言相关函数详解
系统聚类(Hierarchical Method)主要运用到了stats包(R语言内置包)中的hclust();cutree();rect.hclust()三个函数和绘图函数plot()。
- hclust(d, method="complete", members=NULL)
- cutree(tree, k=NULL, h=NULL)
- plot(hc.r, hang=-1, labels=NULL) 或者 plot(hc.r, hang=0.1, labels=F)
- rect.hclust(tree, k=NULL, which=NULL, x=NULL, h=NULL, border=2, cluster=NULL ) r语言中使用 hclust(d, method="complete", members=NULL) 来进行层次聚类,计算各种类与类之间的距离,其中d为距离矩阵,而method表示类的合并方法,有:
- single 最短距离法
- complete 最长距离法
- median 中间距离法
- mcquitty 相似法
- average 类平均法
- centroid 重心法
- ward.D2 离差平方和法r语言中使用 cutree(tree, k=NULL, h=NULL) 从hclust的结果对象中提取每个簇中的成员,其中 k 表示将簇中对象分成几类,h 表示削减树的高度值(通过高度值来确定分成几类)。
r语言中使用 plot(hc.r, hang=-1, labels=NULL) 对聚类结果进行绘图,其中 hang 等于数值,表示标签与末端树杈之间的距离,若是负数,则表示末端树杈长度是0,即标签对齐。而 labels 表示标签,默认是NULL,表示变量原有名称,labels=F:表示不显示标签。
r语言中使用 rect.hclust() 函数可以在plot()形成的系谱图中将指定类别中的样本分支用方框表示出来。其中 tree 表示一个hclust对象,k 指定要聚类的个数, border指定图中边界的颜色。
通过查看R帮助文档可以进一步理解函数各个参数的用法:
# ?hclust 1
hc <- hclust(dist(USArrests), "ave")
plot(hc)
plot(hc, hang = -1)
# ?hclust 2
hc <- hclust(dist(USArrests)^2, "cen")
memb <- cutree(hc, k = 10)
cent <- NULL
for(k in 1:10){
cent <- rbind(cent, colMeans(USArrests[memb == k, , drop = FALSE]))
}
hc1 <- hclust(dist(cent)^2, method = "cen", members=table(memb))
opar <- par(mfrow = c(1, 2))
plot(hc, labels = FALSE, hang = -1, main = "Original Tree")
plot(hc1, labels = FALSE, hang = -1, main = "Re-start from 10 clusters")
par(opar)
# ?cutree
hc <- hclust(dist(USArrests))
cutree(hc, k=1:5) # k = 1 is trivial[K = 1是微不足道]
cutree(hc, h=20)
# ?rect.hclust
hca <- hclust(dist(USArrests))
plot(hca)
rect.hclust(hca, k = 3, border = "red")
x <- rect.hclust(hca, h = 50, which = c(2,7), border = 3:4)
x注意:当用已知距离矩阵进行聚类时,即变量间的距离已经计算完,只是想用已知的距离矩阵进行聚类。这时,需将距离矩阵转成dist类型。然后再执行hclust()聚类和plot()画图。
# mydata作为距离矩阵,且为正方矩阵
mydata<-matrix(1:25,ncol=5);
class(mydata);
# 把mydata变成dist类型
mydist <- as.dist(mydata);
class(mydist);
[1] "dist"1.2 R语言对鸢尾花[iris]数据进行系统聚类分析
1.2.1) 系统聚类分析
步骤:
Step1: 复制上文中鸢尾花数据集到剪切板【通过剪切板为中介将数据转换为R环境中的变量】。
Step2: 运行以下程序进行层次(系统)聚类。
##################################
options(digits=4)
mydata <- read.table("clipboard",header=T)
class(mydata)
dim(mydata)
head(mydata)
# Data
iris_model_data <- mydata[,2:4]
# Step1:计算距离(样本和样本 或 变量与变量)
# 当用已知距离矩阵进行聚类时,即变量间的距离已经计算完,只是想用已知的距离矩阵进行聚类。
# 这时,需将距离矩阵转成dist类型。使用函数: as.dist()
d <- dist(iris_model_data, method="euclidean", diag = F)
# Step2:计算类与类之间的距离 - 注意:method方法可以选择
model <- hclust(d, method='ward.D2')
# Step3:根据hclust结果,选择聚类成几类。
groups <- cutree(model, k=3)
# Step4:绘制聚类结果
# plot(model,labels=F,hang=-1)
plot(model, labels=F, hang=-1, cex=0.6)
rect.hclust(model, k=3, border=2:4)
box()
# Step5:查看分组情况
table(groups,iris$Species)
# Step6:提取各个分类的详细结果
print_result <- function(model_data, groups, k) {
for(i in 1:k){
print(paste("hclust", i))
print(model_data[groups==i,])
}
}
print_result(iris_model_data, groups, 3)
##################################采用不同的类间距离进行聚类,相同样本间距离计算方式(euclidean),得到各种聚类结果,如下:
| model_name_c | model_name_e | category | result | setosa | versicolor | virginica |
|---|---|---|---|---|---|---|
| 最短距离法 | single | setosa | 1 | 50 | 0 | 0 |
| 最短距离法 | single | versicolor | 2 | 0 | 50 | 48 |
| 最短距离法 | single | virginica | 3 | 0 | 0 | 2 |
| 最长距离法 | complete | setosa | 1 | 50 | 0 | 0 |
| 最长距离法 | complete | versicolor | 2 | 0 | 21 | 49 |
| 最长距离法 | complete | virginica | 3 | 0 | 29 | 1 |
| 中间距离法 | median | setosa | 1 | 50 | 0 | 0 |
| 中间距离法 | median | versicolor | 2 | 0 | 38 | 49 |
| 中间距离法 | median | virginica | 3 | 0 | 12 | 1 |
| 相似法 | mcquitty | setosa | 1 | 50 | 0 | 0 |
| 相似法 | mcquitty | versicolor | 2 | 0 | 50 | 38 |
| 相似法 | mcquitty | virginica | 3 | 0 | 0 | 12 |
| 类平均法 | average | setosa | 1 | 50 | 0 | 0 |
| 类平均法 | average | versicolor | 2 | 0 | 50 | 38 |
| 类平均法 | average | virginica | 3 | 0 | 0 | 12 |
| 重心法 | centroid | setosa | 1 | 50 | 0 | 0 |
| 重心法 | centroid | versicolor | 2 | 0 | 46 | 50 |
| 重心法 | centroid | virginica | 3 | 0 | 4 | 0 |
| 离差平方和法 | ward.D2 | setosa | 1 | 50 | 0 | 0 |
| 离差平方和法 | ward.D2 | versicolor | 2 | 0 | 19 | 49 |
| 离差平方和法 | ward.D2 | virginica | 3 | 0 | 31 | 1 |
从上表各种聚类结果可知,采用相似法(mcquitty)和类平均法(average)进行系统聚类时,效果是做好的,这两种方法都正确的将鸢尾花的类别setosa和类别versicolor的样本聚到了一类,而将类别virginica的样本聚到了两类中。综合所有聚类方法结果可知,类别为setosa的鸢尾花,都成功聚到了一起,而类别为versicolor和virginica的鸢尾花聚类时混到了一起,说明类别setosa与其它两类差别很大,而类别versicolor与virginica存在一定的相似性,所以采用不用的聚类方法时,这两种类别都是混合存在。
通过对鸢尾花数据进一步分析可知,当保持类间聚类计算方法不变(如:single),而改变样本间距离计算方式,得到的聚类结果基本一致。
1.2.2) 通过对“距离矩阵”绘制热图
步骤:
Step1: 计算得到“距离矩阵” - d
Step2: 绘制热图--更多查看 ?heatmap.2
##################################
library(gplots)
heatmap.2(as.matrix(d), tracecol=NA)
# heatmap.2(as.matrix(d), labRow=F, labCol=F, tracecol=NA)
##################################图1.1:“距离矩阵”热图
1.2.3) 为了显示聚类的效果,通过对数据降维,使用ggplot2绘制聚类结果
Step1: 结合多维标度和聚类的结果,先将数据用MDS进行降维
Step2: 以不同的形状表示原来的分类,并用不同的颜色来表示聚类的结果
##################################
mds <- cmdscale(d,k=2,eig=T)
x <- mds$points[,1]
y <- mds$points[,2]
library(ggplot2)
ggplot(data.frame(x,y),aes(x,y)) + geom_point(size=3,alpha=0.8,aes(colour=factor(result),shape=iris$Species))
##################################图1.2:MDS降维结果图
1.2.4) 使用各种聚类方法进行聚类并绘图
##################################
options(digits=4)
mydata <- read.table("clipboard",header=T)
class(mydata)
dim(mydata)
head(mydata)
iris_model_data <- mydata[,2:4]
d <- dist(iris_model_data, method="manhattan", diag = TRUE, upper = FALSE)
opar <- par(mfrow = c(3,3))
method_data <- c('single', 'complete', 'median', 'mcquitty', 'average', 'centroid', 'ward.D2')
for(i in 1:length(method_data)){
model <- hclust(d, method=method_data[i])
plot(model, labels=F, hang=-1, cex=0.6)
rect.hclust(model, k=3, border=2:4)
box()
}
par(opar)
##################################图1.3:各种聚类方法聚类结果图
1.2.5) 各种谱系图画法
##################################
options(digits=4)
mydata <- read.table("clipboard",header=T)
class(mydata)
dim(mydata)
head(mydata)
iris_model_data <- mydata[,2:4]
d <- dist(iris_model_data, method="manhattan", diag = TRUE, upper = FALSE)
method_data <- c('single', 'complete', 'median', 'mcquitty', 'average', 'centroid', 'ward.D2')
for(i in 1:length(method_data)){
model <- hclust(d, method=method_data[i])
# 各种谱系图画法
# as.dendrogram()函数 ?as.dendrogram 查看帮助
dend1 <- as.dendrogram(model)
opar <- par(mfrow=c(2,2), mar=c(4,3,1,2))
plot(dend1,cex=0.6)
plot(dend1,nodePar=list(pch=c(1,NA), cex=0.8, lab.cex=0.8), type = "t", center=T)
plot(dend1,edgePar=list(col=1:2,lty=2:3),dLeaf=1, edge.root = T)
plot(dend1,nodePar=list(pch=2:1,cex=.4*2:1,col=2:3), horiz=T)
par(opar)
}
par(opar)
##################################图1.4:single方法进行聚类的各种谱系图画法示例
1.2.6) 美化聚类图形
##################################
options(digits=4)
mydata <- read.table("clipboard",header=T)
class(mydata)
dim(mydata)
head(mydata)
iris_model_data <- mydata[,2:4]
d <- dist(iris_model_data, method="manhattan", diag = TRUE, upper = FALSE)
hc <- hclust(d, method='single')
# 在此基础上,美化图形。
op = par(bg = "grey")
plot(hc, col="blue", col.main="black", col.lab="black", col.axis="black", lwd=1, lty=1, sub="", hang=-1, axes=FALSE)
# add axis
axis(side = 2, at = seq(0, 4, 1), col = "blue", labels = FALSE, lwd = 1)
# add text in margin
mtext(seq(0, 4, 1), side = 2, at = seq(0, 4, 1), line = 1, col = "blue", las = 2,outer = F)
par(op)
##################################图1.5:美化聚类图形
1.2.7) 分割系统树
##################################
options(digits=4)
mydata <- read.table("clipboard",header=T)
class(mydata)
dim(mydata)
head(mydata)
iris_model_data <- mydata[,2:4]
d <- dist(iris_model_data, method="manhattan", diag = TRUE, upper = FALSE)
hc <- hclust(d, method='single')
# 当然我们还可以使用系统树对象!
hcd <- as.dendrogram(hc)
# 绘制图形
op <- par(mfrow = c(3, 1))
plot(hcd)
plot(hcd, type = "triangle")
plot(hcd, type = "rectangle")
# 我们还可以分割系统树
# par(mfrow = c(1, 1))
op <- par(mfrow = c(3, 1))
# 一共分为2组
plot(cut(hcd, h=2)$upper, main="Upper tree of cut at h=2")
# 绘制第一、二组
plot(cut(hcd, h=2)$lower[[1]], main="First branch of lower tree with cut at h=2")
plot(cut(hcd, h=2)$lower[[2]], main="Second branch of lower tree with cut at h=2")
par(op)
# 分支合并
# 我们可不可以将2组里面的分支部分合并呢?
result<-cut(hcd, h=2)
# 将1和2组合并
result1<-merge(result$lower[[1]],result$lower[[2]])
plot(result1)
##################################图1.6:系统树图
图1.7:分割系统树图
1.2.8) 分组结果用颜色区分
##################################
options(digits=4)
mydata <- read.table("clipboard",header=T)
class(mydata)
dim(mydata)
head(mydata)
iris_model_data <- mydata[,2:4]
d <- dist(iris_model_data, method="manhattan", diag = TRUE, upper = FALSE)
hc <- hclust(d, method='single')
# 当然我们还可以使用系统树对象!
hcd <- as.dendrogram(hc)
# 我们还可以将分组结果用颜色区分出来
library(RColorBrewer)
labelColors <- brewer.pal(n=3, name="Set1")
# 聚类分组
clusMember <- cutree(hc, 3)
## 自定义函数
## toy example to set colored leaf labels :
local({
colLab <<- function(n) {
if(is.leaf(n)) {
a <- attributes(n)
i <<- i+1
attr(n, "nodePar") <-
c(a$nodePar, list(lab.col = mycols[i], lab.font = i%%3))
}
n
}
mycols <- grDevices::rainbow(attr(hcd,"members"))
i <- 0
})
clusDendro <- dendrapply(hcd, colLab)
plot(clusDendro, main="Cool Dendrogram", type="triangle", horiz=T)
plot(clusDendro, main="Cool Dendrogram", type="rectangle", horiz=T)
##################################图1.8:triangle系统树图
图1.9:rectangle系统树图
1.2.9) 绘制比较有趣的图形
##################################
options(digits=4)
mydata <- read.table("clipboard",header=T)
class(mydata)
dim(mydata)
head(mydata)
iris_model_data <- mydata[,2:4]
d <- dist(iris_model_data, method="manhattan", diag = TRUE, upper = FALSE)
hc <- hclust(d, method='single')
# 其中,还有一个比较好用的包ape,可以绘制比较有趣的图形。
library(ape)
op <- par(mfrow = c(3, 2))
# plot basic tree
plot(as.phylo(hc), cex = 0.9, label.offset = 1)
# cladogram
plot(as.phylo(hc), type = "cladogram", cex = 0.9, label.offset = 1)
# unrooted
plot(as.phylo(hc), type = "unrooted",no.margin = T,cex = 0.5)
# fan
plot(as.phylo(hc), type = "fan")
# radial
plot(as.phylo(hc), type = "radial")
# 我们还可以添加颜色。
plot(as.phylo(hc),type = "fan",tip.color = hsv(runif(15, 0.65, 0.95), 1, 1, 0.7), edge.color = hsv(runif(10, 0.65, 0.75), 1, 1, 0.7),
edge.width = runif(20, 0.5, 3), use.edge.length = TRUE,
col = "gray80",cex=0.5)
par(op)
mycol <- c("#556270", "#4ECDC4", "#1B676B", "#FF6B6B", "#C44D58")
clus5 <- cutree(hc, 5)
op <- par(bg = "#1B676B") # "#E8DDCB" 白色
plot(as.phylo(hc), type = "fan", tip.color = mycol[clus5], label.offset = 1, cex = 0.7)
par(op)
##################################图1.10:趣图
图1.11: 添加背景色图
1.2.10) 使用包sparcl绘制聚类结果
##################################
options(digits=4)
mydata <- read.table("clipboard",header=T)
class(mydata)
dim(mydata)
head(mydata)
iris_model_data <- mydata[,2:4]
d <- dist(iris_model_data, method="manhattan", diag = TRUE, upper = FALSE)
hc <- hclust(d, method='single')
# 包sparcl也是可以绘制聚类结果的
library(sparcl)
y <- cutree(hc, 3)
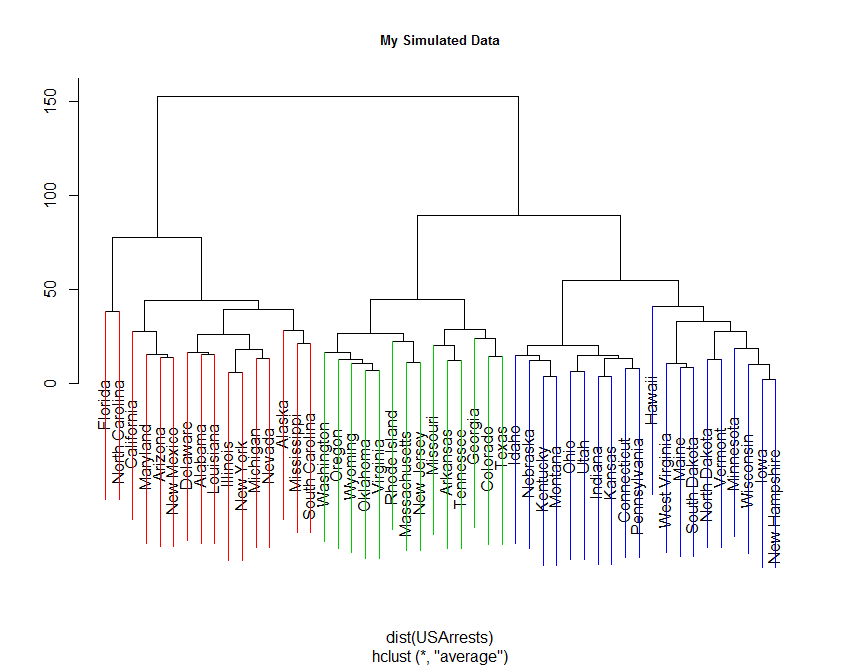
ColorDendrogram(hc, y = y, labels = names(y), main = "My Simulated Data", cex=0.8, branchlength = 100)
##################################图1.12: 包sparcl聚类结果图
1.2.11) 使用包ggdendro绘制聚类结果
##################################
options(digits=4)
mydata <- read.table("clipboard",header=T)
class(mydata)
dim(mydata)
head(mydata)
iris_model_data <- mydata[,2:4]
d <- dist(iris_model_data, method="manhattan", diag = TRUE, upper = FALSE)
hc <- hclust(d, method='single')
# 当然了,你还可以试试这个包ggdendro
library(ggdendro)
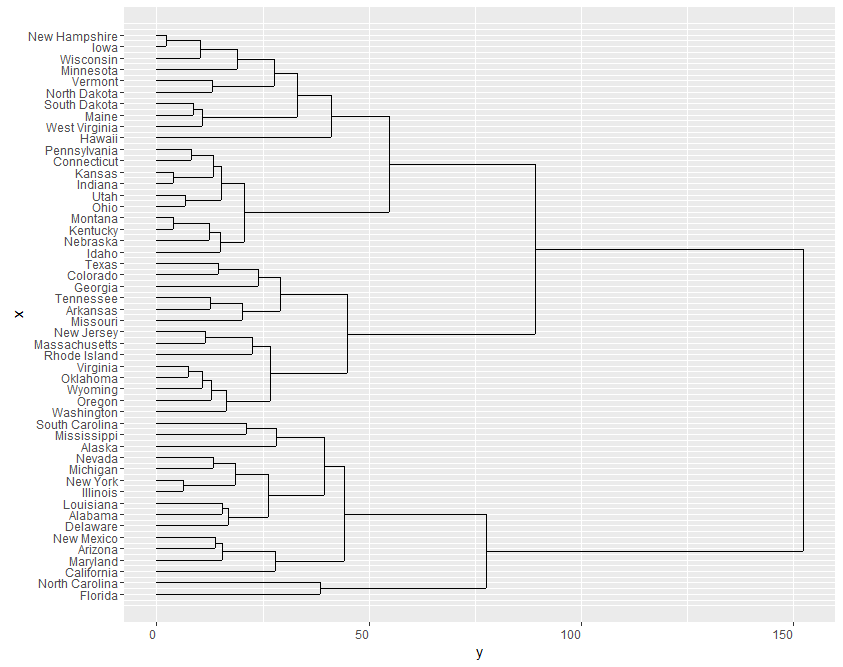
ggdendrogram(hc)
ggdendrogram(hc, rotate = TRUE,size = 1,theme_dendro = FALSE,color = "tomato")
ddata <- dendro_data(as.dendrogram(hc), type = "triangle")
##################################图1.13: 包ggdendro聚类结果图
2)Python实战
3)R语言函数总结
4)Python函数总结