生信_反相蛋白阵列(RPPA)_附实例
反相蛋白阵列(RPPA)
文章目录
- 反相蛋白阵列(RPPA)
-
- RPPA
- RPPA数据的形式
-
-
- level 1
- level 2
- level 3(较常用到)
- level 4
-
- 数据下载
- TCGA蛋白质数据可视化网站TCPA
- RPPA数据分析示例(以GBM癌型为例)
RPPA
- 基本原理:通过特异性抗体对凝胶电泳处理过的细胞或者生物组织样品进行着色。通过分析着色的位置和着色深度获得特定蛋白质在所分析的细胞或组织中表达情况的信息。
- 基因芯片的定量信息是表达量的高低,蛋白芯片的定量是丰度的高低
- 可以同时检测至少150种抗体(现在具体的数字可能更高),大于1000多个样本。
- 样本制备只需要大概40ul
RPPA数据的形式
level 1
- 最原始的数据,也是最少用到的数据
- 一般是图形数据(.tiff文件),数据是把着色的情况直接存成图片,可以经过一定的处理转化为矩阵数据
level 2
系列稀释的校准无的信号强度和相应的蛋白质浓度建立的标准曲线
level 3(较常用到)
每个样本,每个蛋白的标准化后的表达值矩阵。
level 4
同一个样本经过不同批次的检测,将不同批次按照取均值或者其他处理方法合并成的一个表达谱
数据下载
TCGA中提供了16种癌型,大约5000个样本的数据。
-
firebrowse网站下载
- firebrowse,外国网站,可能需要等比较久
2. 点击SELECT COHORT选择想要下载的癌型
3. 点击RPPA Analyses 选择下载RPPA数据
4. 选择右边条形图纵坐标的Rever Phase Protein Array,会弹出下载的链接
5. 这样子就能下载到表达谱和注释文件了。
TCGA蛋白质数据可视化网站TCPA
TCPA提供了对TCGA的蛋白数据进行可视化的服务
- 统一样本内不同蛋白之间的相关分析
- 不同肿瘤或者肿瘤亚型之间同一蛋白的差异分析
- 同一个肿瘤内,按照蛋白的表达中值将样本分为高表达组和低表达组,进行生存分析
- 都是比较简单的生信分析,有兴趣可以点击这里
- firebrowse,外国网站,可能需要等比较久
RPPA数据分析示例(以GBM癌型为例)
-
下载表达谱数据
-
用R包下载TCGA临床数据
-
调用相关R包
library(TCGAbiolinks) library(dplyr) library(DT) library(SummarizedExperiment) library(xlsx) -
下载临床信息代码
cancer_type <- "TCGA-GBM" ##癌型 query.clinical<-GDCquery(project=cancer_type,data.category="Clinical",file.type=".xml") result.clinical<-getResults(query.clinical) GDCdownload(query.clinical)##如果数据一直下载失败,可以尝试files.per.chunk=3,意思是同时只下载3个文件,默认是6个 ##根据自己的需要下载相应的数据 clinical_patient<-GDCprepare_clinic(query.clinical,clinical.info="patient")##病人信息 clinical_drug<-GDCprepare_clinic(query.clinical,clinical.info="drug")##药物信息 clinical_radiation<-GDCprepare_clinic(query.clinical,clinical.info="radiation")##化疗信息 clinical_follow_up<-GDCprepare_clinic(query.clinical,clinical.info="follow_up")##生存信息 clinical_stage_event<-GDCprepare_clinic(query.clinical,clinical.info="stage_event")##分期信息 clinical_new_tumor_event<-GDCprepare_clinic(query.clinical,clinical.info="new_tumor_event")##肿瘤复发信息 clinical_admin<-GDCprepare_clinic(query.clinical,clinical.info="admin")##管理人员信息 ##保存信息 save.image(file="clinical_xml.Rdata") ##设置输出的文件名字 OUT_excel_name=paste(cancer_type,"clinical.xlsx",sep="_") ##根据上面自己选择的信息写入相应的表格 library(xlsx) write.xlsx(clinical_patient,file = OUT_excel_name,sheetName="patient",append = T) write.xlsx(clinical_drug,file = OUT_excel_name,sheetName="drug",append = T) write.xlsx(clinical_radiation,file = OUT_excel_name,sheetName="radiation",append = T) write.xlsx(clinical_follow_up,file = OUT_excel_name,sheetName="follow_up",append = T) write.xlsx(clinical_stage_event,file = OUT_excel_name,sheetName="stage_event",append = T) write.xlsx(clinical_new_tumor_event,file = OUT_excel_name,sheetName="new_tumor_event",append = T) write.xlsx(clinical_admin,file = OUT_excel_name,sheetName="admin",append = T)
-
-
根据自己的需要,用下载的临床信息中寻找相应的信息(肿瘤分期、药物敏感、肿瘤大小等),制作标签文件label.txt。我的代码要求的label文件是有第一行最为表头,第一列是样本名,由于表达谱那边的样本名是以‘.’分隔,所以这里也全部替换成.。第二列是自己制作的标签,
-
打开GBM.protein_exp__mda_rppa_core__mdanderson_org__Level_3__protein_normalization__data.data.txt文件用excel打开,将第二行去掉
-
读入文件
library(stringr) options(stringsAsFactors = F) ## 去掉空行 Exp <- read.delim("E:/daiMa/R/RPPA/gdac.broadinstitute.org_GBM.Merge_protein_exp__mda_rppa_core__mdanderson_org__Level_3__protein_normalization__data.Level_3.2016012800.0.0/GBM.protein_exp__mda_rppa_core__mdanderson_org__Level_3__protein_normalization__data.data.txt", row.names=1) Exp <- na.omit(Exp) Exp <- as.matrix(Exp) label <- read.delim("E:/daiMa/R/RPPA/label.txt") GPL <- read.delim("E:/daiMa/R/RPPA/gdac.broadinstitute.org_GBM.RPPA_AnnotateWithGene.Level_3.2016012800.0.0/GBM.antibody_annotation.txt") -
处理表达谱的样本名
library(stringr)
## 取表达谱的样本名
sample <- colnames(Exp)
## 因为TCGA的样本只需要前十二个字符能匹配
for (i in 1:length(sample)){
sample[i] <- substring(sample[i],1,12)
}
-
根据label文件的标签将表达谱样本分类
## 对标签 loc <- match(sample,label[,1]) na_loc <- which(is.na(loc)) ##去掉对不上标签的 loc <- loc[-na_loc] Exp <- Exp[,-na_loc] Label <- label[loc,2] length(Label) dim(Exp) ## 将表达谱根据标签分成两类 Exp1 <- Exp[,which(Label==1)] Exp0 <- Exp[,which(Label==0)] -
使用t检验
##使用t检验 geneid <- row.names(Exp) t_result=matrix(,length(geneid),3) for (i in 1:nrow(Exp)) { Var.test_p<-var.test(Exp1[i,],Exp0[i,])$p.value if (Var.test_p>0.05) T_test=t.test(Exp1[i,],Exp0[i,],alternative="two.sided",paired=F,var.equal=T) else T_test=t.test(Exp1[i,],Exp0[i,],alternative="two.sided",paired=F,var.equal=F) t_result[i,1:3]=c(geneid[i],T_test$statistic,T_test$p.value) } colnames(t_result)=c("geneid","t_value","p_value") head(t_result) -
用找到的差异基因做KEGG富集
因为R包的KEGG需要的是基因的ENTREZID,所以我们还需要使用bitr函数转换一下
## 取得蛋白名 Gene <- t_result[t_result[,3]<0.05,] ## 将蛋白名转化为基因ID library(clusterProfiler) library(org.Hs.eg.db) ## 将蛋白名和基因symbol进行匹配 loc <- match(Gene[,1],GPL[,2]) Gene[,1] <- GPL[loc,1] ## 将SYMBOL转化为ENTREZID a <- bitr(Gene[,1],fromType = "SYMBOL",toType = "ENTREZID",OrgDb = "org.Hs.eg.db") loc <- match(Gene[,1],a$SYMBOL) na_loc <- which(is.na(loc)) Gene <- Gene[-na_loc,] loc <- loc[-na_loc] Gene[,1] <- a$ENTREZID geneid<- as.numeric(Gene[,1])接下来就是要进行通路富集了
## 通路富集 library(clusterProfiler) KEGGresult <- enrichKEGG(gene = geneid,organism = 'human',pvalueCutoff = 0.05,qvalueCutoff = 0.05) ## 画气泡图 dotplot(KEGGresult,font.size=8,showCategory=10) ## 最多显示10个通路 ## 在网页中显示出通路的信息 browseKEGG(KEGGresult,'hsa04115')-
这里enrichKEGG的参数organism非常的坑,官方每个物种都提供了两种输入形式,以人为例,有hsa或human两种,如果其中一种报错,那就换另外一种,如果两个都报错,可能是代理的问题(起码我把代理关掉报错就没了)
-
结果可以使用以下几个函数来看
KEGGresult@result$ID##富集通路的名称 KEGGresult@result$p.adjust## 矫正后的p值,上面enrichKEGG的p阈值看的就是这个,而不是p值 KEGGresult@result$qvalue## q值,一般默认使用的是“BH”方法 KEGGresult@result$Description## 富集通路的名称 -
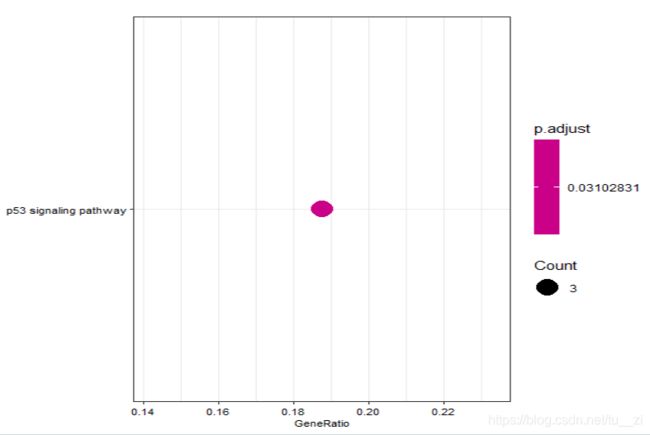
画出的气泡图:
可以看出这些差异基因只显著富集在一个通路上(当然我们也可以调低阈值来增加富集的通路),可以看到富集的通路是“p53 signaling pathway”
-
可以使用以下代码查看富集的通路的ID和通路名称
KEGGresult@result$ID KEGGresult@result$Descriptio -
在网页上查看富集到的通路的信息
## 在网页中显示出通路的信息 browseKEGG(KEGGresult,'hsa04115') ##参数分别是富集的结果和需要查询的通路ID号得到一个这样子的通路图。点击图上的通路名称或者基因名称会跳出相应的信息。p53 signaling pathway与细胞的生长和死亡有关,是一个已经很多研究证实的和癌症发生发展相关的通路。
-
这时候好奇一下我们富集在这个通路的基因究竟有哪些,可以通过以下代码实现
n <- 1## 想查第几个通路的基因 rich_geneID <- strsplit(KEGGresult@result$geneID[n],"/")[[1]] rich_geneID rich_gene <- bitr(rich_geneID,fromType = "ENTREZID",toType = "SYMBOL",OrgDb = org.Hs.eg.db)$SYMBOL rich_gene得到的结果是“BCL2”,“CDK1”,“CHEK1”
当然图中标红的基因就是差异基因中富集到该通路的基因,名称可能有些不同
我们可以点进去看看,可以看到
- bcl2是凋亡调节因子
- CHEK1 是检查点激酶1,通过查资料发现 ChEk1是启动DNA损伤检查点所必需的,并且最近已被证明在正常(未受干扰的)细胞周期中起作用,
- CDK1是细胞周期蛋白依赖性激酶1。
-
-
为了进一步分析,我们做蛋白互作网络图
-
将找到的差异蛋白输出成txt文件
##输出差异基因的蛋白名 Protein <- t_result[t_result[,3]<0.05,1] write.table(Protein,file = "Protein.txt",sep = "\t",quote = F,row.names = F)##quote参数为F的意思是输出的字符串不带“” -
获取蛋白互作网络
-
打开string官网
-
选择Multiple proteins
-
将刚才输出的文件打开,降低航的x去掉,然后复制到List of Names,按search
-
选择人类然后继续
-
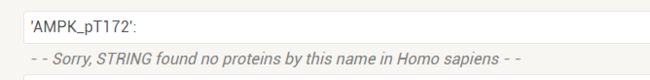
显示没有AMPK_pT172这个蛋白
点击继续
-
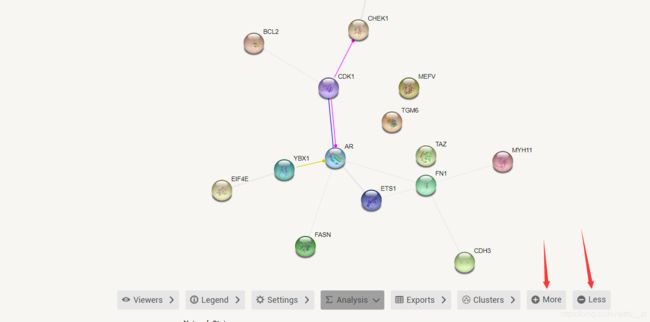
这时候就能看到蛋白互作网络了
按图中的more或者less可以减少基因数量或增加基因数量
-
点击exports,再点击下载TSV文件,得到一个tsv文件
-
打开cytoscape,直接将下载的文件拖入,并按确定,就会得到一个蛋白互作网络图,这要要比string那个网络图要美观一些,也可以自己设置许多样式
-
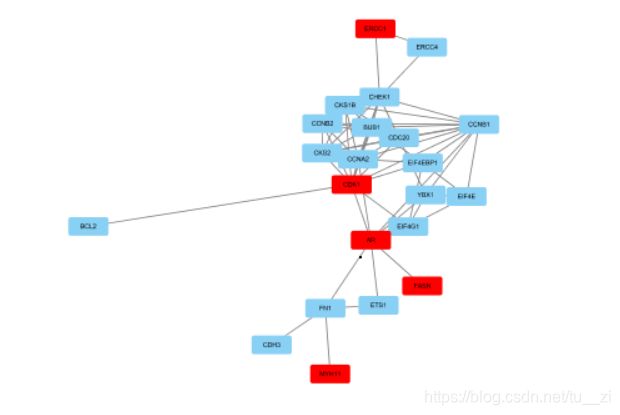
我们会希望在图中标识出我们找到的差异基因
如上图,首先我们选择菜单中的select-nodes-from Id List file,然后选择刚刚的使用R语言生成的Protein.txt,然后选择
然后依次选择图中的位置,就能选择要为目标基因设置的颜色了
-
通过MCODE插件寻找hub蛋白
这么多蛋白,其中哪些是发挥关键作用的蛋白呢?这时候我们就可以使用MCODE插件
-
安装MCODE插件
选择APPs-APP Manger…然后搜索MCODE安装即可
-
打开MCODE
Apps-MCODE
-
使用
- 可以使用找到的子网络在进行一通路富集,找到这个关键字网络主要富集在哪些功能通路,这里就不示范了。
-
-
-
这样子一份简单蛋白质数据的分析就大概这样子结束啦~
博主还只是一个小白,所以如果有错误还请大家帮忙指出来,谢谢大家!希望大家能一起学习。